在之前的一节当中,图型名称有些混乱,从这一节开始将做如下统一(不全面):
| 英文名称 | 中文名称 |
| bar | 条形图 |
| line | 线图 |
| area | 面积图 |
| pie | 饼图 |
| high-low | 高低图 |
| pareto | 帕累托图 |
| control | 控制图 |
| boxplot | 箱线图 |
| error bar | 误差条图 |
| scatter | 散点图 |
| P-P | P-P正态概率图 |
| Q-Q | Q-Q正态概率图 |
| sequence | 序列图 |
| ROC Curve | ROC分类效果曲线图 |
| Time Series | 时间序列图 |
好了,言归正传。那么什么又是点柱图(dot histogram)呢?之前我又称之为蜂群图(beeswarm)。还有称之为抖点图(jitter plots)。总之无论如何,在糗世界里我都称之为点柱图吧。
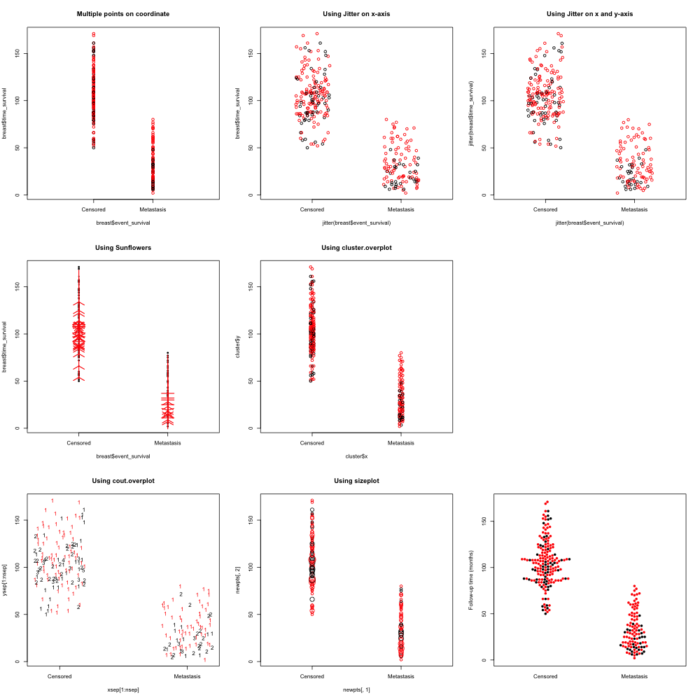
我们先看点柱图效果:
以下是代码
|
以下是解释
在很多情况下,我们画散点图的时候,有许多点拥有相同的横坐标,如果我们简单的使用plot(x,y)的方式,会显得这些点拥挤在一起,象图中左上角一样,非常的不舒服。我们需要把这些点分散开。
最基本的思路是,把横坐标抖散(jitter),使本来都拥有相同坐标的点的横坐标稍有不同。jitter是基类函数{base},无需调用任何包。
|
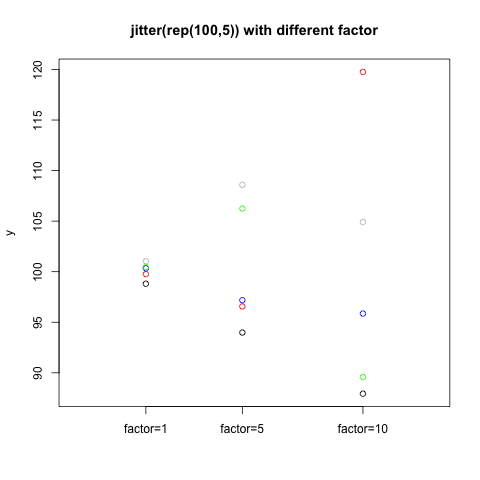
我们比较图中上边靠右的两个图,我们发现,如果只抖散x坐标的话,还是有些点会粘在一起,所以同时抖散y坐标会好一些。我们可以使用factor参数来控制jitter抖散的强度。
|
在graphics包中提供了一个sunflowerplot的函数。它的目的是用花瓣数目多少来显示落在同一坐标上的点的数目。但是从中左图看来,点多的时候效果并非总是那么好。
在plotrix包中提供了一些有意思的函数来解决点挤在一起的这个问题,它们分别是cluster.overplot, count.overplot, sizeplot。这三个函数的效果如图中及下靠左的两个。cluster.overplot的方法类似抖散,count overplot的方法是使用数字来显示落在同一坐标上的点的数目,sizeplot的方法是使用不同大小是点来显示落在同一坐标上的点的数目。从效果来看,点多的时候效果也并非理想。
而上一次提到过的蜂群图似乎是解决这一问题的最佳方案。
我们得出结论,在点数不同的情况下,使用plotrix包及sunflowerplot是不错的。但点数较多的情况下,还是使用jitter和beeswarm较为稳妥。
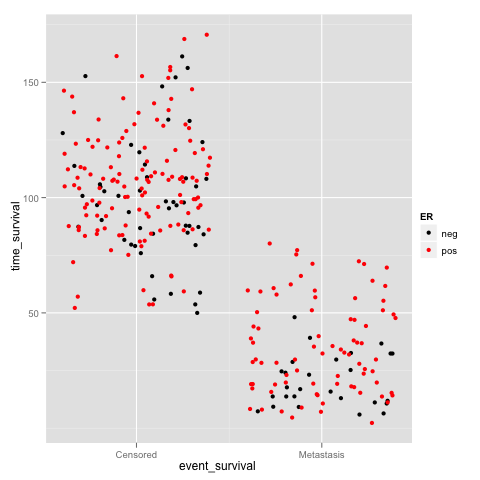
我们也可以使用ggplot2包中的geom来绘制点柱图
|
还有一种图,名称为Engelmann-Hecker-Plot, 由plotrix的ehplot来实现。
|