做完microRNA芯片测序之后预测microRNA的靶基因,筛选重要的靶基因进行验证是研究中的常见套路,做出结果后写到文章Result里大致如下。
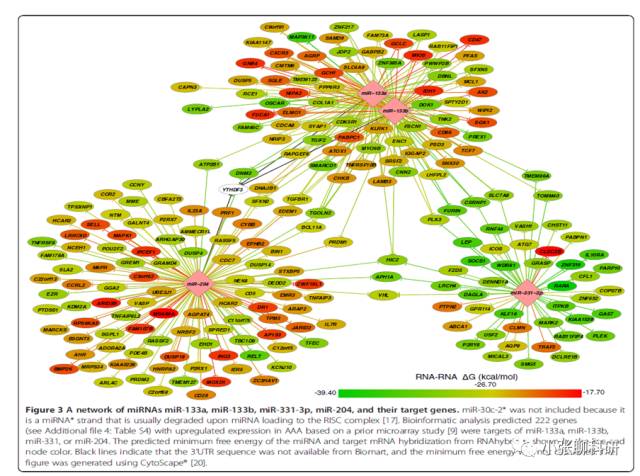
如果我是审稿人,看到这么干巴巴的一段话肯定心情烦躁。故而该文作者增加了Figure 3。这样的一张图放在2-3分的文章里是加分极多的。
这种简洁明了的microRNA-mRNA关系表达正是Cytoscape的强项。下面本宫就来画一个!
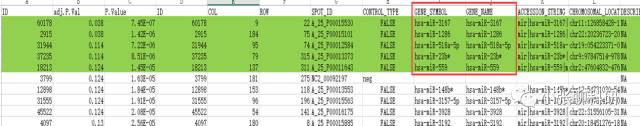
先在Pubmed上找了一个microRNA的数据包GSE72199[2],GEO2R筛选了差异表达的microRNA:
随便找了P值最小的5个,然后去TargetScan(http://www.targetscan.org/)上进行了靶基因预测。(如果确实需要进行靶基因预测并进行验证的话,还是需要多个工具一起使用取交集的,常用的有TargetScan、Mirtrail、Mirdb和MiRbase等)
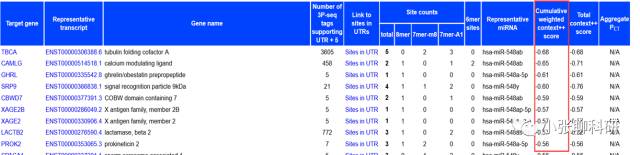
筛选Cumulative weighted context++ score≤-0.4的靶基因,做成如下的EXCEL表格:
再将EXCEL表格中的内容复制到TXT文本中,导入Cytoscape:
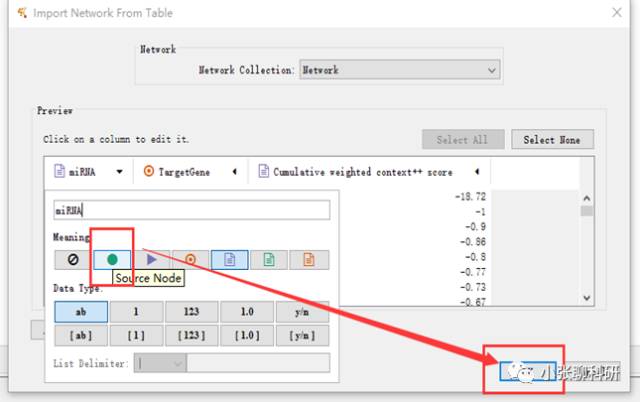
将第一列选择为Source Nodes,导入后得到下图:
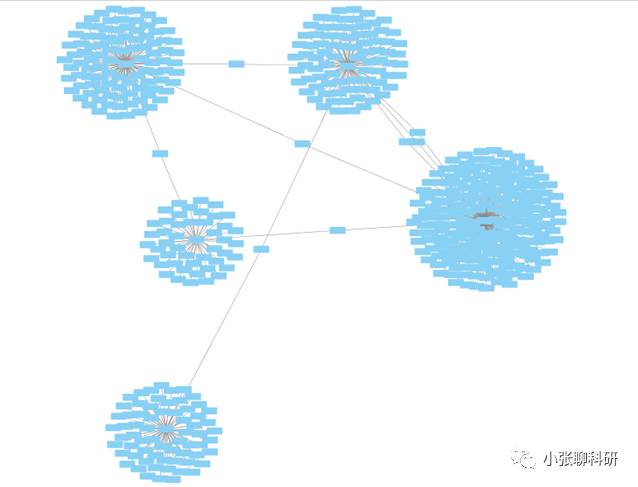
按住SHIFT和鼠标左键画框,把太远的Nodes拖得聚拢一些,不然太占地方了:
处理好了之后,我们需要对Nodes和Edges进行个性化的标注。Nodes的注释这里分为两类:1、mRNA还是microRNA;mRNA的颜色随Cumulative weighted context++渐变。Edges的注释为:作用关系随Cumulativeweighted context++ score渐变(可以是颜色也可以是线条粗细)。
首先导入Table文件,还是之前那个TXT文件:
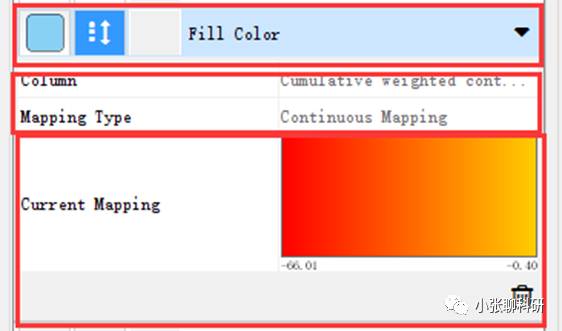
选择设置Nodes的颜色,Column选择Cumulative weighted context++ score,颜色选成渐变,然后编辑色阶
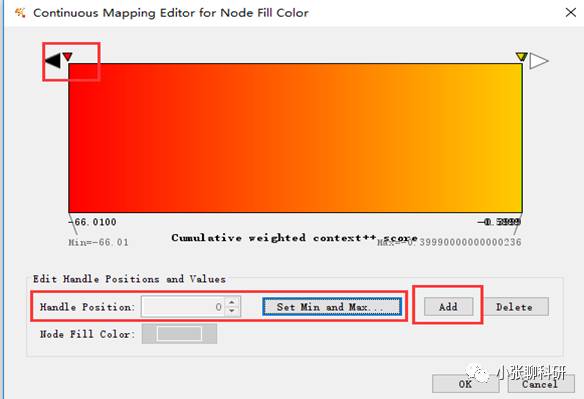
小三角设置色阶颜色,Handle Postion设置色阶对应的数值,Add增加色阶数量。(这里需要注意一点,Cumulative weighted context++ score的最小值是-66.01,最大值是-0.4,我们设置的范围略宽于这个范围为好,保证每个Nodes的颜色都能正常显示)
然后再编辑形状,mRNA选择圆形,microRNA选择菱形。
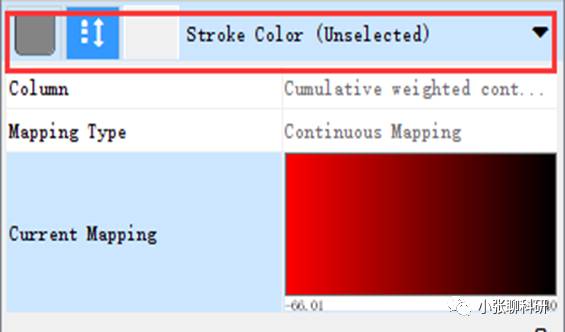
之后再编辑Edges的颜色:
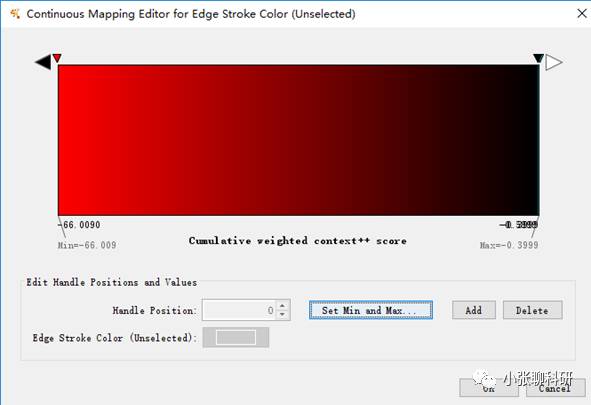
选择设置Edges的颜色,Column选择Cumulative weighted context++ score,颜色选成渐变,然后编辑色阶。
做完是这样的:
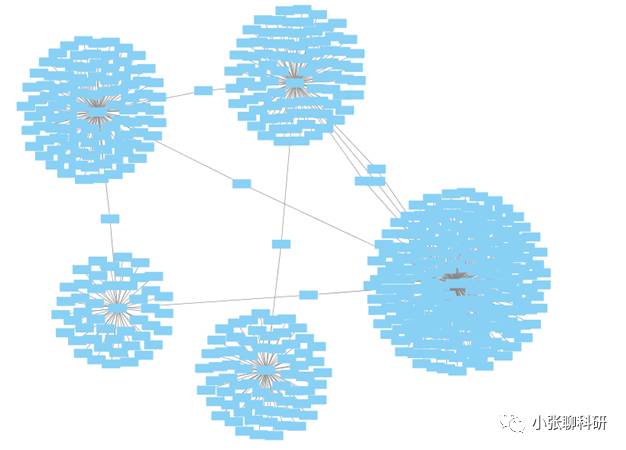
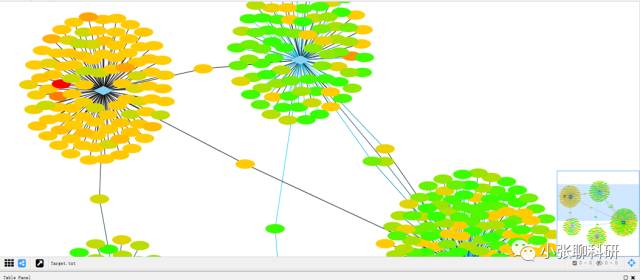
最后效果如下:
当然了,我们还可以设置和2个microRNA相关的mRNA的圆圈大小,从而更进一步突出我们的筛选结果。
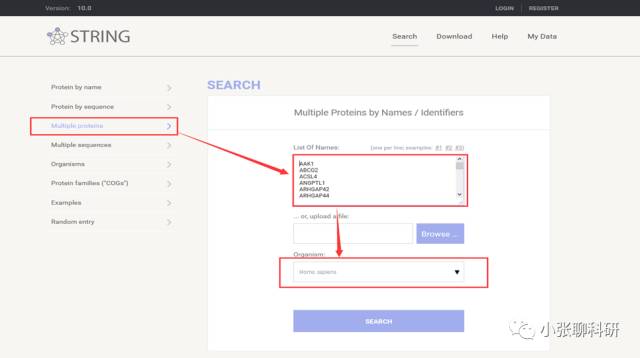
下面我们讲一讲从STRING数据库(http://www.string-db.org)得到分析结果之后的作图。
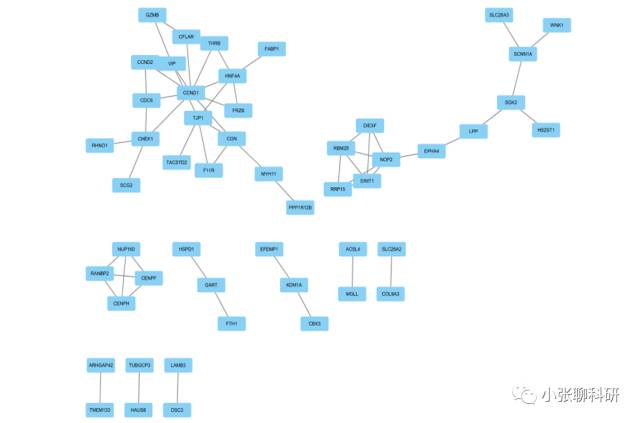
首先从GEO上找了两个数据包,分别筛选差异基因再取交集,一共得到163个差异基因,丢到STRING数据库里进行分析:
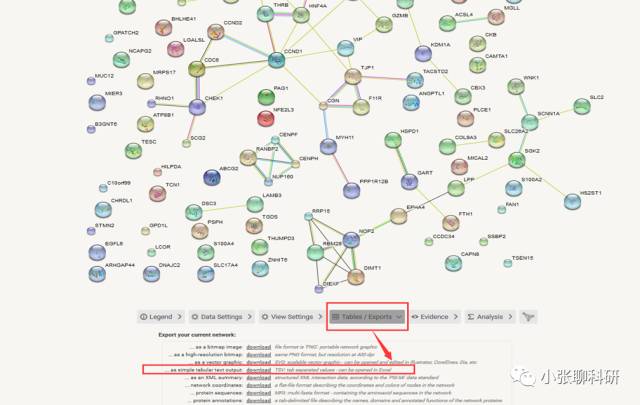
得到下面的图,下载下面标注的那个TXT文档,导入到Cytoscape里:
导入后得到下面的图:
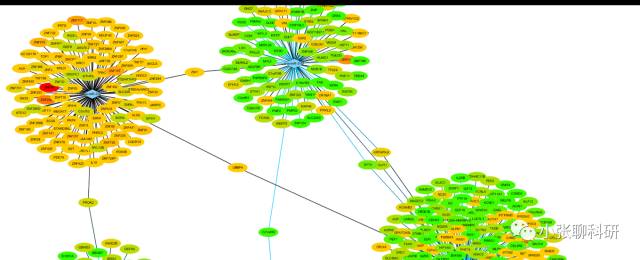
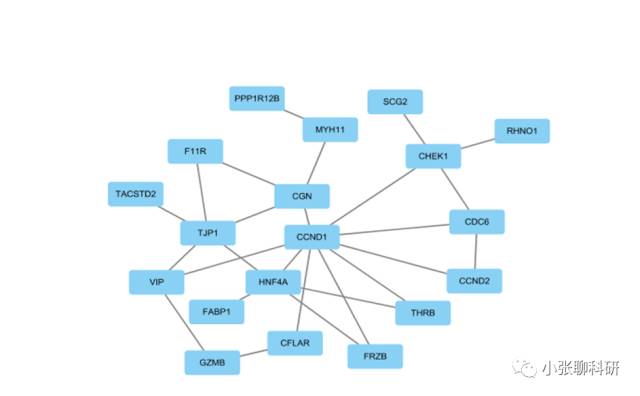
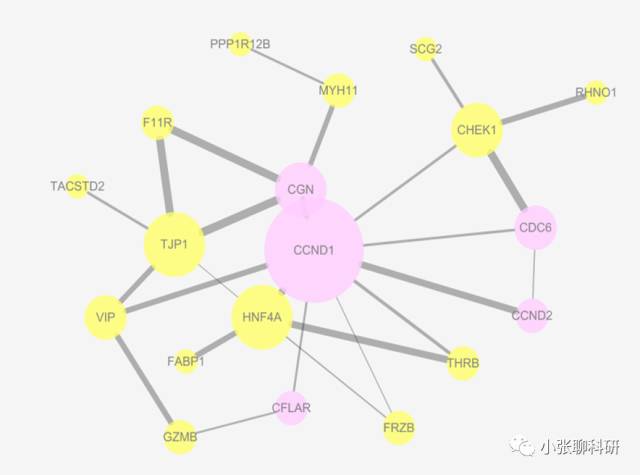
假设我们比较关注以CCND1为中心的Network,我们先把别的不相关的都删掉,再调整下各个Nodes的位置:
这里我们对Nodes和Edges的作出如下注释:1、Edges的粗细由combined_score渐变调整;2、mRNA的上下调分别注释为粉色和黄色;3、Nodes的大小由相互作用的Count数决定。
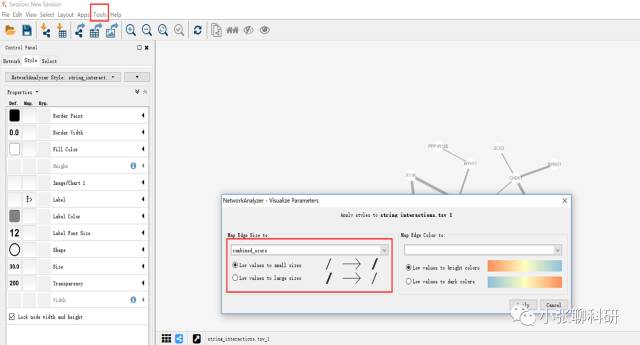
首先选择Tool里的Network Analysis工具中的Generate Style from Statistics:
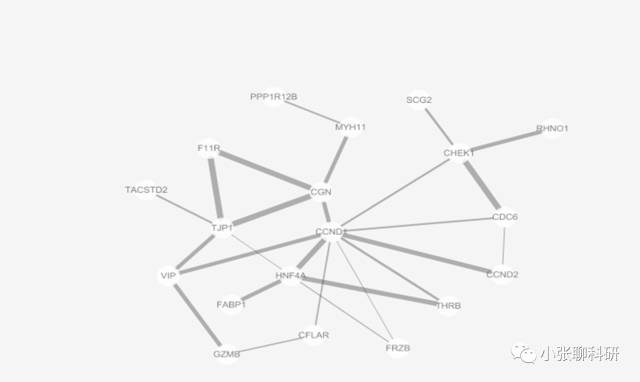
如下图粗细做好了,然后做Nodes的颜色和大小:
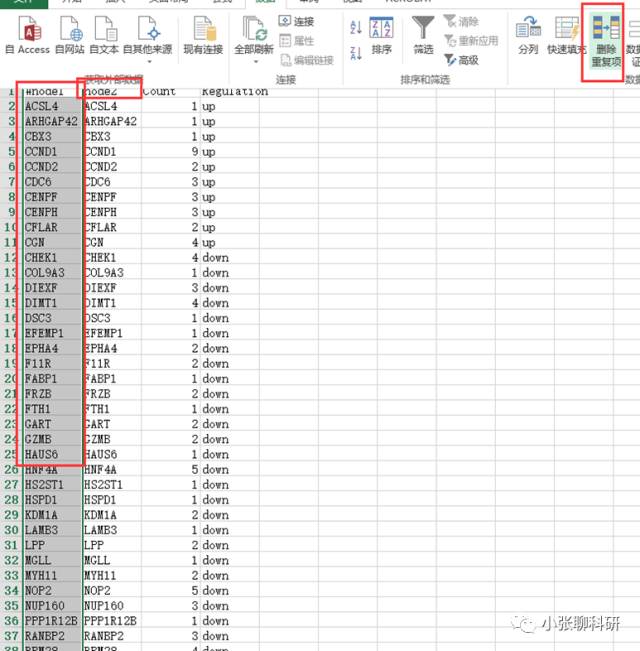
将TXT文件复制到EXCEL里处理一下,需要对Nodes1增加Count和Regulation两列的注释(Regulation也可以根据Fold Change的数值进行颜色渐变处理)。
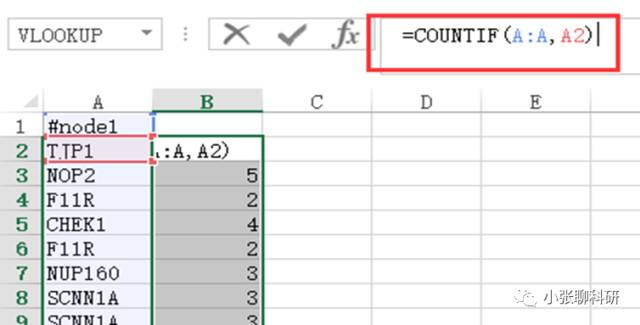
Count的计算如下图,将Nodes1和Nodes2复制到一列中,然后利用=COUNTIF(A:A,A2)的公式进行计算
增加上下调关系,然后选中第一列删除重复项,将处理完的数据保存为TXT文件作为Table导入到Cytoscape中(这里要注意的是还要添加一列Nodes2将其注释进去):
根据Regulation调整节点颜色,根据Count渐变调整Nodes大小。调整完结果如下:
小结:Cytoscape主要是通过Edges连接Nodes构成Network,通过导入Table对Network中的Edges和Nodes进行注释,从而得到我们想要的效果,导入的Table则源于我们对数据的处理,所以数据的处理分析是Cytoscape作图的重要前提。好了,今天Cytoscape的作图就扯到这了,以后会给大家讲讲CO-EXPRESSION的作图。
1.Pahl, M.C., et al., MicroRNA expression signature in humanabdominal aortic aneurysms. BMC Med Genomics, 2012. 5: p. 25.
2.Mokutani,Y., et al., Down-Regulation ofmicroRNA-132 is Associated with Poor Prognosis of Colorectal Cancer. AnnSurg Oncol, 2016. 23(Suppl 5): p.599-608.