Velvet简单介绍:
velvet是一款常用的基因组拼接软件,与soapdenovo一起常用于二代测序数据的处理,相对于soapdenovo拼接的完整性更好。他能同时支持fasta、fastq格式的数据,同时支持多个文库的数据同时使用。
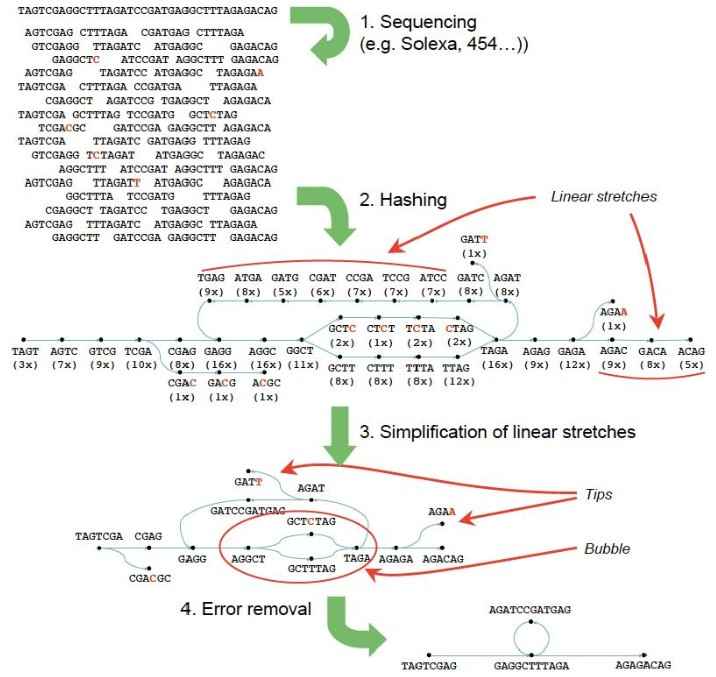
其工作的一般过程简化为:输入short read序列,排除错误,产生高质量的contigs。然后用paired-end read和long read信息,检索contigs之间的重复区域。
Velvet工作原理:
1、Bruijn算法
给定的数据总是存在着冗余信息,因此,算法的使用很重要。当前大多数是用的overlap-consensus-layout算法,它的每一个read都是单独的实体。而Bruijn算法,将分析基于observed words(或者k-mers)。给定k-mers,不管发现多少次,都是唯一的node。此外,他可以无差别的容纳long和short read的混合物。
构建Bruijn图关键在于对所有的reads进行hash。Hash值通过设置参数给定,或者是默认的21。这对所有的序列进行成对比对时会减少时间,一旦所有的reads已经被hash,由k-mer node可以追溯他们的每一个路径。增值覆盖率和创建arcs就属于这种方法。
2、错误移除
错误移除算法:Tour Bus,对没有错误连接的graph进行移除。这要确保基因组中低覆盖率的唯一的点没有被破坏。因此,这一步在画图之后进行。
错误的类型主要有:
- Tips:起源于低质量的read的结尾部分没有任何的重叠。
- Bubbles:他产生于长read的中间或两个错误的read结尾部分重叠。
首先去除tips,由长度和覆盖率来定义。然后去除bubbles,运用的是与Dijkstra’s类似的算法,最后低覆盖率的点会被去掉。
使用方法
在前面的文章中已经介绍了velvet的安装方法(https://www.plob.org/article/199.html)。
1.准备输入数据
无论是fasta格式的数据还是fastq格式数据,首先利用velvet自带的两个脚本程序对每一个pair-end数据进行合并;方法如下:
对于fasta格式数据
shuffleSequences_fasta.pl s1_1.fasta s1_2.fasta s1.fasta
最后的得到的数据格式如下(成对的正反向reads连续放在一起):
>2:1:3789:2197:Y/1
CAGACTAATACCAATCCAACCCATTTCATGAATAAAATGTGACCAATTAACCAACCAACAAAACTGCTTGTTACAAATAA
>2:1:3789:2197:Y/2
ATACAATAACTGTACTAGCTCTACCGTATCTTTTGTTTCATTTCTTCTGGAATAATCACAAAAACTTTTTTGATTATGGA
>2:1:4209:2183:Y/1
TTATAGAAAAGAAAGAAAAGAATGAAAAATAAAGTAATTTTTGATTGGGCCGGGAATCCTATTTTGGATTCGGTATGGAT
>2:1:4209:2183:Y/2
ATCTTTCGCCTATAAATTCAATGAATGAATTACCTCTCAATGATCTTGAATCGGATCAAGATCATGAATAACAATATCTG
>2:1:6191:2192:Y/1
AAAGCATTCACTCCTCTTTTATATCTTGCATCTCTAGTATTTTTGCCCTGGTGGATTTCTCTTTCATTTAATAAAAGTCT
>2:1:6191:2192:Y/2
ATTCCTATACGAAGGTTTTGTAGATGTGTCTCCGAGTATTCCTTGATCATTTCCTCCAAGGGAAGGATTTCCTCTAATTC
对于fastq格式数据
shuffleSequences_fastq.pl s1_1.fq s1_2.fq s1.fq
得到的数据格式与上面类似,只是数据是fastq格式而已,以上得到的s1.fasta或s1.fq可以直接被后续使用。
2.格式化reads
Velveth用一些序列文件产生一个hashtable,并输出两个文件:Sequences和Roadmaps。一般使用以下命令即可,蓝色标记的部分可以重复,即当你有多个文库的时候都按照蓝色部分的格式来写
./velveth directory hash_length { [-file_format] [-read_type] [filename] } [options]
directory:输出文件所在路径的名字(即创建一个文件夹存放结果文件)
hash_length:也叫k-mer length,默认为31bp,储存hash length,值越大,内存需求越大
filename:标准输入文件名
Options:
-strand_specific:转录组序列数据,默认为off
支持的文件格式:fasta,fastq,fasta.gz,fastq.gz,eland,gerald。默认为fasta
读类别:short,shortPaired,short2,shortPaired2,long,longPaired。默认为short
例子1:
> ./velveth output_directory/ 21 –fasta –short solexa1.fa solexa2.fa solexa3.fa –long capillary.fa ;
默认参数为fasta、short,因此可以写成:
>./velveth output_directory/ 21 solexa*.fa –long capillary.fa
例子2:
假如你用一个标准的转录组序列来做,为使结果更好,可用一下选项(-strand_specific)。
> ./velveth output_directory/ 21 (...data files...) -strand_specific
3.序列组装
Velvetg是velvet的核心,他建立bruijn图并对reads进行组装。
./velvetg directory [options]
directory:工作路径名
Standard options:
-cov_cutoff <floating-point|auto>:移除低覆盖率的node,默认不移除
-ins_length <integer>:two paired end reads之间的期望距离,默认no read pairing
-read_trkg <yes|no>:在集合中对short read位置进行跟踪,默认不跟踪
-min_contig_lgth <int>:导出到contig.fa文件中的最小contig长度,默认为hash长度的2倍
-amos_file <yes|no>:导出到AMOS文件中,默认不导出(no)
-exp_cov <floating point|auto>:唯一区域的期望覆盖率
Advanced options:
-ins_length2 <int>:两个paired-end reads在第二个short-read数据集中的期望距离,默认否
-ins_length_long <integer>:两个long paired-end reads的期望距离,默认否
-ins_length*_sd <int>:数据集的标准差,默认corresponding length的10%(*代表:nothing,2,_long)
-scaffolding <yes|no>:scaffolding of contigs used paired end information (default: on)
-max_branch_length <integer>:在bubble中的碱基对的最大长度,默认100-max_divergence <floating-point>:在一个bubble中的两个分支的最大分歧率,默认0.2
-max_gap_count <integer>:在一个bubble中的两个分支比对中允许的最大gap值,默认3-min_pair_count <integer>:构成两个长contigs的paired end的最小值,默认10
-max_coverage <floating point>:在tour bus后移除高覆盖率的node
-long_mult_cutoff <int>:合并contig的long reads的最小值,默认2
-unused_reads <yes|no>:将不用的reads导出到UnusedReads.fa文件中,默认否
-alignments <yes|no>:导出一个主要的contig并和参照序列对其,默认否
例子3、
设置覆盖截断值:
> ./velvetg output_directory/ -cov_cutoff 5.2
排除最高的覆盖数据,设置最大覆盖截断值:
> ./velvetg output_directory/ -max_coverage 300 (... other parameters ...)
自动设置覆盖截断值:
> ./velvetg output_directory/ -cov_cutoff auto
例子4、
如果你有一个足够覆盖面的短read,有任意数量的长read,你可以用长read做repeat。为了达到他,velvet需要对短read的序列有一个期望覆盖的适当的估计。简单的方法是重叠群被发现的分布,也要看覆盖点的聚类情况。如果期望覆盖是19x:
> ./velvetg output_directory/ -exp_cov 19 (... other parameters ...)
假如你有理由相信合理的统一的覆盖你的sample,你可以要求velvet估计他:
> ./velvetg output_directory/ -exp_cov auto (... other parameters ...)
例子5:
Minimum long read connection cuto?:对于用long reads拼接完成contigs,需要设置一个阈值来减少误差(默认为2)。假如你的长序列没有与之重叠的序列(例如contigs),你可以设为0,对他直接进行使用。相反的,如果你的长序列覆盖范围很高,就需要设一个高的阈值。
Minimum read-pair validation:假如你的数据不是paired-end reads,只是偶然成对,那么为减少误差,就需要你在对两个contigs进行拼接时至少要有10对pairs(默认)。
> ./velvetg output_directory/ -min_pair_count 20 (...other parameters...)