关于对照
说到实验设计就不得不提到对照。从小老师教育我们,没有对照组的实验是不完整的,或者说就没有任何意义。所有的生化湿实验都很讲究完整的正对照和负对照。当然,下面的实验我都没做过。。。
蛋白相关实验真真假假的事情太多。要把蛋白和已知相关的蛋白放在一起, 和已知不相关的放在一起, 检验实验手段是否能够区分这两种情况。所谓的正负对照,就是告诉别人我这个实验本身做的没问题。如果再往下做还要通过移除一个蛋白, 来看另一个蛋白的生理表现,看是不是"没有对方就活不下去",是不是可能有其他的couple。
比如GST pull down 实验 ,如果要证明A蛋白和B蛋白相互作用,做SDS-PAGE要有一个纯粹的B蛋白溶液(input)作为阳性对照,必须看到条带说明B蛋白是真实存在的而且可以显色;还需要一个纯粹的GST蛋白作为阴性对照,必须看到纯粹的GST蛋白是无法拉下B蛋白的。真正的实验里面是融合GST标签的A蛋白去拉B蛋白,如果有条带,说明A,B蛋白的确有相互作用。
ChIP 中的对照
ChIP实验,ChIP实验的对照可以说是挺麻烦的一件事情。
首先是input 对照,Input对照不仅可以验证染色质断裂的效果,还可以根据Input中的靶序列的含量以及染色质沉淀中的靶序列的含量,按照取样比例换算出ChIP的效率。
然后是阳性对照需要使用阳性抗体,一般用anti-RNA Polymerase II抗体,因为RNA Polymerase II是通用转录因子,在所有细胞中都能结合基因(为了保证阳性对照有效,一般选择阳性对照的引物是根据管家基因设计的)的核心启动子区,因此,理论上ChIP后PCR都会有条带。
最后阴性对照需要用阴性抗体,用普通的IgG做为抗体,理论上不会ChIP下来任何DNA片段,但是由于非特异结合,或者实验过程中没发生结合的DNA清除不完全,可能也会出现比较浅的条带。
注意, 上述所谓的阴性阳性对照都是针对具体的一次ChIP PCR实验而言的,只能说明这个实验做的有没有物理上的问题。
但是如果想说明的问题是对照组中某个基因的组蛋白修饰相比于实验组有个明显的变化,这个时候还需要另外一个基因或者位点作为负对照。
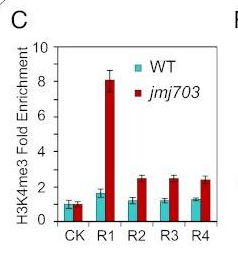
如果想研究的是一个组蛋白修饰,那么需要找一个在实验组和对照组中都有该修饰且修饰表达量不受影响的基因当作ChIP qpcr的对照。用来说明目的基因测出来的变化是有意义的。如下图所示的CK就是一个负对照基因。(类似于基因定量中的actin 功能)文章中的描述为
Anti-H3 was used as an internal reference for ChIP -qPCR. Os04g22450 was used as a negative control
如果做的是某一个转录因子的结合情况,通常你可以在一个基因的不同位置(非启动子区)设计引物进行对照。也就是说在实验组之中也要有对照。其通常要选三个不同的位点来说明问题。一般启动子区域会有变化,而基因的其它区域是没变话的。
PS: 果子评价说,转录因子结合启动子的chip实验,除了正负对照说明实验没有问题,会使用10%input作为内参。
ChIP-seq 中的对照
ChIP-seq 为什么要有对照?因为通过超声处理DNA片段并不是一个真正随机化的过程,一些开放的染色质区域在破碎的样品中会非常多。而且不同的测序平台测出的东西也有差别。
An appropriate control data set is critical for analysis of any ChIP-seq experiment because DNA breakage during sonication is not uniform. In particular, some regions of open chromatin are preferentially represented in the sonicated sample There are also platform-specific sequencing efficiency biases that contribute to nonuniformity
不同于ChIP实验,一个完整的ChIP-seq数据通常应该包含下面两个对照之一。
- DNA is isolated from cells that have been cross-linked and fragmented under the same conditions as the immunoprecipitated DNA (“Input” DNA)
- a “mock” ChIP reaction is performed using a control antibody that reacts with an irrelevant, non-nuclear antigen (“IgG” control).
所谓input 是指断裂后的基因组DNA,它需要与沉淀后的样品DNA一起经过逆转交联,DNA纯化,以及最后的PCR或其他方法检测,但是不进行免疫沉淀。任何HA-tagged protein对染色质状态的影响都会在input中呈现出来。
如果是input chip-seq,结果中会不会有我们想要看到的真的位点呢?
是有的,因为它就是整个基因组上被打断的未经特异性选择过的随机(理论上)片段。如果用PCR来做的话,也同样可能在我们想看的位置看到条带,因为它有基因组上的所有位置。但是,在ChIP-seq中,input阳性位点的相对表达量应该比我们的实验组中低得多得多,毕竟实验组的那些位点是我们特意富集出来的结果。通过Input对照排除因本底表达水平高或一些非特异性结合所造成的假阳性peaks。
而所谓的lgG mock 是说找非特异抗体作为阴性对照,其理论上不会ChIP下来任何DNA片段。但是近来也有很多文章认为用它是有问题的,最好还是用input来作为对照。
如果没有input对照怎么办呢?其实通过对大量的公用数据input组进行分析,我们可以发现在拟南芥中总有那么一百多个位点,不管是什么实验的input都会出现peaks,通常我们会把这些位置列为黑名单不予考虑。
(每个物种的peaks黑名单都是可以谷歌搜索到下载的)
参考资料
Getting Started with ChIP-Seq
ChIP-Seq: advantages and challenges of a maturing technology
ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia
最后一篇文章专门讨论了ChIP-seq实验中的各种建议和问题,可以参考。