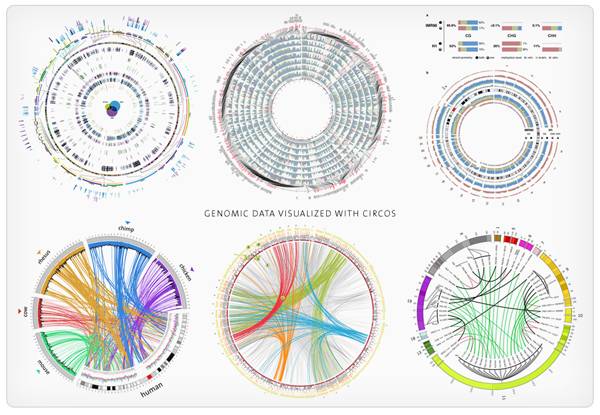
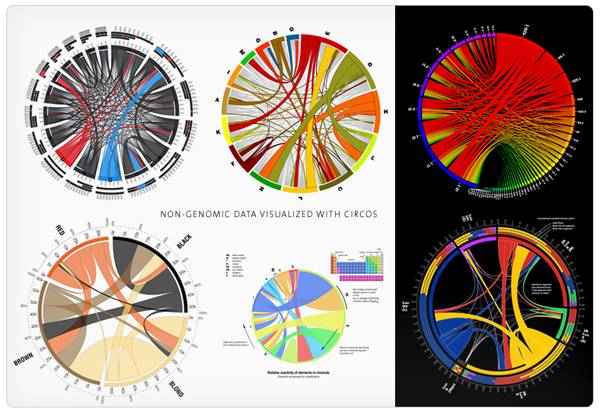
Circos是绘制圈图的神器,在http://circos.ca/images/页面有很多CIRCOS可视化的示例。
Circos可以在线使用,在线使用时是把表格转为圈图,不过只允许最大75行和75列;做一些简单的示意图会比较好,最后时会介绍下在线的tableviewer的使用。
也可以安装在本地,在本地可以绘制基于基因组的更复杂的图。
Circos由Perl写成,安装相对简单,只要Perl的包都装好了就可以了。
Circos安装
- 从http://circos.ca/software/download/circos/下载Circos安装包,并解压,把安装包内的bin目录加载到环境变量。
- wget http://circos.ca/distribution/circos-0.69-6.tgz
- tar xvzf circos-0.69-6.tgz
- ln -s `pwd`/circos-0.69-6/bin/* ~/bin #make sure ~/bin is in $PATH
- or
- # 注意pwd两侧的反引号
- export PATH=$PATH:`pwd`/circos-0.69/bin
- 安装依赖的Perl包
配置CPANM (CPANM是一个文件,下载下来,增加可执行属性,放到环境变量中即可使用)
- # 若无根用户权限,也可放入自己家目录下在环境变量内的目录中就可以
- wget https://raw.githubusercontent.com/miyagawa/cpanminus/master/cpanm -O /sbin/cpanm
- chmod +x /sbin/cpanm
获得依赖的Perl包,circos -module
- ok 1.11 Carp
- missing Clone
- missing Config::General
- ok 3.3 Cwd
- ok 2.124 Data::Dumper
- ok 2.55 Digest::MD5
- ok 2.77 File::Basename
- ok 3.3 File::Spec::Functions
- ok 0.22 File::Temp
- ok 1.50 FindBin
- missing Font::TTF::Font
- missing GD
- missing GD::Polyline
- ok 2.38 Getopt::Long
- ok 1.14 IO::File
- missing List::MoreUtils
- ok 1.21 List::Util
- missing Math::Bezier
- ok 1.60 Math::BigFloat
- missing Math::Round
- missing Math::VecStat
- ok 1.01_03 Memoize
- ok 1.17 POSIX
- missing Params::Validate
- ok 1.36 Pod::Usage
- missing Readonly
- missing Regexp::Common
- missing SVG
- missing Set::IntSpan
- missing Statistics::Basic
- ok 2.20 Storable
- ok 1.11 Sys::Hostname
- ok 2.0.0 Text::Balanced
- missing Text::Format
- ok 1.9721 Time::HiRes
获取缺失模块并安装
- # 第一句话的目的只是查看需要运行的命令,直接运行第二句就好
- circos -module | grep 'missing' | awk '{a=a" "$2;}END{print "cpanm"a}'
- circos -module | grep 'missing' | awk '{a=a" "$2;}END{system("cpanm"a);}'
再运行circos -module发现GD和GD::Polyline没安装上,查看错误信息是No package 'gdlib' found
- # 根用户
- yum install gd-devel
然后再用cpanm安装GD和GD::Polyline。
最简单出图
把下面的内容存储到任意目录下的任意文件比如ehbio.conf下,然后运行circos -conf ehbio.conf就可以获得circos的图circos.png和circos.svg。
- karyotype = data/karyotype/karyotype.human.txt
- <ideogram>
- <spacing>
- default = 0.005r
- </spacing>
- radius = 0.9r
- thickness = 20p
- fill = yes
- </ideogram>
- ################################################################
- # The remaining content is standard and required. It is imported
- # from default files in the Circos distribution.
- #
- # These should be present in every Circos configuration file and
- # overridden as required. To see the content of these files,
- # look in etc/ in the Circos distribution.
- <image>
- # Included from Circos distribution.
- <<include etc/image.conf>>
- </image>
- # RGB/HSV color definitions, color lists, location of fonts, fill patterns.
- # Included from Circos distribution.
- <<include etc/colors_fonts_patterns.conf>>
- # Debugging, I/O an dother system parameters
- # Included from Circos distribution.
- <<include etc/housekeeping.conf>>
上述命令是怎么运行的呢?
- # karyotype定义染色体的名字、ID、起始位置信息,是绘制图的根本
- karyotype = data/karyotype/karyotype.human.txt
- # 必须的部分,控制染色体信息显示
- <ideogram>
- # 定义染色体之间的间距,为图形半径的5% (r代表radius,半径)
- <spacing>
- default = 0.005r
- </spacing>
- # 染色体区域的绘制位置,默认所有染色体都处于远离圆心同样距离的位置
- # 这里设置的是图形半径的0.9倍的位置
- # 也可以设置绝对像素值
- radius = 0.9r
- # 染色体区域的宽度,可以是相对图形半径,也可以说绝对像素值
- thickness = 20p
- # 染色体区域填充颜色
- fill = yes
- </ideogram>
- ################################################################
- # The remaining content is standard and required. It is imported
- # from default files in the Circos distribution.
- #
- # These should be present in every Circos configuration file and
- # overridden as required. To see the content of these files,
- # look in etc/ in the Circos distribution.
- # 这些都是引用文件,暂时不去管什么意思,后面用到再逐个解释。
- # 但是绘图时这些必须引用。下面会解释下最关键的引用位置。
- <image>
- # Included from Circos distribution.
- <<include etc/image.conf>>
- </image>
- # RGB/HSV color definitions, color lists, location of fonts, fill patterns.
- # Included from Circos distribution.
- <<include etc/colors_fonts_patterns.conf>>
- # Debugging, I/O an dother system parameters
- # Included from Circos distribution.
- <<include etc/housekeeping.conf>>
最开始看CIRCOS的配置文件时,不理解其是如何查找用到的数据和配置目录时,不过上面配置文件中被注释掉的一句话泄露了天机look in etc/ in the Circos distribution。配置文件和数据文件默认先在当前目录查找,若没有则去CIRCOS的安装目录下查找。
下面列出了CIRCOS搜索配置文件时查找的目录(搜索数据时也类似)
- .
- ./etc
- /MPATHB/soft/circos-0.69-6/bin/etc
- /MPATHB/soft/circos-0.69-6/bin/../etc
- /MPATHB/soft/circos-0.69-6/bin/..
- /MPATHB/soft/circos-0.69-6/bin
不得不敢写CIRCOS的日志输出,很详细,每次都刷屏。当我们运行CIRCOS失败时,看下日志信息,会得到很多提示。
数据和配置文件都在CIRCOS安装目录下,那么先看看它的目录结构吧。
circos安装目录介绍
bin: 目录下是circos可执行程序,加入环境变量即可
data: 目录下有一个文件夹karyotype,里面收录了几个物种染色体信息文件。
- karyotype.chimp.txt karyotype.arabidopsis.tair10.txt
- karyotype.human.hg38.txt karyotype.human.hg19.txt
- karyotype.mouse.mm10.txt karyotype.yeast.txt karyotype.zeamays.txt
这个是绘制CIRCOS图所必须的一个文件 (文件的内容虽然通常是染色体的信息,但不局限于染色体信息,其它的区域信息、时间序列信息都可以使用)
文件内容如下 (#后面是注释,会被忽略)
前两列是固定的,chr -。chr表示定义一条染色体;-表示指定这个区域的父区域,染色体没有父区域,用-代替。
ID是当前区域的名字,其子区域的父区域列都使用这个名字。如果同时绘制多个物种,可在ID中包含物种的代号。
LABEL是当前区域显示的名字。
START END是当前区域的范围,必须是整个的区域。如果想显示部分区域,可在后续配置中修改。
COLOR是当前区域的颜色,CIRCOS为每个染色体有定义好的颜色,存储于etc/colors.conf。除了预先定义了染色体的颜色,还定义了一些颜色变量可以直接使用。
- #chr - ID LABEL START END COLOR
- chr - hs1 1 0 248956422 chr1
- chr - hs2 2 0 242193529 chr2
- chr - hs3 3 0 198295559 chr3
- chr - hs4 4 0 190214555 chr4
- chr - hs5 5 0 181538259 chr5
- chr - hs6 6 0 170805979 chr6
- chr - hs7 7 0 159345973 chr7
- chr - hs8 8 0 145138636 chr8
- chr - hs9 9 0 138394717 chr9
etc目录下是配置文件,前面引用到了3个。
image.conf内容如下
- <<include image.generic.conf>>
- <<include background.white.conf>>
image.generic.conf内容如下,定义了输出的图形的名字、格式、大小等,这些都可以在自定义配置文件,即前面提到的ehbio.conf中覆盖。
- dir = .
- #dir = conf(configdir)
- file = circos.png
- png = yes
- svg = yes
- # radius of inscribed circle in image
- radius = 1500p
- # by default angle=0 is at 3 o'clock position
- angle_offset = -90
- #angle_orientation = counterclockwise
- auto_alpha_colors = yes
- auto_alpha_steps = 5
background.white.conf只定义了背景是白色。
colors_fonts_patterns.conf引用了颜色、字体、预定义的图形文件信息的配置
- <colors>
- <<include etc/colors.conf>>
- </colors>
- <fonts>
- <<include etc/fonts.conf>>
- </fonts>
- <patterns>
- <<include etc/patterns.conf>>
- </patterns>
colors.conf及其引用文件内容摘录如下,利用RGB组合设置了颜色变量、系列颜色和染色体的颜色。(染色体的名字全部使用小写)
- dpblue = 0,153,237
- vdpblue = 0,136,220
- vvdpblue = 0,120,204
- vvlpurple = purples-7-seq-1
- vlpurple = purples-7-seq-2
- gpos100 = 0,0,0
- gpos = 0,0,0
- gpos75 = 130,130,130
- chr1 = 153,102,0
- chr2 = 102,102,0
- chr3 = 153,153,30
patterns.conf定义特殊小图形的信息
- # pattern fills for PNG files
- vline = tiles/vlines.png
- vline-sparse = tiles/vlines-sparse.png
- hline = tiles/hlines.png
- hline-sparse = tiles/hlines-sparse.png
- checker = tiles/checkers.png
- checker-sparse = tiles/checkers-sparse.png
- dot = tiles/dots.png
- dot-sparse = tiles/dots-sparse.png
- solid = tiles/solid.png
housekeeping.conf必须在最上层自定义配置文件中(也就是ehbio.conf)中引用。这个文件名字起的很生物,持家配置,必须要,而且不建议修改。具体内容就不列出了,感兴趣的自己去看。