1 表达式数据格式
1.1 GCT:基因簇文本文件格式(* .gct)
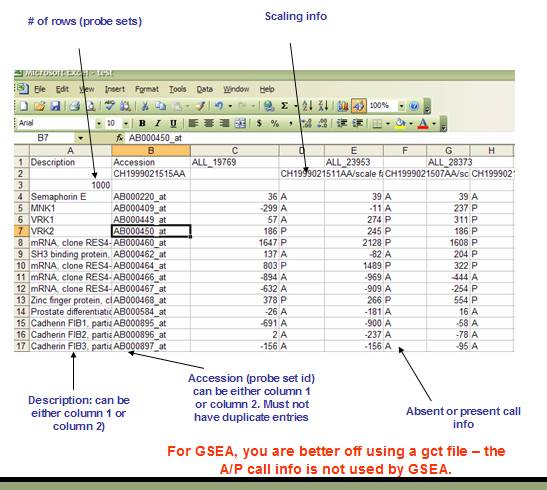
1.2 RES:ExpRESsion(带P和A调用)文件格式(* .res)
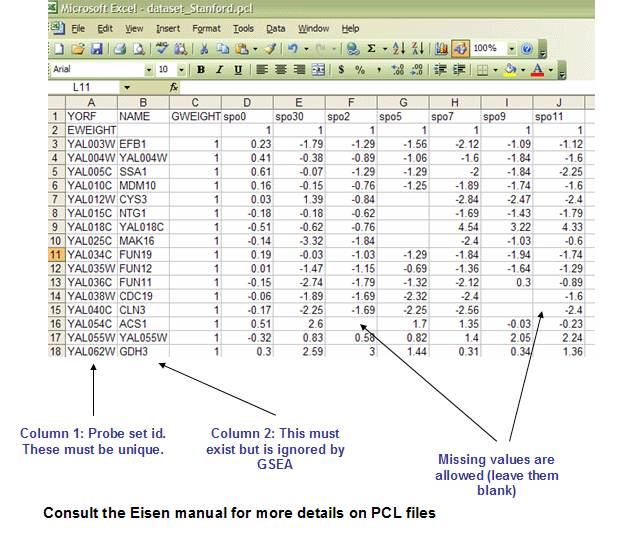
1.3 PCL:斯坦福cDNA文件格式(* .pcl)
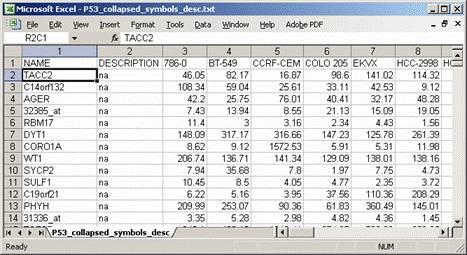
1.4 TXT:表达式数据集的文本文件格式(* .txt)
2 表型数据格式
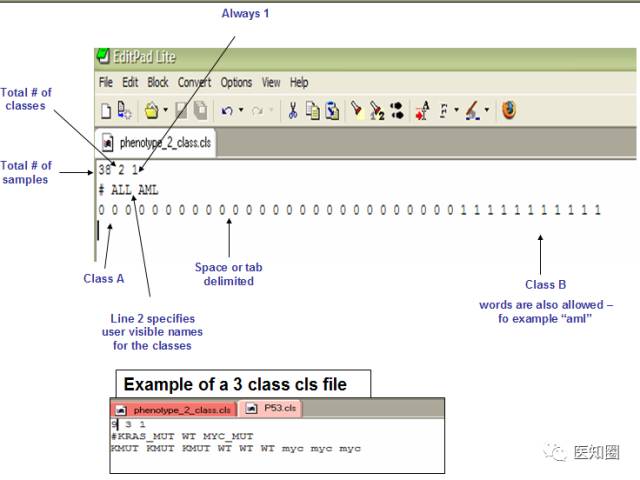
2.1 CLS:分类(例如肿瘤vs正常)类文件格式(* .cls)
2.2 CLS:连续(例如时间序列或基因谱)文件格式(* .cls)
3 基因集数据库格式
3.1 GMX:Gene MatriX文件格式(* .gmx)
3.2 GMT:基因矩阵转置文件格式(* .gmt)
3.3 GRP:基因集文件格式(* .grp)
3.4 XML:分子签名数据库文件格式(msigdb _ *。xml)
4 微阵列芯片注释格式
4.1 芯片:芯片文件格式(* .chip)
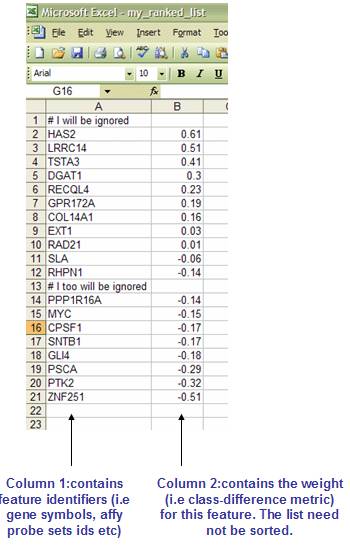
5 排名的基因列表
5.1 RNK:排名列表文件格式(* .rnk)
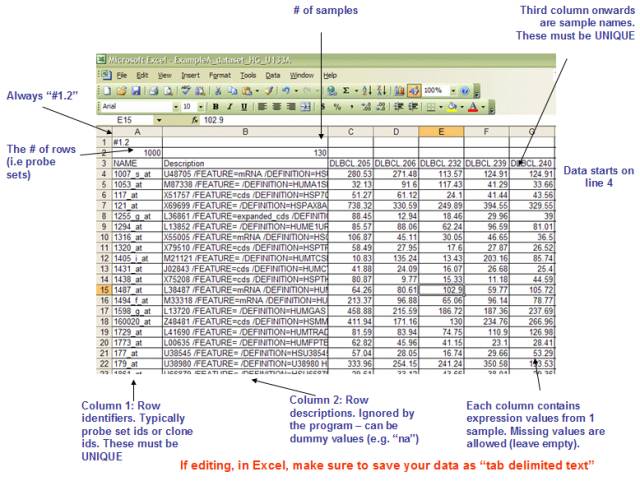
1:GCT格式是一个制表符分隔的文件格式,用于描述表达式数据集。它的组织如下:
RES: ExpRESsion (with P and A calls) file format (*.res)
PCL: Stanford cDNA file format (*.pcl)
TXT: Text file format for expression dataset (*.txt)
CLS: Categorical (e.g tumor vs normal) class file format (*.cls)
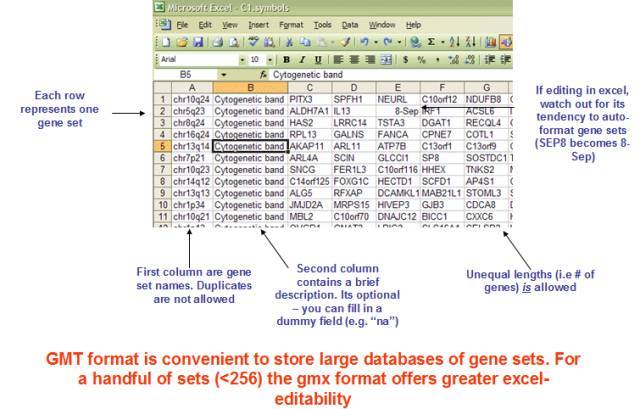
GMX: Gene MatriX file format (*.gmx)
The GMX file format is a tab delimited file format that describes gene sets. In the GMX format, each column represents a gene set; in the GMT format, each row represents a gene set. The GMX file format is organized as follows:
每个基因集由名称,描述和基因组中的基因描述。GSEA使用描述字段来确定报告中提供的基因集描述的超链接:如果描述是“na”,则GSEA提供到MSigDB中指定基因集的链接。
GMT:基因矩阵转置文件格式(* .gmt)
GMT文件格式是描述基因集的制表符分隔文件格式。在GMT格式中,每行代表基因集; 在GMX格式中,每列表示基因组。GMT文件格式的组织如下:
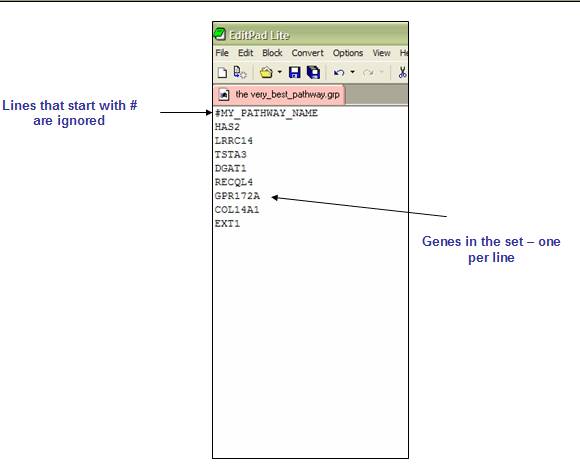
GRP:基因集文件格式(* .grp)
GRP文件以简单的换行符文本格式包含单个基因集。通常,您使用GMT或GMX文件格式创建基因集,而不是使用GRP文件格式。GRP文件格式组织如下:
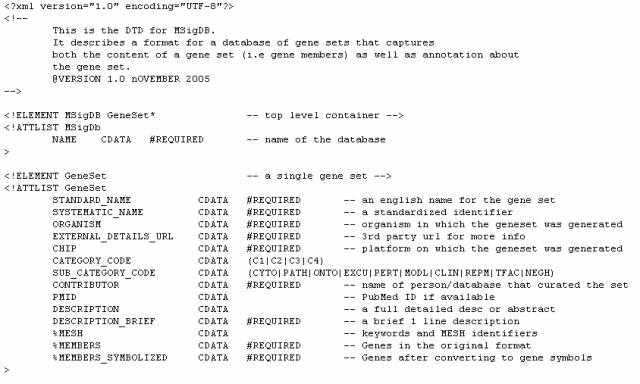
XML:分子签名数据库文件格式(msigdb _ *。xml)
芯片:芯片文件格式(* .chip)
CHIP文件包含有关微阵列的注释。它应列出微阵列中使用的特征(即探针集)及其与基因符号的映射(如果可用)。虽然该文件不是直接在GSEA算法中使用,但它用于注释输出结果,也可用于将表达式数据集中的每个探针集折叠为单个基因载体。
CHIP文件格式的组织如下:
文件名必须以.chip扩展名结尾。
在第一行包含标识在文件的剩余部分每列的内容列标题。该文件必须包含三个列标题,以标签分隔:
- 探头组ID
- 基因符号
- 基因标题该GENE_SYMBOL.chip文件包含一个额外的列别名,这里没有显示。当基因被多个HUGO基因符号识别时,Gene Symbol列包含出现在GSEA报告中的基因符号,“Alias”列识别用于引用相同基因的其他基因符号。如果基因集或芯片注释文件在“别名”列中包含基因,则GSEA会自动将其转换为基因符号列中的基因。文件的其余部分包含微阵列中使用的每个探针组ID的数据。
线格式:(探针集ID)(标签)(基因符号)(标签)(基因标题)
RNK:排名列表文件格式(* .rnk)
RNK文件以简单的换行符分隔的文本格式包含单个排序的基因列表(不是基因集)。当您有要预先排序的排名列表,您要使用GSEA进行分析时使用。例如,您可能已经使用您最喜爱的tTest样统计来从您现在要测试浓缩的数据集中产生排序的排序基因列表。线顺序无关紧要。然而,重要的是,第二列将具有数值 - 它们将用于由GSEA对顺序基因进行排序。