1 聚类分析介绍
1.1基本概念
聚类就是一种寻找数据之间一种内在结构的技术。聚类把全体数据实例组织成一些相似组,而这些相似组被称作聚类。处于相同聚类中的数据实例彼此相同,处于不同聚类中的实例彼此不同。聚类技术通常又被称为无监督学习,因为与监督学习不同,在聚类中那些表示数据类别的分类或者分组信息是没有的。
通过上述表述,我们可以把聚类定义为将数据集中在某些方面具有相似性的数据成员进行分类组织的过程。因此,聚类就是一些数据实例的集合,这个集合中的元素彼此相似,但是它们都与其他聚类中的元素不同。在聚类的相关文献中,一个数据实例有时又被称为对象,因为现实世界中的一个对象可以用数据实例来描述。同时,它有时也被称作数据点(Data Point),因为我们可以用 维空间的一个点来表示数据实例,其中 表示数据的属性个数。
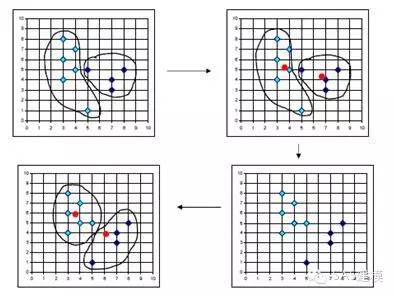
下图显示了一个二维数据集聚类过程,从该图中可以清楚地看到数据聚类过程。虽然通过目测可以十分清晰地发现隐藏在二维或者三维的数据集中的聚类,但是随着数据集维数的不断增加,就很难通过目测来观察甚至是不可能。
1.2算法概述
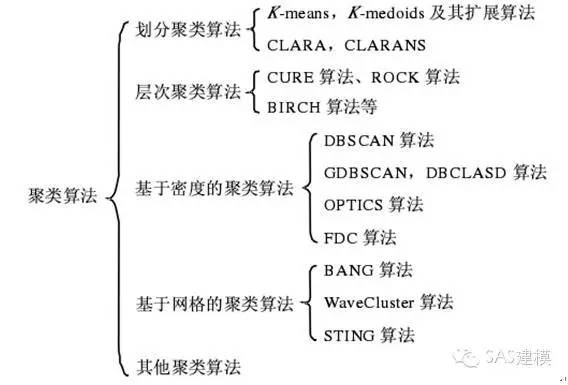
目前在存在大量的聚类算法,算法的选择取决于数据的类型、聚类的目的和具体应用。大体上,主要的聚类算法分为几大类。
聚类算法的目的是将数据对象自动的归入到相应的有意义的聚类中。追求较高的类内相似度和较低的类间相似度是聚类算法的指导原则。
一个聚类算法的优劣可以从以下几个方面来衡量:
(1)可伸缩性:好的聚类算法可以处理包含大到几百万个对象的数据集;
(2)处理不同类型属性的能力:许多算法是针对基于区间的数值属性而设计的,但是有些应用需要针对其它数据类型(如符号类型、二值类型等)进行处理;
(3)发现任意形状的聚类:一个聚类可能是任意形状的,聚类算法不能局限于规则形状的聚类;
(4)输入参数的最小化:要求用户输入重要的参数不仅加重了用户的负担,也使聚类的质量难以控制;
(5)对输入顺序的不敏感:不能因为有不同的数据提交顺序而使聚类的结果不同;
(6)高维性:一个数据集可能包含若干维或属性,一个好的聚类算法不能仅局限于处理二维或三维数据,而需要在高维空间中发现有意义的聚类;
(7)基于约束的聚类:在实际应用中要考虑很多约束条件,设计能够满足特定约束条件且具有较好聚类质量的算法也是一项重要的任务;
(8)可解释性:聚类的结果应该是可理解的、可解释的,以及可用的。
1.3聚类应用
在商业上,聚类分析被用来发现不同的客户群,并且通过购买模式刻画不同的客户群的特征。聚类分析是细分市场的有效工具,同时也可用于研究消费者行为,寻找新的潜在市场、选择实验的市场,并作为多元分析的预处理。在生物上,聚类分析被用来动植物分类和对基因进行分类,获取对种群固有结构的认识。在地理上,聚类能够帮助在地球中被观察的数据库商趋于的相似性。在保险行业上,聚类分析通过一个高的平均消费来鉴定汽车保险单持有者的分组,同时根据住宅类型,价值,地理位置来鉴定一个城市的房产分组。在因特网应用上,聚类分析被用来在网上进行文档归类来修复信息。在电子商务上,聚类分析在电子商务中网站建设数据挖掘中也是很重要的一个方面,通过分组聚类出具有相似浏览行为的客户,并分析客户的共同特征,可以更好的帮助电子商务的用户了解自己的客户,向客户提供更合适的服务。
2 Kmeans算法
2.1基本思想
划分聚类算法是根据给定的n个对象或者元组的数据集,构建k个划分聚类的方法。每个划分即为一个聚簇,并且,该方法将数据划分为k个组,每个组至少有一个对象,每个对象必须属于而且只能属于一个组。该方法的划分采用按照给定的 个划分要求,先给出一个初始的划分,然后用迭代重定位技术,通过对象在划分之间的移动来改进划分。
为达到划分的全局最优,划分的聚类可能会穷举所有可能的划分。但在实际操作中,往往采用比较流行的k-means算法或者k-median算法。
2.2算法步骤
k-means算法最为简单,实现比较容易。每个簇都是使用对象的平均值来表示。
步骤一:将所有对象随机分配到 个非空的簇中。
步骤二:计算每个簇的平均值,并用该平均值代表相应的值。
步骤三:根据每个对象与各个簇中心的距离,分配给最近的簇。
步骤四:转到步骤二,重新计算每个簇的平均值。这个过程不断重复直到满足某个准则函数或者终止条件。终止(收敛)条件可以是以下任何一个:没有(或者最小数目)数据点被重新分配给不同的聚类;没有(或者最小数目)聚类中心再发生变化;误差平方和(SSE)局部最小。
SAS kmeans实现主要是通过proc fastclus过程实现,示例如下:
- proc import datafile="E:\SAS\cars.txt" out=carsdbms=dlm replace;
- delimiter='09'x;
- getnames=yes;
- run;
- proc print data=cars;
- run;
- proc standard data=cars out=stdcars mean=0 std=1;
- var Mpg Weight Drive_Ratio Horsepower Displacement;
- run;
- proc fastclus data=stdcars summary maxc=5 maxiter=99
- outseed=clusterseed out=clusterresult cluster=clusterleast=2;
- id Car;
- var Mpg Weight Drive_Ratio Horsepower Displacement;
- run;
2.3算法分析
k-means算法对于大型的数据库是相对高效的,一般情况下结束于局部最优解。但是,k-means算法必须在平均值有意义的情况下才能使用,对分类变量不适用,事先还要给定生成聚类的数目,对异常数据和数据噪声比较敏感,不能对非凸面形状的数据进行处理。另外,k-means算法在聚类过程中可能有的聚类中心没有被分配任何数据而使得某些聚类变为空,这些聚类通常被称为空聚类。为了解决空聚类问题,我们可以选择一个数据点作为替代的聚类中心。例如,某一个含有大量数据的聚类的聚簇中心最远的数据点。如果算法的终止条件取决于误差平方和,具有最大误差平方和的聚类可以被用来寻找另外的聚类中心。
3层次聚类算法
3.1基本思想
层次聚类主要有两种类型:合并的层次聚类和分裂的层次聚类。前者是一种自底向上的层次聚类算法,从最底层开始,每一次通过合并最相似的聚类来形成上一层次中的聚类,整个当全部数据点都合并到一个聚类的时候停止或者达到某个终止条件而结束,大部分层次聚类都是采用这种方法处理。后者是采用自顶向下的方法,从一个包含全部数据点的聚类开始,然后把根节点分裂为一些子聚类,每个子聚类再递归地继续往下分裂,直到出现只包含一个数据点的单节点聚类出现,即每个聚类中仅包含一个数据点。
层次聚类技术是一种无监督学习的技术,因此可能没有确定的、一致的正确答案。正是由于这个原因,并且在聚类的特定应用的基础之上,可以设计出较少或较多数量的簇。定义了一个聚类层次,就可以选择希望数量的簇。在极端的情况下,所有的对象都自成一簇。在这样的情形下,聚类的对象之间非常相似,并且不同于其他的聚类。当然,这种聚类技术就失去了实际意义,因为聚类的目的是寻找数据集中的有意义的模式,方便用户理解,而任何聚类的数目和数据对象一样多的聚类算法都不能帮助用户更好地理解数据,挖掘数据隐藏的真实含义。这样,关于聚类的很重要的一点就是应该比原先的数据的数目更少的簇。到底要形成多少个聚类数目,要根据实际业务的理解,这是如何解释实际项目的事情。层次聚类算法的好处是它可以让用户从这些簇中选择所感兴趣的簇,这样更具有灵活性。
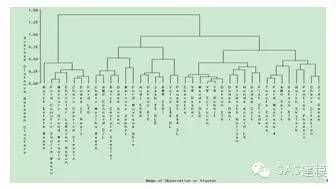
层次聚类通常被看做成一棵树,其中最小的簇合并在一起创建下一个较高层次的簇,这一层次的簇再合并在一起就创建了再下一层次的簇。通过这样的过程,就可以生成一系列的聚类树来完成聚类。单点聚类处在树的最底层,在树的底层有一个根节点聚类。根节点聚类覆盖了全部数据节点,兄弟节点聚类则划分了它们共同的父节点中的所有的数据点。图1-5是采用统计分析软件SAS对Cars数据集进行层次聚类的层次聚类结果图。通过该层次聚类树,用户可以选择查看在树的各个层次上的聚类情况。如图所示。
层次聚类结果图
基于层次的聚类算法方法比较简单,但是缺乏伸缩性,一旦一个合并或者分裂被执行,就不能撤销。为了改进层次聚类的效果,可以将层次聚类算法和其他聚类算法结合使用,形成多阶段的聚类算法。
3.2算法步骤
层次聚类(hierarchical clustering)算法递归的对对象进行合并或者分裂,直到满足某一终止条件为止。层次聚类分为两种,按自底向上层次分解称为聚合的层次聚类,反之,称为分解的层次聚类。层次聚类算法的计算复杂度为O(n2),适合小型数据集的分类。
CURE、ROCK、BIRCH和CHAMELEON是聚合层次聚类中最具代表性的方法。CURE(Clustering Using REpresentatives)算法采用了抽样和分区的技术,选择数据空间中固定数目的、具有代表性的一些点来代表相应的类,这样就可以识别具有复杂形状和不同大小的聚类,从而很好的过滤孤立点。ROCK(RObust Clustering using linKs)算法是对CURE算法的改进,除了具有CURE算法的一些优良特性外,还适用于类别属性的数据。BIRCH(Balanced Iterative Reducing and Clustering using Hierarchy)算法首次提出了通过局部聚类对数据库进行预处理的思想。CHAMELEON是Karypis等人1999年提出的,它在聚合聚类的过程中利用了动态建模技术。
SAS实例
- options nocenter nodate pageno=1 linesize=132;
- title h = 1 j = l 'File:cluster.mammalsteeth.sas';
- title2 h = 1 j = l 'Cluster Analysis of Mammals'' teethdata';
- data teeth;
- input mammal $ 1-16
- @21 (v1-v8) (1.);
- label v1='Top incisors'
- v2='Bottom incisors'
- v3='Top canines'
- v4='Bottom canines'
- v5='Top premolars'
- v6='Bottom premolars'
- v7='Top molars'
- v8='Bottom molars';
- cards;
- BROWNBAT 23113333
- MOLE 32103333
- SILVER HAIR BAT 23112333
- PIGMYBAT 23112233
- HOUSEBAT 23111233
- REDBAT 13112233
- PIKA 21002233
- RABBIT 21003233
- BEAVER 11002133
- GROUNDHOG 11002133
- GRAYSQUIRREL 11001133
- HOUSEMOUSE 11000033
- PORCUPINE 11001133
- WOLF 33114423
- BEAR 33114423
- RACCOON 33114432
- MARTEN 33114412
- WEASEL 33113312
- WOLVERINE 33114412
- BADGER 33113312
- RIVEROTTER 33114312
- SEAOTTER 32113312
- JAGUAR 33113211
- COUGAR 33113211
- FURSEAL 32114411
- SEALION 32114411
- GREYSEAL 32113322
- ELEPHANTSEAL 21114411
- REINDEER 04103333
- ELK 04103333
- DEER 04003333
- MOOSE 04003333
- ;
- proc princomp data=teeth out=teeth2;
- var v1-v8;
- run;
- proc cluster data=teeth2 method=average outtree=ttree
- ccc pseudorsquare;
- var v1-v8;
- id mammal;
- run;
- proc tree data=ttree out=ttree2 nclusters=4;
- id mammal;
- run;
- proc sort data=teeth2;
- by mammal;
- run;
- proc sort data=ttree2;
- by mammal;
- run;
- data teeth3;
- merge teeth2ttree2;
- by mammal;
- run;
- symbol1 c=black f=, v='1';
- symbol2 c=black f=, v='2';
- symbol3 c=black f=, v='3';
- symbol4 c=black f=, v='4';
- proc gplot;
- plotprin2*prin1=cluster;
- run;
- proc sort;
- by cluster;
- run;
- proc print;
- by cluster;
- var mammal prin1prin2;
- run;