时至今日,科研文章也紧随时代潮流,文章中各种高大上的配图层出不穷。究其原因主要包括两点:一是在科研数据和内容更加丰满而文章发表配图张数受限的情况下组合图更具实用性;二是一幅高颜值的配图不仅能提升文章的档次,也更容易获取编辑的青睐。
近两年肠道微生态的研究火的一塌糊涂,我们经常在CNS级别的文章中看到类似下图的进化树。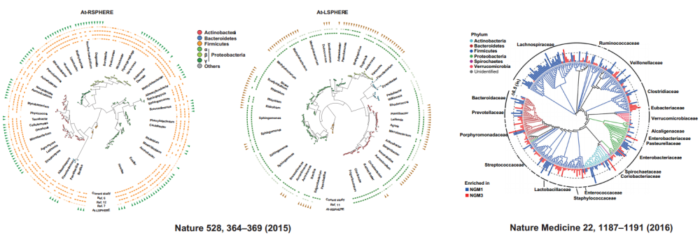
在感慨人家配图碉堡了的同时,不禁思索:哥(姐)的文章要加张这个图...
那么今天小编就来给大家介绍一个简单的在线工具,教你如何绘制高颜值的进化树。
1. iTOL简介
ITOL是Interaction Tree Of Life(网址:http://itol.embl.de/)的简称,它是一个集在线展示、注释和管理进化树的交互工具。绘图过程中可以随意调整树枝、标签的颜色、形状和字体。而iTOL最大的特点是可以同时展示不同的数据集,并按照个性化的需求控制数据集的位置、大小和颜色,并允许导出高质量的位图和矢量图。
2. 输入文件准备
输入文件主要包含两个部分:
1)原始进化树文件
只能识别Newick、Nexus、PhyloXML、Text和Jplace等格式的纯文本文件(plain text files),比如MEGA等软件输出的Newick文件,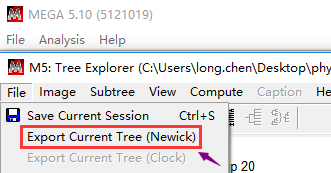
或者i-Sanger云平台(www.i-sanger.com)系统进化树分析产生的.tre文件。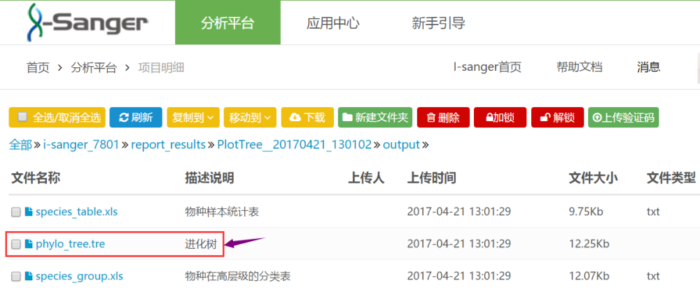
2)进化树注释文件
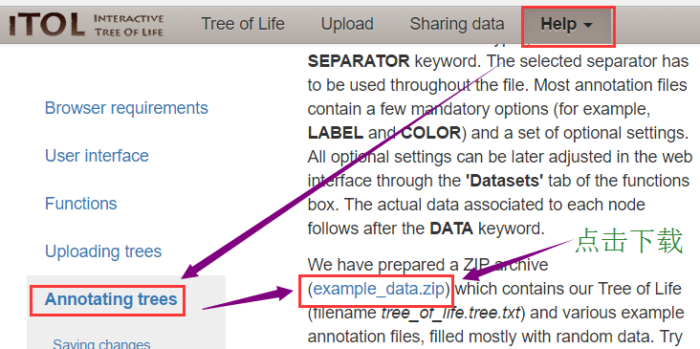
通过下载iTOL官网上提供的进化树不同数据集注释模板(网址:http://itol.embl.de/help.cgi#annot,模板下载参考下图操作),将自己的数据按照模板修改从而获得注释文件。
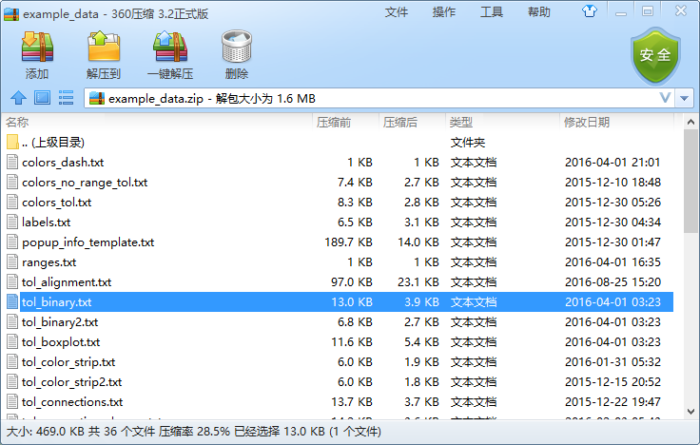
下载的进化树数据集注释模板文件解压后如下所示:
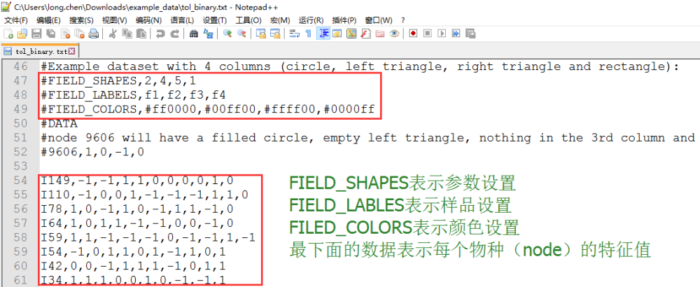
以tol_binary.txt文件为例:使用Notepad++打开该文件,翻到如下图所示界面,对图中的4个参数进行编辑,保存后即可直接用于进化树的注释
3. 输入文件上传
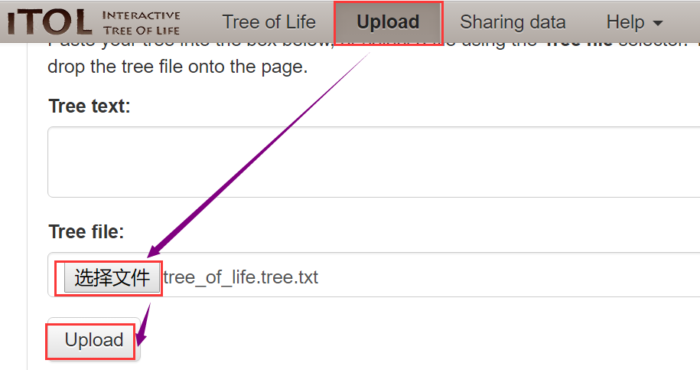
将原始进化树文件按如下图流程进行上传,需要注意的是进化树注释文件直接拖动至进化树绘制面板中即可,无需上传。
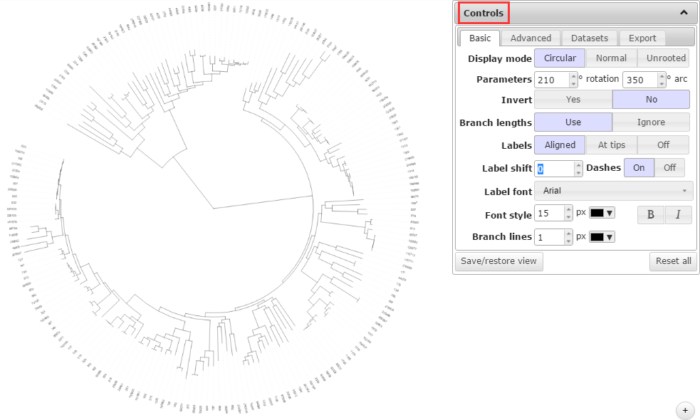
4. 进化树绘制面板介绍
数据上传后,网页会跳转至进化树绘制面板界面。
通过调整控制面板(Controls)中的参数可对进化树进行简单编辑,如调整图形模式(Display mode),物种字体(Label font),树枝粗细,颜色(Branch lines),甚至任意旋转进化树的方向(Parameters)等。比如下图就是经过简单编辑后产生的进化树图
5. 高颜值进化树
现在就让小编来揭晓iTOL最具特色的功能——通过添加各种数据集来嗖嗖地增加你的进化的颜值,直观展示更多重要的数据。咱们一起欣赏一下8种高大上的进化树吧!
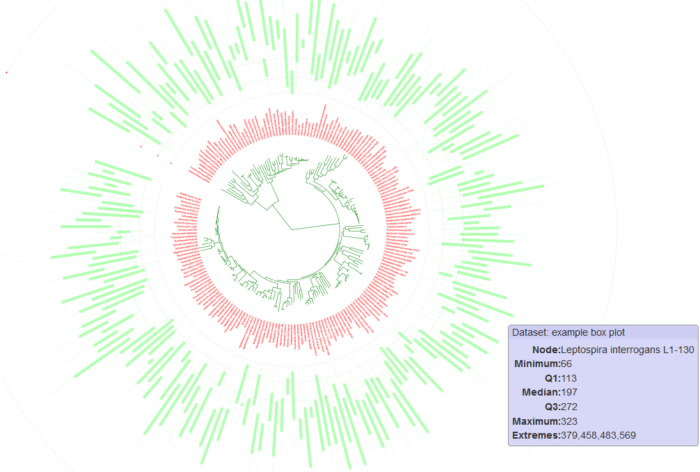
1)进化树与箱线图(tol_boxplot)
箱线图表示样本组不同物种进化分支的相对丰度分布情况。
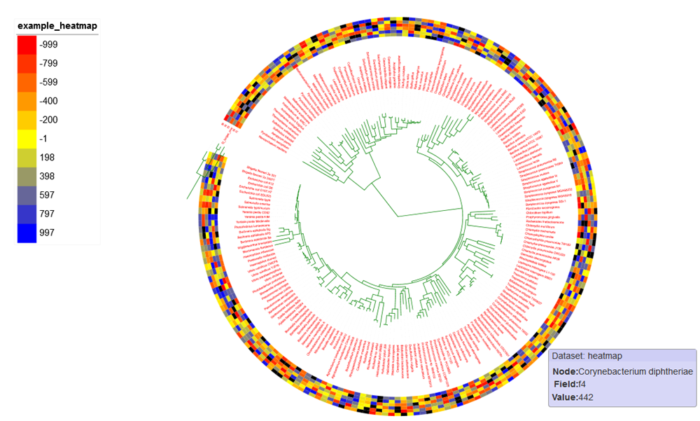
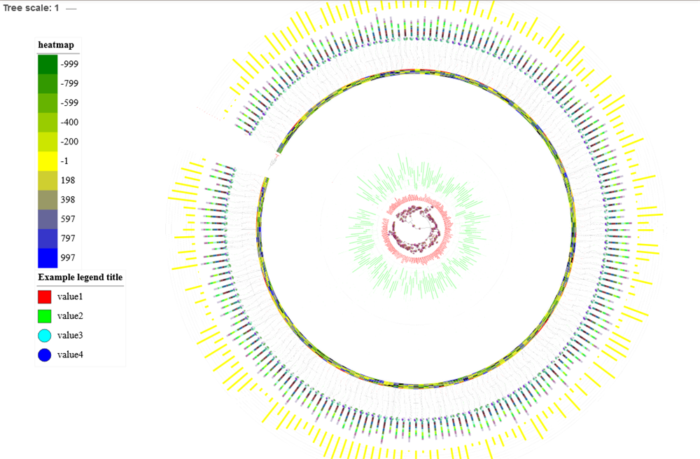
2)进化树与Heatmap图(tol_heatmap)
Heatmap图表示不同物种进化分支在不同(组)样本中的相对丰度分布情况。
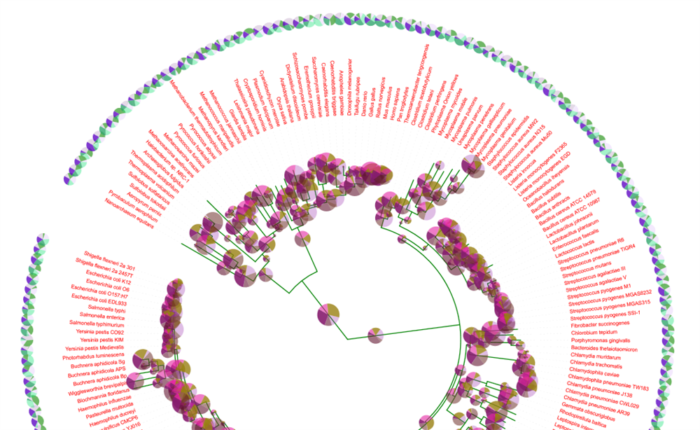
3)进化树与饼形图(tol_pies)
饼形图表示不同物种进化分支在不同(组)样本中的占比信息。
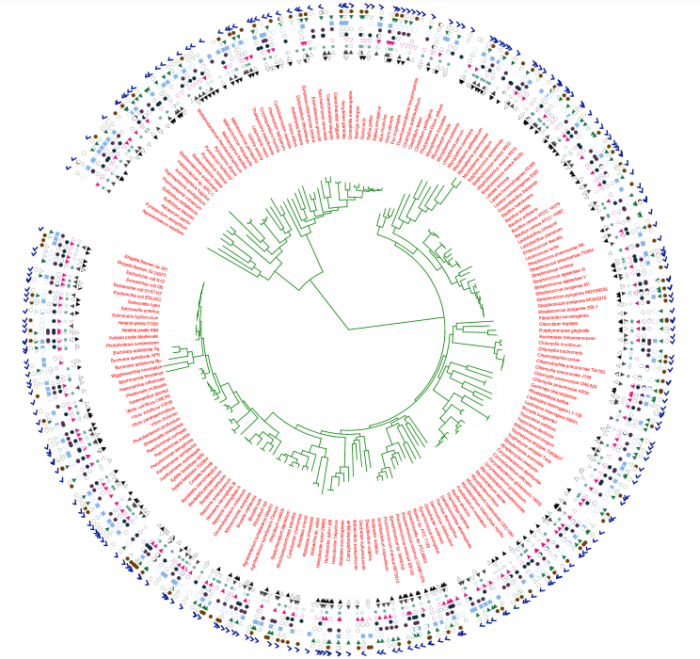
4)进化树与物种分布特征图(tol_binary)
不同的形状代表不同的样品,实心形状赋值为1,空心形状赋值为0,空白位置赋值为-1,实际作图可根据需要进行赋值操作。
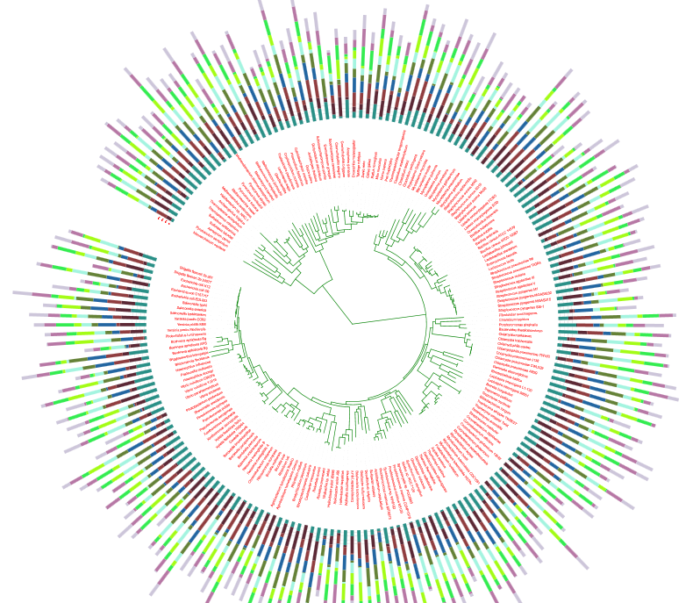
5)进化树与物种丰度堆叠柱状图(tol_multibar)
柱状图中同一颜色代表同一样本(组),柱子的长短代表不同物种的相对丰度大小。
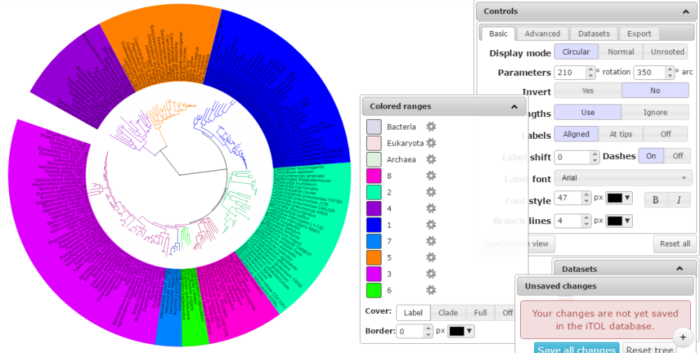
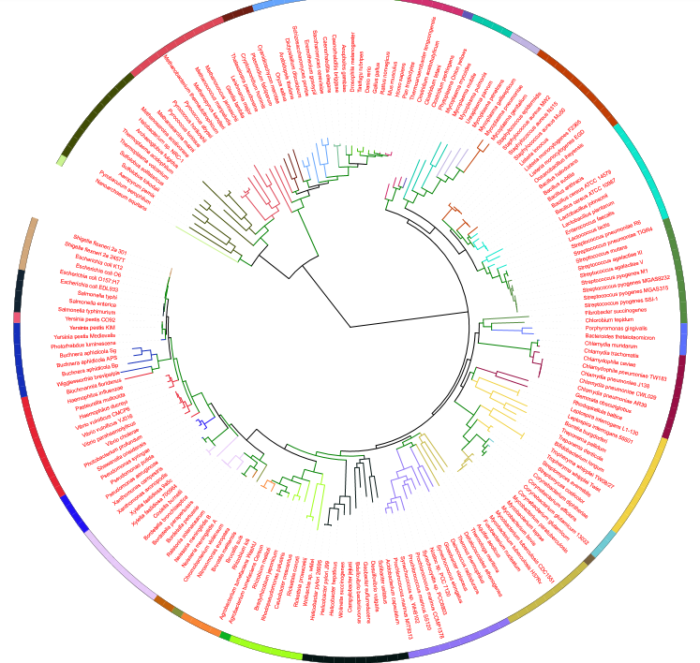
6)分类进化树(tol_color_strip)
同一颜色的进化树枝和最外圈彩带表征某一分类学水平下的同一物种类别,不同颜色代表不同物种类别。
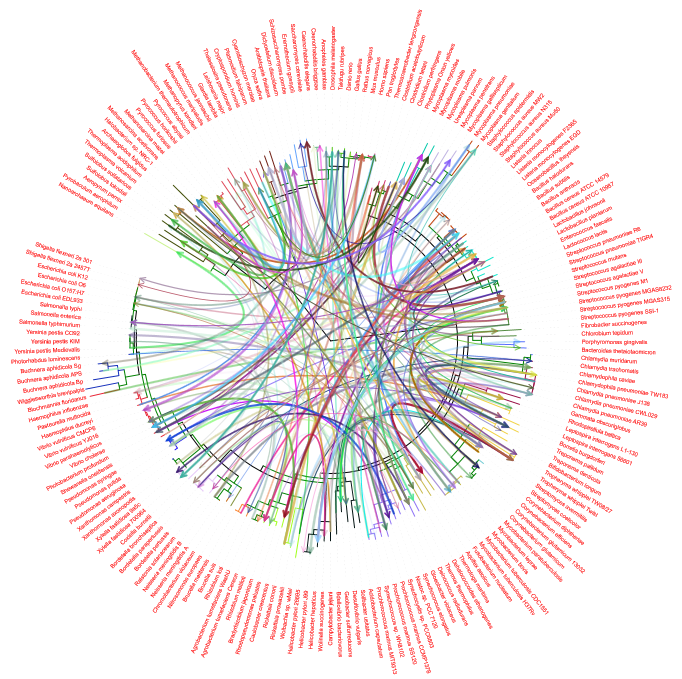
7)物种相关性进化树(tol_connections_leaves)
两端箭头连接的物种具有一定的相关性,线条的粗细可表征相关性数值的大小。
当然iTOL在线工具的功能还有很多,同时也提供相应的在线学习视频,各位感兴趣的看官可登陆官网进行查看和学习。
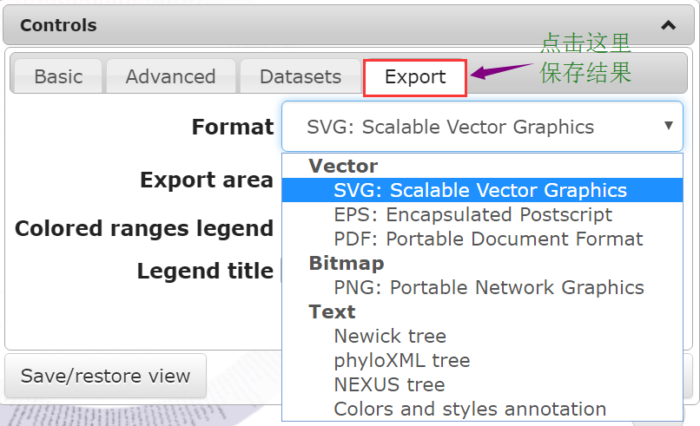
6. 进化树的导出
最后的最后千万别忘了把已经做好的进化树导出来。ITOL提供多种格式图形导出功能包括矢量图(Vector)、位图(Bitmap)和树文件(Text)。
那么