1.背景
- GWAS研究产生了大量的SNP,大部分在非编码基因组
- 这些SNP其实是Lead/Tag SNP,所以需要同时关注与这些SNP处于高LD(linkage disequilibrium)的其它SNP
- ENCODE产生的大量数据,可以用来注释这些SNP,为我们研究这些SNP的功能提供一些参考。
- 本篇介绍的两个数据库RegulomeDB和HaploReg的套路就是将ENCODE产生的注释数据与dbSNP和GWAS Catalog的SNP取交集。
2. 注释SNP的数据
可以将RegulomeDB和HaploReg使用的注释数据分成以下几类:
- 预测该SNP的目标基因:eQTLs(RNA-Seq)
- 是否在基因内部:GENCODE注释文件
- 转录因子结合位点:ChIP-Seq
- 蛋白结合Motif的改变:PWM分数
- 调控元件分类结果(promoter,enhancer等):Chromatin States(来自Roadmap项目)
- 染色质开放区域(open chromatin):DHS区域
- 碱基在人群中比例:1000 Genomes等项目
- 人工注释:Vista enhancer数据库
- 序列保守性:SiPhy软件注释
当我们查看特定的SNP,或者特定的基因组区域,以上返回的结果就能提示值不值得深入研究了。
虽然两个数据库的信息大同小异,但是细节上还有一些不同,比如:
RegulomeDB链接到UCSC Browser,可以查看这些支持性信息的track
HaploReg可以同时展示处于高LD区域的其它SNP的信息
查看完整的注释信息请参考页面下方的官网链接。
3. 用法示例
文献报道rs2816316与腹腔疾病疾病相关
(Hunt, …, van Heel (2008) Nature Genetics和Dubois, …, van Heel (2010)Nature Genetics).
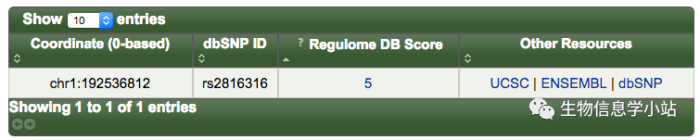
- 在RegulomeDB里查询该SNP。RegulomeDB将所有SNP按照Fig 1的优先性打分。Fig 2说明该SNP与转录调控关系不大。

Fig 1. RegulomeDB的打分规则
Fig 2. rs2816316在RegulomeDB的得分
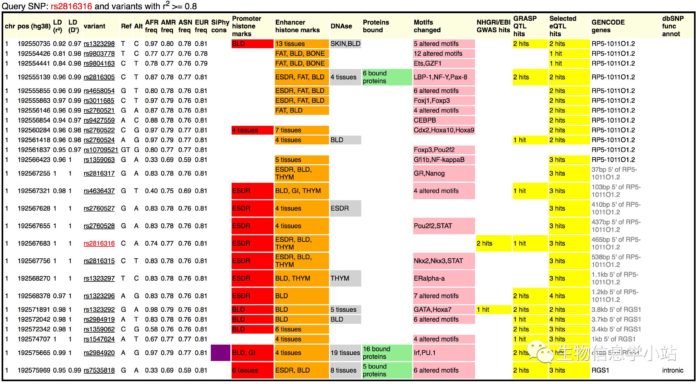
- 在HaploReg里查询,有25个SNP与rs2816316处于高LD(r^2>0.8)(Fig 3),有三个SNP(rs2816305, rs2984920和rs7535818)是转录因子结合位点,并且也与DHS和enhancer区域重叠。
Fig 3. rs2816316在HaploReg的查询结果
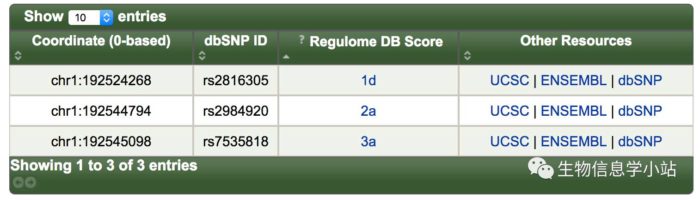
- 返回Regulome中继续查询上述三个SNP (Fig 4)。
Fig 4. rs2816305, rs2984920和rs7535818在Regulome的查询结果
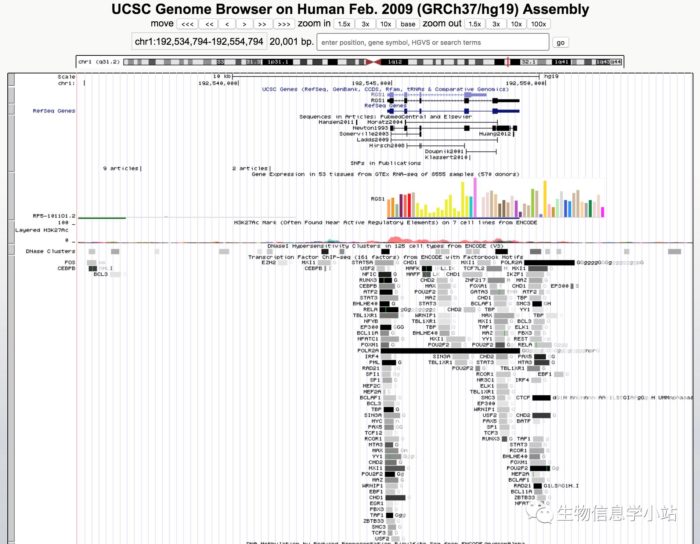
- rs2816305为RGS1的eQTL,同时处于调控元件内部。rs2984920在RGS1启动子内部,并且处于 PU.1和NFKB的motif内部(Fig 5)。这两个SNP都值得继续深入研究。
Fig 5. 在UCSC Browser查看rs2984920
4. 结语
这样的数据叠加,可以为该SNP的功能提供一些参考信息。如果我们能直接知道它调控的基因就更好了,当然有人早就想到这个问题,比如下篇要介绍的资源。。
参考资料
NCBI dbSNP Build 141: https://www.ncbi.nlm.nih.gov/projects/SNP/snp_summary.cgi?view+summary=view+summary&build_id=141
RegulomeDB: http://www.regulomedb.org/
HaploReg: http://archive.broadinstitute.org/mammals/haploreg/haploreg.php
ENCODE Document: https://www.encodeproject.org/documents/