Hi-C技术是高通量染色体构象捕获技术(High-throughput chromosome conformation capture)。
利用高通量测序技术,结合生物信息学分析方法,研究全基因组范围内整个染色质DNA在空间位置上的关系,构建染色体跨度单体型,同时捕获不同基因座位上之间的空间交互信息,获得高分辨率的染色质三维结构信息,并能开发调控基因的DNA元件。
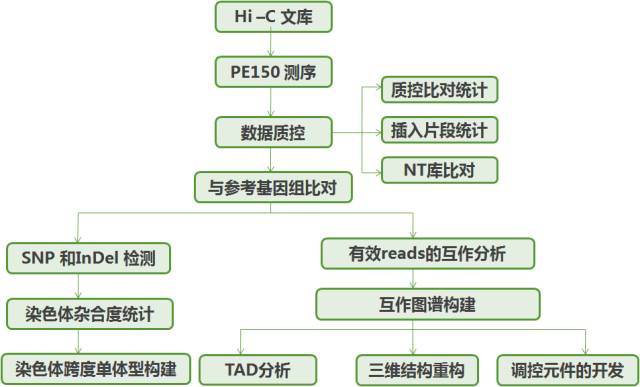
【Hi-C技术路线】
下面我们来结合文献看看Hi-C技术主要有哪些方面的应用~
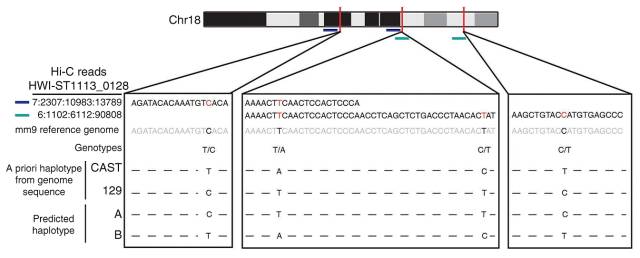
一、应用Hi-C技术构建染色体跨度单体型
基于Hi-C技术,将高通量测序与生物信息学技术结合起来,根据染色体边界的存在构建出了染色体跨度水平的单体型(Haplotype),与传统的单体型构建相比,利用Hi-C技术构建染色体跨度的单体型图谱,跨度更长,完整性更高。
图1 杂交小鼠胚胎干细胞染色体跨度单体型图谱[1]
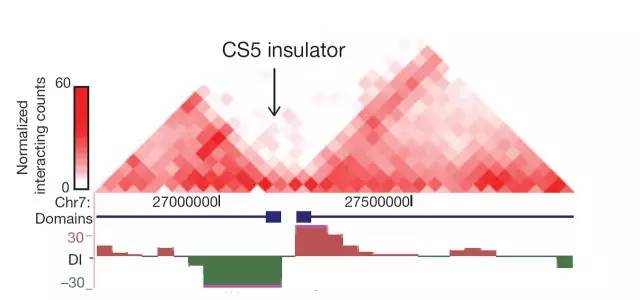
二、基于Hi-C技术进行TAD分析
TAD(topologically associating domains)是拓扑关联域,一般指具有“特殊生物学功能”的相互作用“方块”,一般区域内部的互作频率显著高于毗邻的两个区域的互作频率,是基因组在空间结构中基本的组织形式,在不同的细胞类型间和物种间存在较高的保守性。
图2 TAD结果图[2]
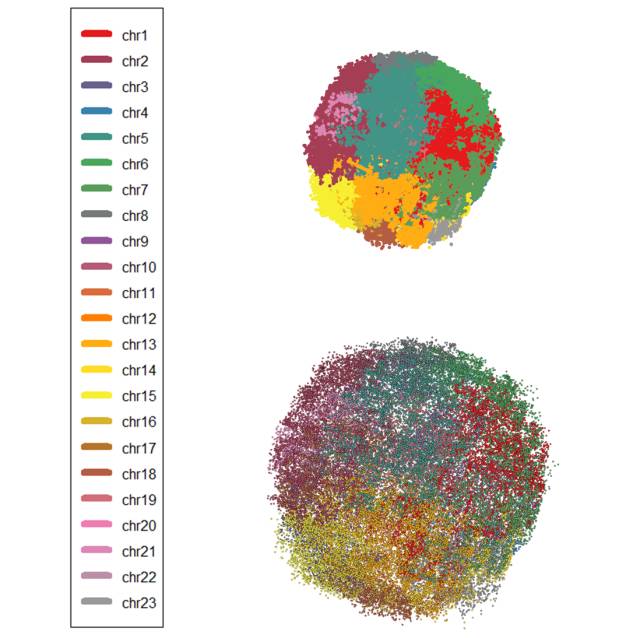
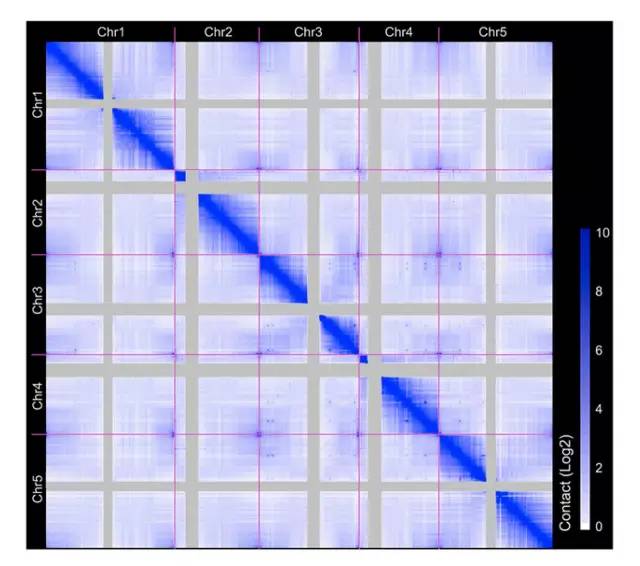
三、基于Hi-C技术进行三维结构重构
基于染色体全部的互作信息,利用一定的数学模型,将二维的染色体互作信息转化成三维空间结构的物理坐标,构建全基因组的三维空间结构,基因组范围内的三维空间结构反映染色体间的互作情况。
图3 基于Hi-C数据进行三维结构重构[3]
四、应用Hi-C技术开发调控元件
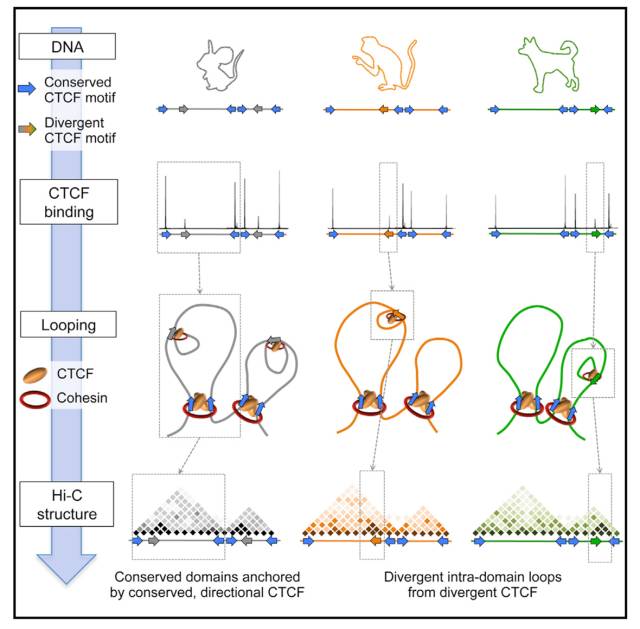
运用Hi-C技术的测序数据进行分析,发现CTCF在不同物种中既结合保守DNA序列,又因物种差异存在特异的DNA结合序列。而在不同物种中,由于其结合DNA序列不同,可能导致内部结构域发生结构变化,造成功能的差异,进而揭示了一系列局部结构的变化造成了生物的进化。
图4 CTCF在不同物种中的研究结果[4]
五、Hi-C技术揭示细胞不同时期染色体构型变化
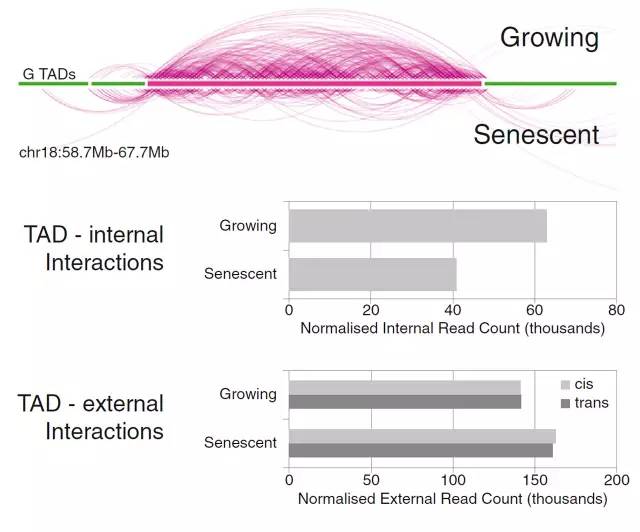
在对正常细胞和衰老细胞进行分别测序,在进行质控和TAD分析后发现,在衰老细胞中,TAD的内部互作减少,TAD间的互作加强(图5),研究者同时利用FISH实验验证了这一点,这表明,局部互作(TAD内部)强度的改变会改变衰老细胞的染色体构型。
图5 生长细胞与衰老细胞染色体构型比较[5]
六、Hi-C技术之植物Hi-C
通过Hi-C技术,研究者绘制了植物拟南芥精细的基因组三维结构Hi-C图谱。研究发现拟南芥的局部染色质包装不同于动物中所观察到的模式:大部分Kb级大小的片段中,染色质区域内部互作的比例比与邻近区域互作的比例高,这是拟南芥基因组构象中主要的局部结构特征。
图6 拟南芥基因组相互作用图(20kb分辨率)[6]
参考文献:
- Selvaraj S, R Dixon J, Bansal V, et al. Whole-genome haplotype reconstruction using proximity-ligation and shotgun sequencing. Nat Biotechnol. 2013, 31(12): 1111-8.
- Dixon JR, Selvaraj S, Yue F, et al. Topological domains in mammalian genomes identified by analysis of chromatin interactions. Nature. 2012, 485: 376-380.
- Trieu T, Cheng J. Large-scale reconstruction of 3D structures of human chromosomes from chromosomal contact data. Nucleic Acids Research. 2014, 42(7): e52-e52.
- Rudan MV, Barrington C, Henderson S, et al. Comparative Hi-C reveals that CTCF underlies evolution of chromosomal domain architecture. Cell Rep. 2015, 10(8): 1297-1309.
- Chandra T, Ewels PA, Schoenfelder S, et al. Global reorganization of the nuclear landscape in senescent cells. Cell Rep. 2015, 10(4): 471-83.
- Wang C, Liu C, Roqueiro D, et al. Genome-wide analysis of local chromatin packing in Arabidopsis thaliana. Genome Res. 2015, 25(2): 246-56.