60年来,生物学家一直只了解两条规范“DNA语言”的类似语法的规则。现在他们又发现四条
 奥地利生物化学家埃尔文·查戈夫(Erwin Chargaff)因于1950年发现了两条“DNA语言”语法规则而著名,这两条规则以他的姓氏命名。在当时,生物学界最大的问题就是理解DNA的结构。查戈夫发现的规则成了解决这个问题的重要线索。
奥地利生物化学家埃尔文·查戈夫(Erwin Chargaff)因于1950年发现了两条“DNA语言”语法规则而著名,这两条规则以他的姓氏命名。在当时,生物学界最大的问题就是理解DNA的结构。查戈夫发现的规则成了解决这个问题的重要线索。
生物学家早就知道,DNA是由四种分子组成的:腺嘌呤(adenine)、鸟嘌呤(guanine)、胸腺嘧啶(thymine)和胞嘧啶(cytosine)。但他们假定,这些分子在DNA组成里是等量出现的,并将暗示其他情况存在的任何测量结果都归为实验误差。
而查戈夫通过仔细测量证明,这种假定是错误的。他发现,腺嘌呤的数量与胸腺嘧啶相等,而鸟嘌呤的数量与胞嘧啶相等,但是,四种分子之间在数量上并不两两相等。查戈夫测量的大致比例为:腺嘌呤=胸腺嘧啶=30%,鸟嘌呤=胞嘧啶=20%。
如今人们所称的查戈夫第一等比规则,当时成了一条重要线索,詹姆斯·沃森(James Watson)和弗朗西斯·克里克(Francis Crick)在研究双螺旋结构的碱基配对模型时利用了这条线索。生物学家现在了解,腺嘌呤与胸腺嘧啶的结合以及鸟嘌呤与胞嘧啶的结合,共同形成了一股双螺旋,因此查戈夫规则适用于所有双链DNA。
查戈夫进一步研究发现,上述规则的近似描述也适用于多数(但不是所有)单链DNA。这是一个更大的难题,生物学家仍不十分清楚为什么会是这样。
查戈夫规则很重要,因为它们指明了一种“生物学语法”——一套规范DNA结构的潜在规则。这种语法应该是DNA的结构组成模式,在所有物种中都是一致的。
但是,自查戈夫发现这些普遍规则60年来,再没有人发现其他类似规则,直到现在。
现在,巴西应用生物信息学实验室(Applied Bioinformatics Laboratory)的米歇尔·山岸(Michel Yamagishi)和圣保罗金边大学(Unicamp)的罗伯特·赫尔瑞(Roberto Herai)说,他们发现了若干新模式,大大扩展了DNA语法规则。
他们的方法简单明确,就是利用数学中的集合论证明,查戈夫的已有规则暗示还有其他更高级的模式存在。
他们是这样证明的。研究DNA结构组成的一种方法就是,将DNA序列分割成一定长度的片段,长度记为k。查戈夫规则适用于k=1的片段,即适用于单个核苷酸。
但是,如果k=2(比如AA, AC, AG, AT等等)或k=3(比如AAA, AAG, AAC, AAT等等),情况会如何呢?生物化学家称这类片段为寡核苷酸。集合论表明,这些长度为k的片段的全集,必然也遵守特定的类似分形模式。
山岸和赫尔瑞将它们提炼为四个等式。
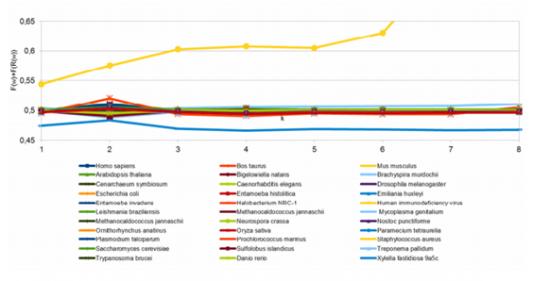
当然,只有在大型DNA数据库中才可能观察到这类模式。山岸和赫尔瑞对32个物种的DNA序列数据进行了反复数学运算,以寻找这类新分形模式。果然,他们找到了。
他们说,所研究的32个物种中,有30个物种相当精准地出现了这些模式,包括人类、大肠杆菌(e.coli)和植物拟南芥(arabidopsis)。只有人类免疫缺陷病毒(即艾滋病毒,HIV)和侵染桃树的苛养木杆菌(Xylella fastidiosa)9a5c的DNA与这类模式不一致。
他们说:“这些新规则首次证明,寡核苷酸的出现频率在大量不同基因组中具有一致性。”
该结论可能会极有价值,可以用于评估高速全基因组测序新技术的性能。
这类测序技术的一个问题就是,它们的测序精确度如何?山岸和赫尔瑞建议,进行一个简单的实验,测试一下新测序出来的基因组序列是否含有这类一致模式。如果没有,就说明该技术可能在测序过程中产生了某类偏差。
这有点像在大量数据中寻找随机误差的“校验和”实验,像一门简洁精巧的科学。
参考来源:生物探索 与 麻省理工科技创新


