GATK 是 Genome Analysis ToolKit 的缩写,是一款从高通量测序数据中分析变异信息的软件,是目前最主流的snp calling 软件之一。GATK 设计之初是用于分析人类的全外显子和全基因组数据,随着不断发展,现在也可以用于其他的物种,还支持CNV和SV变异信息的检测。在官网上,提供了完整的分析流程,叫做 目前最新版本文为安装过程如下:解压缩之后,可以看到两个后缀为这个命令能够打印出所有的子命令,如果打印出来结果,说明程序安装正确。部分子命令截图如下
目前最新版本文为安装过程如下:解压缩之后,可以看到两个后缀为这个命令能够打印出所有的子命令,如果打印出来结果,说明程序安装正确。部分子命令截图如下 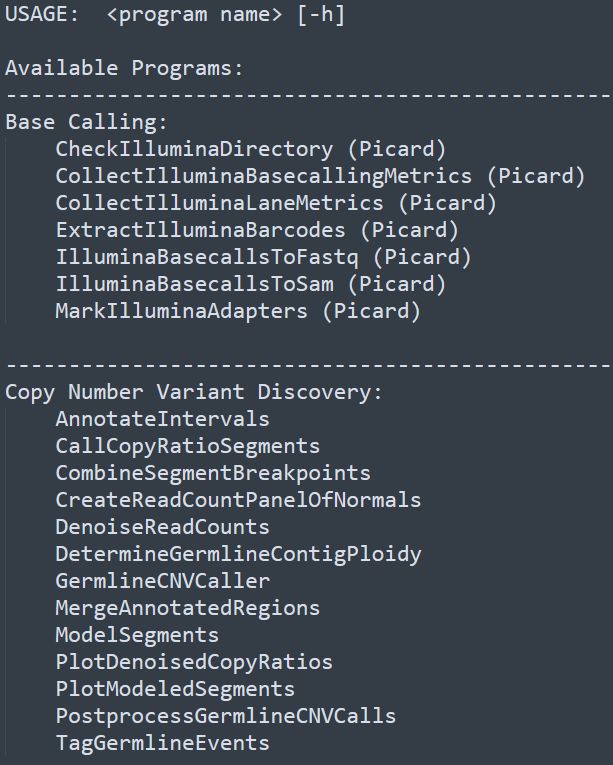 子命令后面如果有
子命令后面如果有
GATK Best Practices。  目前最新版本文为
目前最新版本文为4.0.4.0, 叫做GATK4。 和之前的版本相比,GATK4在算法上进行了优化,运行速率有所提高,而且整合了picard 软件的功能。GATK4基于java 语言开发的,需要java 1.8 版本。下载链接如下
- https://software.broadinstitute.org/gatk/download/
- wget https://github.com/broadinstitute/gatk/releases/download/4.0.4.0/gatk-4.0.4.0.zip
- unzip gatk-4.0.4.0.zip
- tree -L 1 gatk-4.0.4.0/
- gatk-4.0.4.0/
- ├── gatk
- ├── gatk-completion.sh
- ├── gatkcondaenv.yml
- ├── GATKConfig.EXAMPLE.properties
- ├── gatkdoc
- ├── gatk-package-4.0.4.0-local.jar
- ├── gatk-package-4.0.4.0-spark.jar
- ├── gatkPythonPackageArchive.zip
- └── README.md
.jar的文件,local用于本地运行,spark用于在spark集群上运行。实际使用时,直接用gatk这个可执行文件就行了。 通过一个简单的命令,查看程序是否正确安装
- gatk —list
 子命令后面如果有
子命令后面如果有(picard), 说明这个功能是继承于picard软件,从这里也可以看出,GATK4集成了picard软件的功能。再不需要像之前版本一样,混合使用picard 和 gatk 了。 GATK4 的最佳实践给出了5套pipeline- Germline SNPs + Indels
- Somatic SNVs + Indels
- RNAseq SNPs + Indels
- Germline CNVs
- Somatic CNVs
Germline和Somatic的区别。 Germline指的是在胚胎发育早起出现的变异,这种变异会在所有细胞中广泛存在,是可以遗传给后代的变异;Somatic指的是体细胞变异,身体特定区域或者组织中出现的变异。通常不会遗传给后代。 在所有的pipeline之前,都存在一个数据预处理步骤data pre-processing。
GATK4 版本的最佳实践并不是直接给出了每个步骤对应的代码,而是给出了几套它们自己编写的流程,以供参考。这些流程以WDL这种workflow 语言进行编写。官方对于WDL, 也给出了详细的文档,帮助我们了解。总结
- GATK4整合了picard软件,在算法上进行了优化,新增了许多新的功能。
- 官网给出了基于GATK4的pipeline, 以
WDL这种workflow 流程管理语言编写。



1F
变异检查的原理是什么?