Assay for Transposase-Accessible Chromatin with highthroughput sequencing(ATAC-Seq)即利用转座酶探究可接近性染色质高通量测序技术。通俗来说就是利用转座酶来获取开放性染色质,再通过高通量测序及生物信息学分析来挖掘相关基因信息,以此探究生物学相关问题。
Q:为什么研究染色质开放区域?
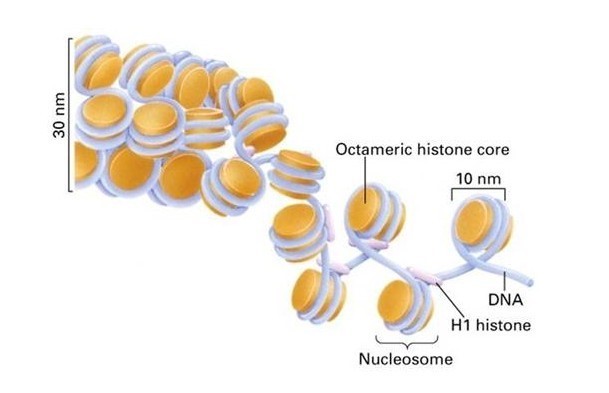
A:染色质分为常染色质和异染色质,在结构上常染色质折叠压缩程度低,处于伸展状态,DNA复制,基因转录都发生在DNA的致密高级结构变为松散的状态;这部分打开的染色质,就叫开放染色质(open chromatin)。而打开的染色质,就有足够的区域允许一些调控蛋白(比如转录因子和辅因子)过来与之相结合。而染色质的这种特性,就叫做染色质的可接近性(chromatin accessibility)。通过研究细胞特定状态下开放的染色质区域可以在DNA水平上了解其转录调控。
Q:如何寻找开放的染色质区域?
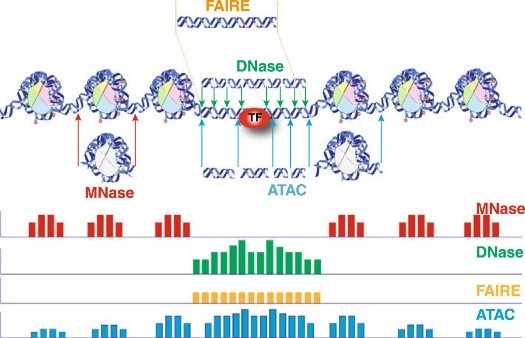
A:传统使用的的实验方法主要是有MNase-seq和DNase-seq ,这两种实验方法的主要思路是:染色质变得开放,就意味着DNA和组蛋白的聚集程度降低,就会有一部分DNA暴露出来。而一旦失去了蛋白质的保护,这部分DNA就可以被DNA酶(MNase或DNase I)所切割。然后,我们再把切割完的DNA拿来测序,和已知的全基因组序列相比较,就能发现被切割的是哪些序列,没有被切掉的基因序列又在哪里,就知道开放的染色质区域在哪里了。不过,这两个方法有明显的缺陷,即耗时费力与重复性差。虽然FAIRE-seq 不依赖酶和抗体,但其检测背景较高,测序信噪比低,甲醛交联时间不好把握等缺陷,限制其使用范围。
Q:有什么新技术方法来研究开放染色质?
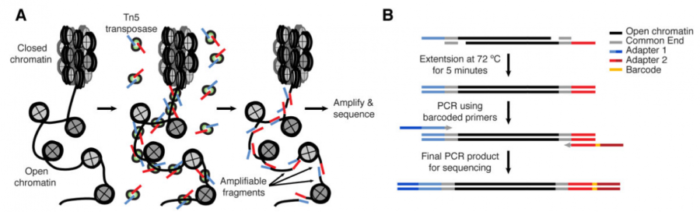
A:新推出的ATAC-seq利用Tn5转座酶(DNA转座,是一种把DNA序列从染色体的一个区域搬运到另外一个区域的现象,这一过程就由转座酶参与完成。Tn5转座酶:“标签片段化工具”,Tn5转座体可将其衔接子负载整合到可接近的染色质区域,而空间位阻较不可接近的染色质使得转座不可能发生。)人为将将携带已知DNA序列标签的转座复合物,加入到细胞核中,再利用已知序列的标签进行PCR建库测序,就知道哪些区域是开放染色质了。ATAC-seq出来的结果,和传统方法出来的结果具有很强的一致性,同时也和ChIP-seq有较高的吻合程度。而相比较而言,ATAC-seq的重复性,比MNase-seq和DNase-seq的更强,操作起来也更加简便,而且只需要很少的细胞/组织量,同时测序信号更加好。目前已经成为研究染色质开放性首选的技术方法。
Q:ATAC-seq主要优势是什么?
A:面对现实需求ATAC-seq有以下四个方面的强大优势:
- 灵敏性高:低细胞起始量(500-50000个);
- 操作简单,耗时短;
- 实验重复性好:技术重复间表现出非常好的可重复性( R = 0.98) ,并与DHs 测序数据间也有着较好的一致性(R>0.79);
- 能同时揭示开放染色质的基因组位置,DNA结合蛋白,转录结合位点的相互作用
Q:ATAC-seq能用在那哪些方面?
A:结合ATAC-seq优势以及研究热潮ATAC-seq有以下用途;当然进一步的开发也可能在不久的将来让其使用更加广泛。
1. 非生物逆境,病虫害,营养,激素等处理前后及动物疾病,转录活性差异。
2. 不同组织,器官转录活性差异,找到组织特异基因和启动子。
3. 利用ATAC-seq技术来研究A、B、D三个亚基因组的转录因子结合位点的差异,从而研究同源基因的表达调控差异。而六倍体与二倍体、四倍体等位基因间调控位点的比较。
4 通过ATAC-seq定义的open chromatin区域 ,再结合motif 分析,识别哪种转录因子参与了基因表达调控(对于抗体质量不好的TF,尤其有效)
5 而将ATAC-seq和RNA-seq进行整合研究,将会获得对生物体(动物或植物)中的转录调控机制;宏观分析细胞在该特定时空下整个基因组的调控网络。
Q:ATAC-seq 有哪些缺陷呢?
A:任何技术都有其限制因素,ATAC-seq也不例外;
1. Tn5通过插入剪断DNA 并将测序接头连接到剪断的两个DNA 片段的末端,因此对于一个DNA 片段而言,其两端的接头连接是随机的,这便导致同一片段两端的接头有50%的概率是同一接头。而只有连接不同接头的片段才可用于富集扩增及测序,因此,有一半的片段无法利用;
2. 大量剪断的DNA 由于片段过大,无法进行PCR富集;
3. Tn5 的活性受反应溶液的组成及反应条件影响,仍然需要优化以便提高剪切效果;
4. ATAC-seq在植物细胞中存在以下难点:细胞壁的存在,叶绿体、线粒体等细胞器的污染,缺少稳定遗传的细胞系;
Q:现在用ATAC-seq发表文章多么?
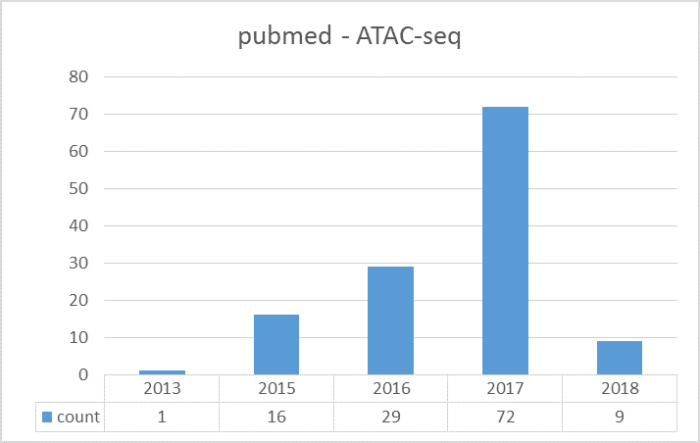
A:ATAC-seq在2013年问世,文章数量逐年增加,2018年刚开年就已经有近10篇文章已经发表;把握机会让您的研究如虎添翼。
Q:ATAC-seq文章推荐
A:ATAC-seq文章数量不断增长,希望下一篇就是您的杰作。
[1] Ackermann, A. M., Wang, Z., Schug, J., Naji, A., & Kaestner, K. H. (2016). Integration of ATAC-seq and RNA-seq identifies human alpha cell and beta cell signature genes. Mol Metab, 5(3), 233-244. doi: 10.1016/j.molmet.2016.01.002
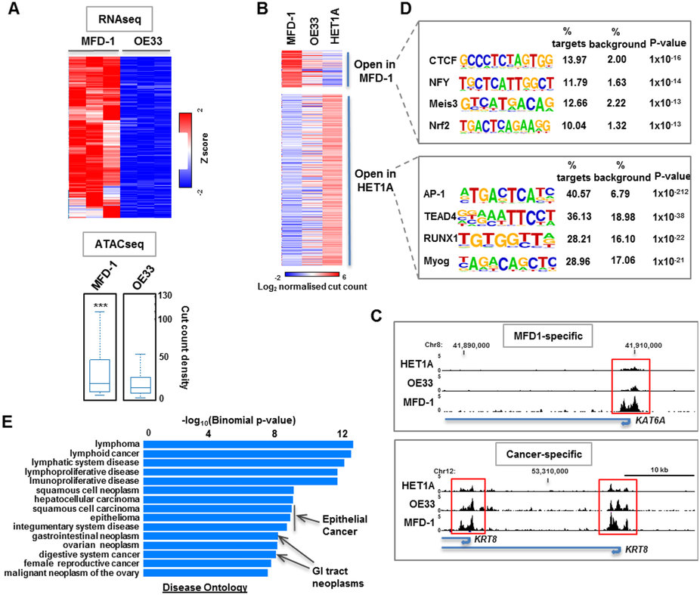
从55岁的男性病人中获取了一种新的食管癌细胞系:MFD-1,采用ATAC-seq方法与经典的食道癌细胞系OE33和人食管上皮细胞HET1A比较差异,进行MFD-1癌细胞特性研究,绘制MFD-1染色质开放位点图谱。和正常上皮细胞相比,MFD-1特异的染色质开放位点显著富集着CTCF、NFY、Meis3 、Nrf2 的motif,且这些位点附近基因富集在和上皮癌及消化道肿瘤相关的功能分组内。
结合全基因组测序(WGS)和表达谱测序(RNA-seq)技术,发现细胞系中4 个基因(ABCB1, DOCK2, SEMA5A 和TP53)存在5个突变位点,提供了MFD-1的基因表达特性。说明MFD-1能作为一种代表性的临床前原发性食管癌模式细胞系,可用于对应突变类型的研究。
[2] Tan, L., Ke, Z., Tombline, G., Macoretta, N., Hayes, K., Tian, X. Gorbunova, V. (2017). Naked Mole Rat Cells Have a Stable Epigenome that Resists iPSC Reprogramming. Stem Cell Reports, 9(5), 1721-1734. doi: 10.1016/j.stemcr.2017.10.001
成年裸鼹鼠skin fibroblasts (NSF)的iPSC诱导率显著低于小鼠mouse skin fibroblasts (MSF)。筛选了12个能提高重编程效率的因子,发现其中的LT(一种能够抑制Rb基因表达的东西)能提高裸鼹鼠的iPSC诱导效率。
此研究首先用组蛋白修饰ChIP-qPCR和组蛋白质谱对比了裸鼹鼠和小鼠组蛋白修饰的差异,又用ATAC-seq查看了开放区的差异。ATAC-seq结果跟前面复杂的组蛋白转录后修饰的结果非常符合ATAC-seq分析结果。
ATAC-seq实验表明:裸鼹鼠中重编程基因的promoter区跟小鼠比起来处于一种都关起来了(more closed)的状态,表达LT之后(LT可以抑制Rb, 提高重编程效率),发现裸鼹鼠这些基因的promoter区又大量开放了(massive opening)。
结论:裸鼹鼠具有更加稳定的表观基因组,这一特征帮助它抵抗脱分化,从而为抗癌和长寿做出了贡献。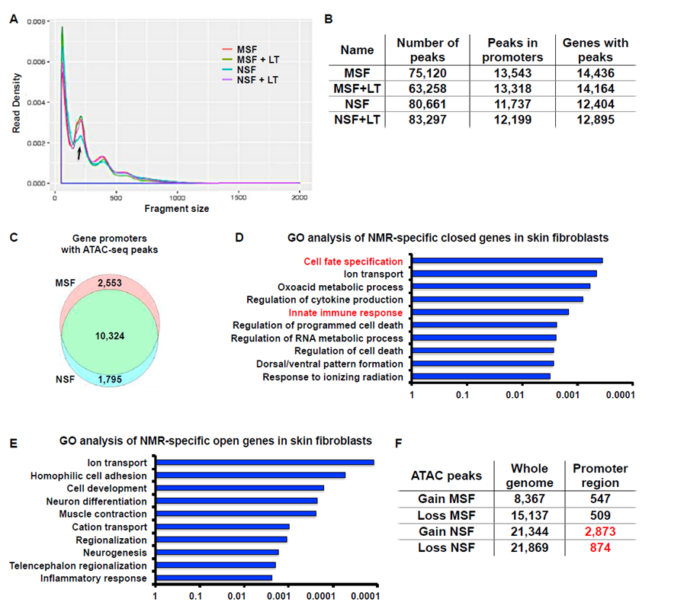
Q:ATAC-seq送样标准?
A:ATAC-seq因其限制性因素较少样品准备也变得简单,以下是其送样标准:
| 样品准备 | ||||
| 样品类型 | 样品量 | 样品处理 | 运输方式 | 备注 |
| 细胞系 | >5×104个 | 取样后直接速冻 | 干冰寄送(3d内) | 细胞是完整的且为均匀的单细胞悬液; |
| 动物组织 | ≥1g | |||
| 植物组织 | ≥1g | |||
| 真核微生物 | 需和老师协商 | |||
1. 样本要求
1)新鲜细胞
细胞培养于培养瓶中,在运输前灌满培养基,室温运输。
具体要求为:细胞数量>20 万,细胞状态良好无污染。
2)冻存细胞
培养细胞进行计数,按照5*104个细胞每管进行分装,分装好的细胞 4℃、500 G 离心 5 min;PBS洗涤,4℃、500G离心5min。小心吸去全部液体,将细胞沉淀用液氮进行速冻,干冰运输。
具体要求:细胞严格按照 5*104每管分装,每个样品分装三管。
3)动物组织
新鲜的组织取到1.5ml EP管中,用液氮进行速冻,干冰运输。 动物组织质量应当>1g,并且组织新鲜无降解,无结缔组织或血块存在。
4)植物组织
选取新鲜的植物组织,用液氮进行速冻,干冰运输。 尽量选取新鲜的嫩叶或者花等作为实验样品,样品量尽可能多;样品应保证新鲜。
2. 样品包装
所有的容器应当用parafilm进行封口
注意:
生物学重复:2-4次
测序深度:100 M以上,保证50 M 有效reads
样本要求:ATAC-seq实验对于细胞数量和质量有着严格的要求,实验的成败很大程度上细胞的状态以及细胞量与Tn5转座酶的比例。若细胞状态差,污染严重,那么测序的结果会出现很大的偏差。若果细胞数量过少,过多的酶会进行过度的切割,导致测序结果异常;而细胞量过多可能导致切割不充分,使得原本应当测到的开放区域未被测出。
Q:ATAC-seq-数据交付及周期
A:ATAC-seq数据质控标准:
①采用PE50测序策略数据量满足要求clean data≥50M clean reads(特别注意研究转录因子测序数据量需要≥200M);
②Q20 ≥ 80% ;
交付周期:45个工作日(样本数≤20个);大项目周期需评估沟通。