在bismark中提供了两种生成html报告的方式,对应两个命令
- bismark2report
- bismark2summary
第一个命令针对单个样本。每个样本在比对之后,都会生成一个report 文件。bismark2report 命令读取这个reort 文件,然后生成单个样本对应的html报告。
用法如下
- bismark2report —dir test4 —
- alignment_report test/test_data_bismark_bt2_SE_report.txt
--dir指定结果的输出目录,--alignment_report 指定比对产生的report文件的路径
最终会在输出目录生成一个html文件,包含3个方面的统计信息
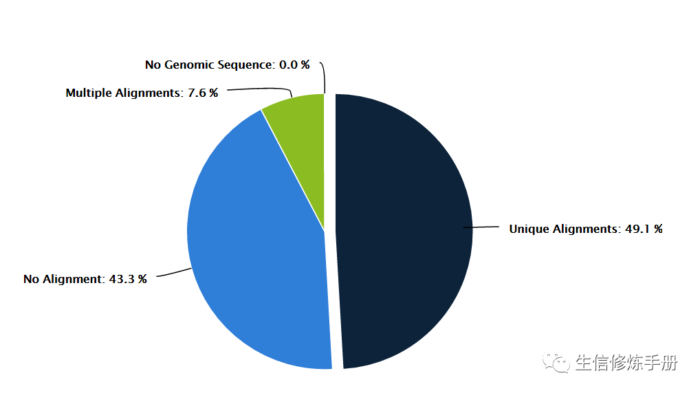
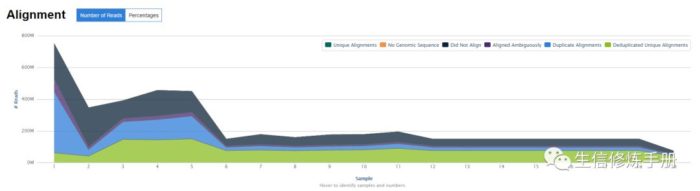
1. Alignment
在表格中,会给出总的序列数,没有比对上的序列数,唯一比对是序列数和比对到基因组多个位置的序列数。
采用扇形图来展示比对比例的分布情况
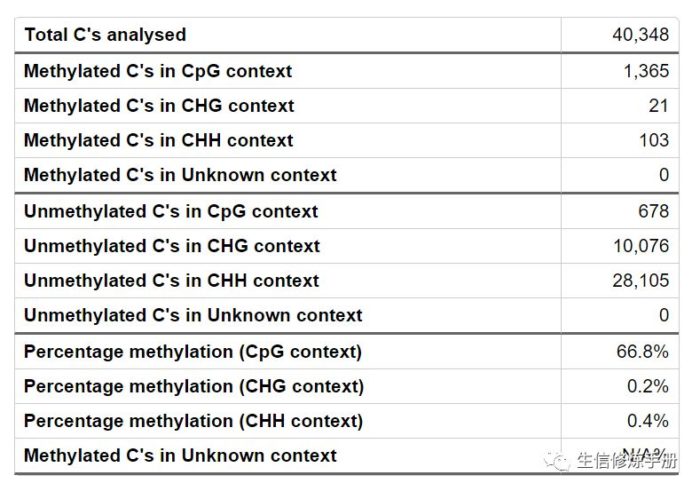
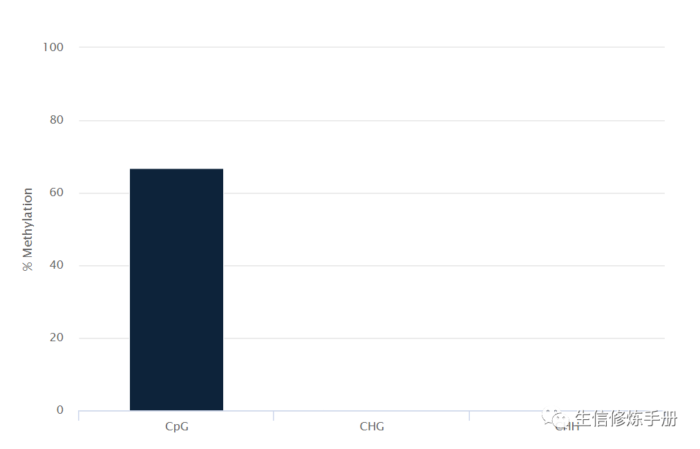
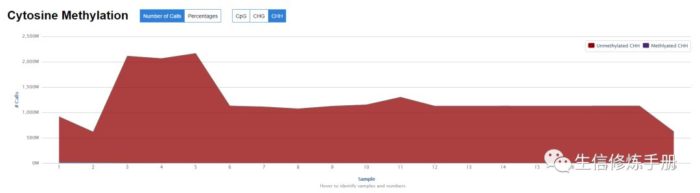
2. Cytosine Methylation
甲基化位点的汇总信息,包括CpG, CHG, CHH 下的甲基化和非甲基化C的数目和比例
采用柱状图的方式展示甲基化的C在CpG, CHG, CHH3种不同区域的分布比例
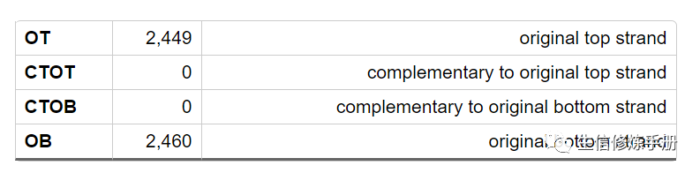
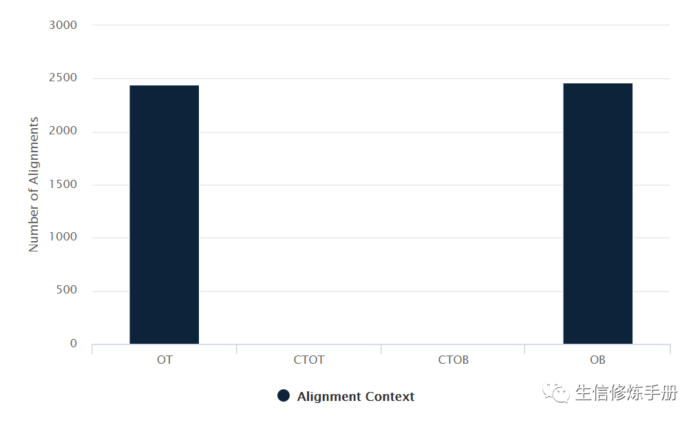
3. Alignment to Individual Bisulfite Strands
比对到OT, CTOT, CTOB, OB 4种链的reads 数
采用柱状图的形式展示比对到4种链上的reads 数目
第二个命令针对所有样本进行汇总,用法如下
- bismark2summary sampleA_bismark_bt2.bam sampleB_bismark_bt2.bam
生成的 html 文件包含以下两个方面的内容
1. Alignment
所有样本比对情况的汇总
2. Cytosine Methylation
所有样本甲基化C的个数,可以选择CpG, CHG, CHH,查看不同情况下的甲基化C的分布
除了html 格式的报告之外,还会生成.txt 格式的文件,每个样本是一行,包含的信息和html 中展示的信息是一样的。
作者:庐州月光
链接:
來源:简书
简书著作权归作者所有,任何形式的转载都请联系作者获得授权并注明出处。