最近有人问我关于ATAC-Seq与ChIP-Seq的区别。刚好在分析ATAC-Seq和DNase-Seq的数据,于是做了个简单介绍。在六六_ryx的博客里面看到下面的介绍,做了简单修改放在这里跟大家分享。
ATAC-Seq、DNase-Seq与ChIP-Seq的不同的是:
ATAC-Seq和DNase-Seq是用来鉴定全基因组范围内染色质的开放区域(open chromatin region),可以得到全基因组范围内的蛋白质可能结合区域信息,一般用于不知道特定的转录因子,用此方法与其他方法结合筛查感兴趣的特定调控因子。
ChIP-Seq是明确知道感兴趣的转录因子是什么,根据感兴趣的转录因子的抗体去富集转录因子结合的DNA区域。同时,该方法也用于鉴定组蛋白结合位置和组蛋白修饰的区域,例如:H3K4me3,H3K27ac等。
除了这三种实验方法外,还有其他多种鉴定染色体元件的实验方法。这些方法基本整体思路比较类型,都是对特定区域进行富集,然后根据不同实验原理开展不同的后续分析。下面是下面的这张图是从《Identifying and mitigating bias in next-generation sequencing methods for chromatin biology》这篇文章中截取的ATAC-Seq、ChIP-Seq、Dnase-Seq、MNase-Seq、FAIRE-Seq进行简单比较,希望对大家有所帮助。
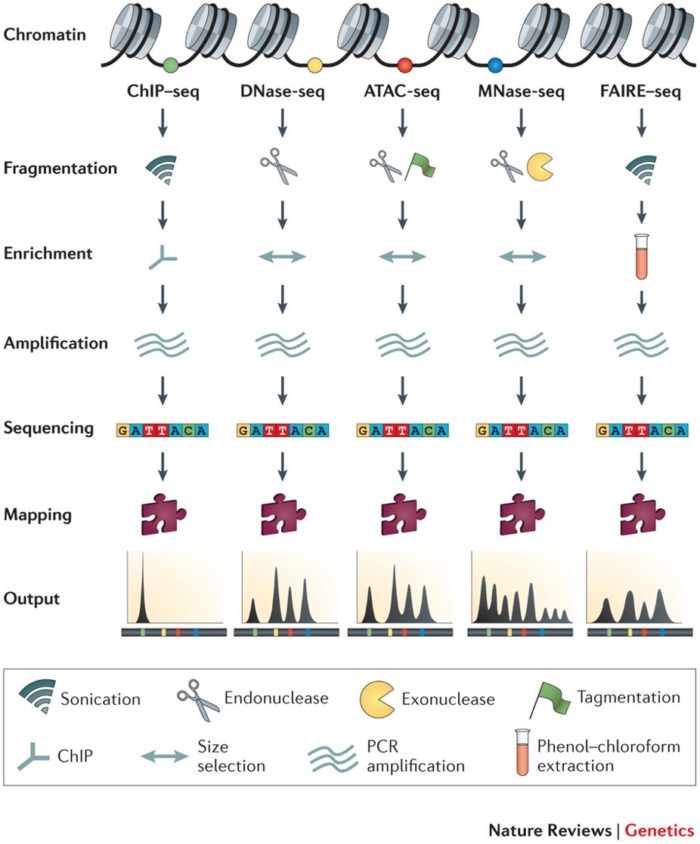
An overview of ChIP–seq, DNase-seq, ATAC-seq, MNase-seq and FAIRE–seq experiments
以上方法可以简单概括为:
- ChIP-Seq是揭示特定转录因子或蛋白复合物的结合区域,实际是研究DNA和蛋白质的相互作用,利用抗体将蛋白质和DNA一起富集,并对富集到的DNA进行测序。
- DNase-Seq、ATAC-Seq、FAIRE-Seq都是用来研究开放染色质区域。DNase-Seq是用的DNase I内切酶识别开放染色质区域,而ATAC-seq是用的Tn5转座酶,随后进行富集和扩增;FAIRE-Seq是先进行超声裂解,然后用酚-氯仿富集。
- MNase-Seq是用来鉴定核小体区域。
参考:


