在生信分析过程中,尤其是转录组分析中,经常会遇到测得数据不足,需要利用公共数据库中已有的数据,那么能将这些数据直接和测序的数据混合吗?如果贸然混合,会有什么问题?
10年nature有一篇综述,专门讲这个问题。
不同平台的数据,同一平台的不同时期的数据,同一个样品不同试剂的数据,以及同一个样品不同时间的数据等等都会产生一种batch effect 。这种影响如果广泛存在应该被足够重视,否则会导致整个实验和最终的结论失败。
我简单说下什么叫做batch effect。比对实验组和对照组,不同的处理是患病和不患病(测序时,先测得疾病,然后测得正常),然后你通过分析,得到很多差异表达的基因。现在问题来了,这个差异表达的结果是和你要研究的因素有关,还是时间有关,这个问题里时间就会成为干扰实验结果的因素,这个效应就是batch effect。
如何检测是否存在这种效应呢
最简单的就是记录实验中时间这个变量,然后对差异表达的基因进行聚类,看是否都和时间相关,如果相关就证明存在batch effect。
同样,如果不同平台的数据之间存在batch effect ,就不能简单的整合。
大家可能都会问标准化,会不会处理掉batch effect ?
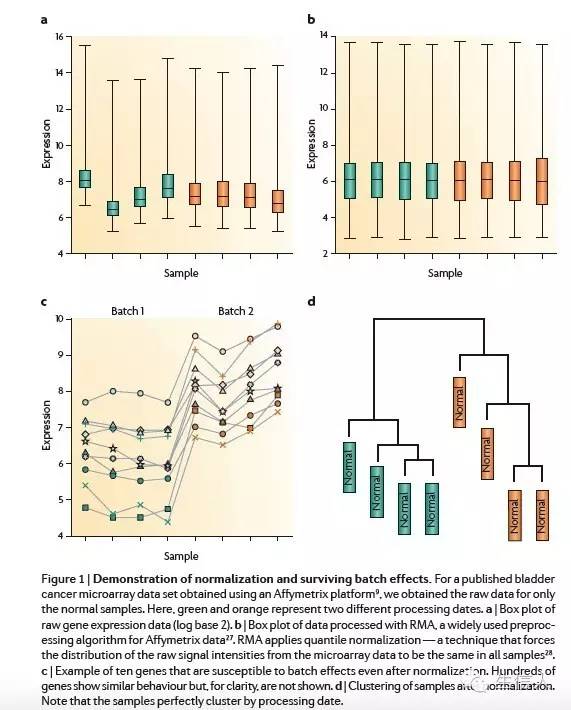
答案是能够减弱,不能从根本上消除。如下图,b是a进行过标准化的结果,从样本上看都一直,没有什么问题,但是落实到基因层面,c图中还是有明显的batch effect,d图中通过时间进行聚类,很明显可以看出差异表达主要是由于时间引起的。
通常情况下我们只考虑实验室情况和时间影响,并且只考虑线性关系,其实还是有一些其他的因素在影响,但是如果他并不能作为首要因素影响实验结果的时候,我们就可以忽略这些因素啦。
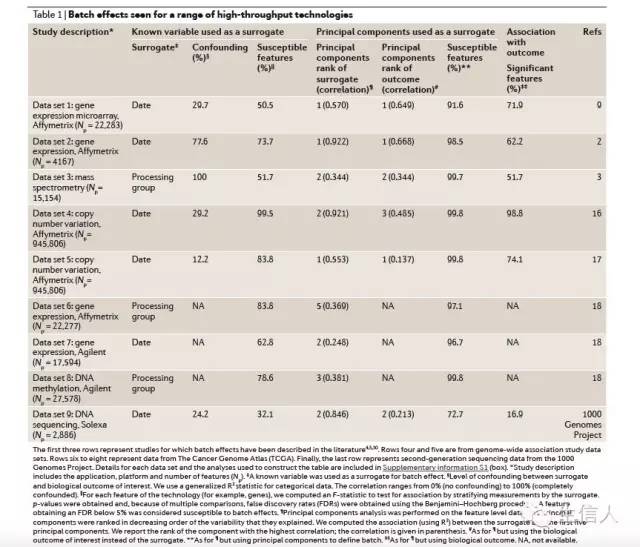
通过对公开数据的分析,可以很明显的看出往往混合数据都有很高的batch effect。
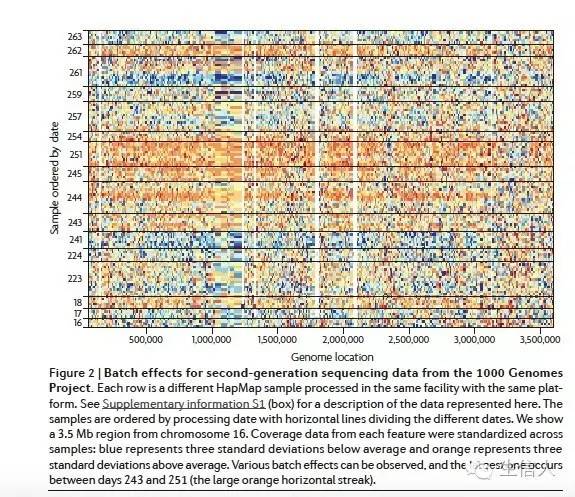
千人基因组计划中,按照时间聚类,可以看到很明显的蓝色条和黄色条,说明也存在明显的batch effect。
如何处理batch effect?
首先如果是自己设计实验,应该尽量分散掉这种不相关因素的影响,比如测正常和患病组织时不要集中的上午测正常,下午测患病,应该随机分散开,破坏掉时间效应,另外还有其他的因素,也应该进行分散。
文章中建议
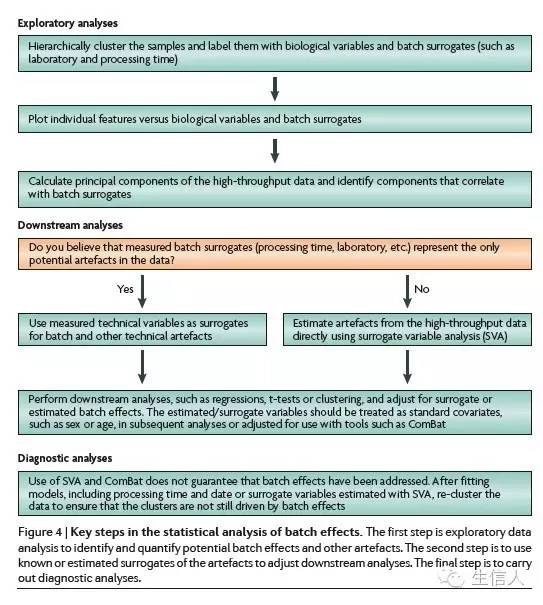
对样本加标签,然后通过聚类看是否存在某种效应,然后确认那种因素最为相关,然后利用统计模型进行过滤,然后再验证下,是否还有batch effect。
在线评估工具和参考资料
TCGA:http://bioinformatics.mdanderson.org/tcgabatcheffects
http://www.itl.nist.gov/div898/handbook/eda/section4/eda42a3.htm
http://www.biomedsearch.com/nih/Removing-batch-effects-in-analysis/21386892.html
http://www.molmine.com/magma/global_analysis/batch_effect.html
http://www.bioconductor.org/packages/



1F
可以把原文发给我吗,谢谢
B1
@ 一夕 https://www.ncbi.nlm.nih.gov/pubmed/20838408