seqlogo图可以直观清晰的反应序列偏好特征,每个位置出现的碱基或氨基酸类型反映了该位置序列的偏好性,每个字母的大小与该碱基在该位置上的出现频率成正相关。这种表现方式对研究转录因子结合、RNA修饰等有重要指导意义,时常被应用到论文中。前面介绍了关于如何利用R包绘制seqlogo图,本文接着推荐一个在线绘制seqlogo的工具Weblogo.
Weblogo在线工具
WebLogo(http://weblogo.threeplusone.com)绘制seqlogo的老牌在线工具。相比于在R上绘制seqlogo图,网页版在线工具更加轻松容易。但同时也存在一定的局限性,就是不适合分析大批量数据。
结合利用ggseqlogo绘制seqlogo图我们可以分别体验在网页绘制的轻松感,也可以体验本地运行代码的快感。下面就来看一下WebLogo的使用:
Weblogo的首页
用户指南
网站中给出了用户指南,里面详细介绍了上传数据要求的格式、logo大小及颜色调整、输出格式等。
在此列出需注意的几项,更多详情参见网页:
- Sequence Data:上传的文件格式要求为 CLUSTALW, FASTA, plain flatfile, MSF, NBRF, PIR, NEXUS和PHYLIP文件,其中要求每段序列的长度需保持一致
- Output format:最终结果可存储PNG、JPEG、EPS、PDF、SVG格式
- Sequence Type:序列类型可以是蛋白序列、DNA、RNA
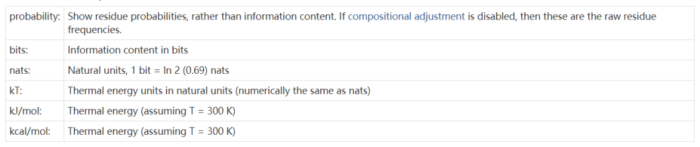
- Y轴的单位设定见下表
- logo字符颜色更改:支持更改既定的配色方案,更改颜色需使用指定的CSS2语法输入颜色
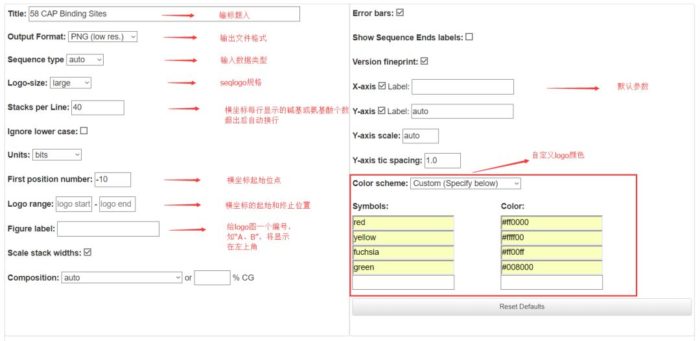
- 具体参数见下文图示
examples
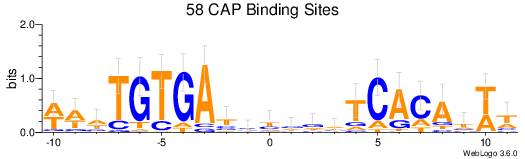
Examples界面下展示了多个经典蛋白序列、核酸序列保守性logo图,点击Edit Logo可以查看这些例子的数据输入格式及作图时标题、横纵坐标设定等参数,可以试着重新更改参数后看看效果。
Create——绘制自己的seqlogo
在Create界面选择上传数据文件或者直接输入序列,调好各项图片参数后即可得到logo图!注意上面提到的数据格式要求,每一段序列要求相同的长度。而在R包中,除了可以输入序列外还可以输入PFM矩阵进行分析。
核酸序列的输入格式
- >aldB -18->4
- attcgtgatagctgtcgtaaag
- >ansB 103->125
- ttttgttacctgcctctaactt
- >araB1 109->131
- aagtgtgacgccgtgcaaataa
- …...
蛋白序列的输入格式
- >Q9EXQ1/196-227
- LTMT.-RGDIGNYLGLTVETISRLLGRFQKLGVL
- >Q46158/72-92
- LTMT.-RGDIGNYLGLTVETISR-----------
- >Q46157/72-92
- LTMT.-RGDIGNYLGLTVETISR-----------
- >Q46159/72-92
- LTMT.-RGDIGNYLGLTVETISR-----------
- >Q47948/72-92
- LTMT.-RGDIGNYLGLTVETISR-----------
- >FNR_HAEIN/196-227
- LTMT.-RGDIGNYLGLTVETISRLLGRFQKLGVI
- >ETRA_SHEPU/193-224
- LTMT.-RGDIGNYLGLTVETISRLLGRFQKSGLI
- ……<
可以在下面Color scheme指定自定义配色方案,在左边指定颜色,右边指定相关符号,使用CSS2语法输入颜色。这样我们就给seqlogo图换了一身衣服~ (Version fineprint勾掉后就会隐去右下角的WebLogo水印,用完别忘记引用哦)