R包ggseqlogo 绘制seq logo图和Seq logo 在线绘制工具–Weblogo介绍了如何用R脚本和在线工具绘制seq logo图,用于展现转录因子或修饰酶等结合序列的偏好性。
JASPAR数据库 (http://jaspar.genereg.net/) 提供了转录因子与DNA结合位点motif最全面的公开数据,共收集了脊椎动物、植物、昆虫、线虫、真菌和尾索动物六大类不同类生物的数据。2018年更新发布的Jaspar中(https://academic.oup.com/nar/article/46/D1/D260/4621338#107184533),新增322种新物种的Position Frequency Matrix (PFMs),更新33个物种的PFMs。引入316个物种的TF flexible models (TFFMs), 对每个物种和物种内TF家族的结合PFM进行相似性聚类。基于这些收集的PFMs预测了人全基因组范围的TF结合位点,以UCSC track hub的形式供大家使用。
界面介绍
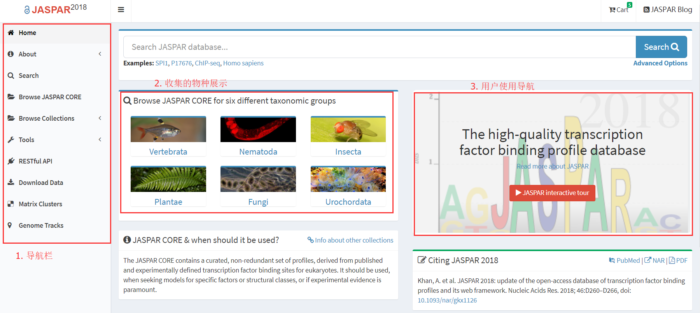
如下是Jaspar主页面,左边是工具栏;中间显示的是数据库中收录的六大类生物,可点击查看每个大类中收集的数据总量;右侧是用户使用导航,第一次使用的用户可以点击JASPAR interactive tour ,可跟随该导航一步步学习网站的使用方法。
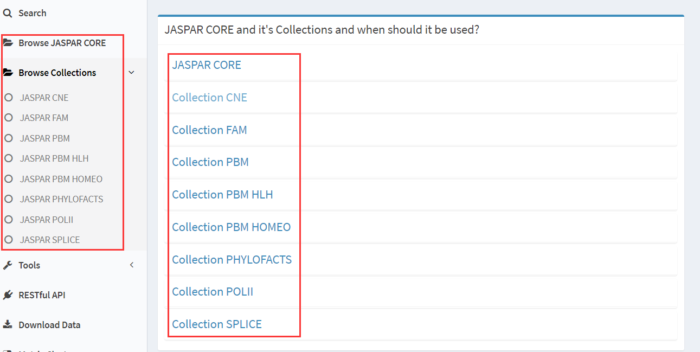
Jaspar中有9个子数据库,CORE, CNE, FAM, PBM, PBM HLH, PBM HOMEO等,关于什么时候使用哪个数据库,在About下有详细介绍。
| DB | Description |
|---|---|
| JASPAR CORE | Seeking models for specific factors or structural classes. |
| CNE | Characterizing of regulatory inputs in long-range developmental gene regulation in vertebrates. |
| FAM | Searching large genomic sequences with no prior knowledge. For classification of new user-supplied profiles. |
| PBM | Characterizing of regulatory inputs in long-range developmental gene regulation in vertebrates. All the PBM s are built by using new in-vitro techniques, based on k-mer microarrays. |
| PHYLOFACTS | Chraacterizing other factors might determine promoter characteristics, such as structural aspects and tissue specificity. |
| POLII | Investigating core promoters from multicellular eukaryotes. |
| SPLICE | Aanalyzing splice sites and alternative splicing. |
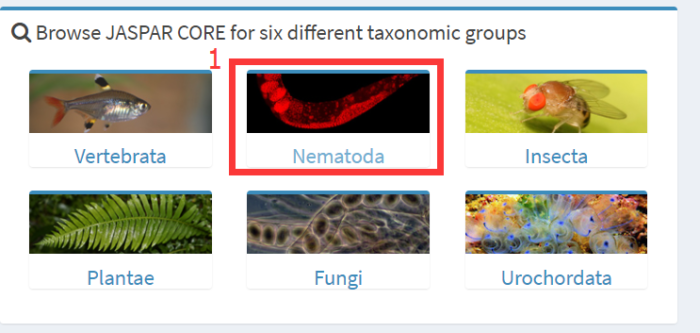
根据所研究的物种,选择对应的库,这里以Nematoda(线虫纲)为例,点击进入。
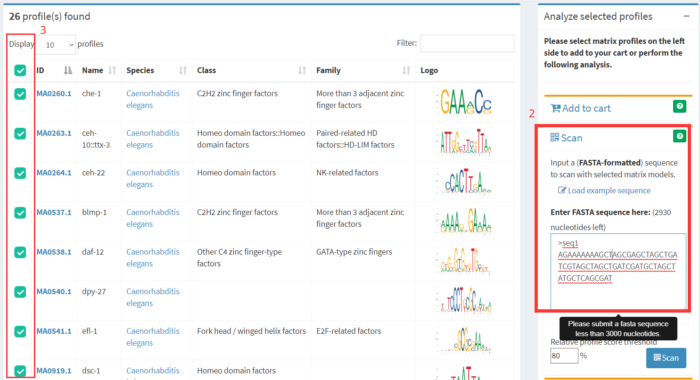
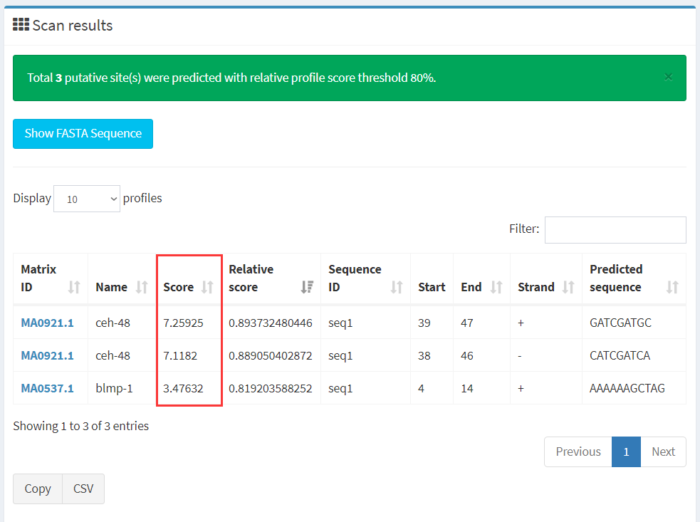
在Scan序列输入框中输入我们想要查找的启动子区域序列或增强子区域序列或其它关注的区域,注意需要输入FASTA格式。
在左侧列表中勾选待预测结合的转录因子,或者将同一物种的转录因子都勾选上,点击SCAN即出现结果展示。Score评分越高,表示该转录因子与输入序列结合的可能性越大。
基本搜索
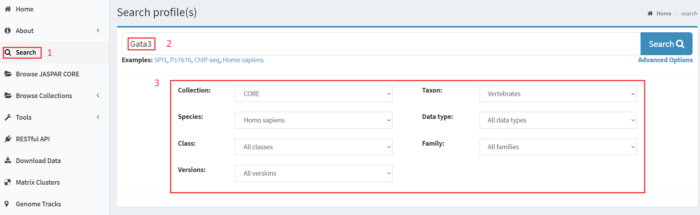
点击左侧工具栏中search,查找感兴趣的数据。可以通过TF名称或ID、物种、分类单元、uniprot ID或任何其他关键字进行搜索。我们以Gata3(GATA binding protein 3)为例,下方可选框中选择筛选要求,快速锁定目标数据。
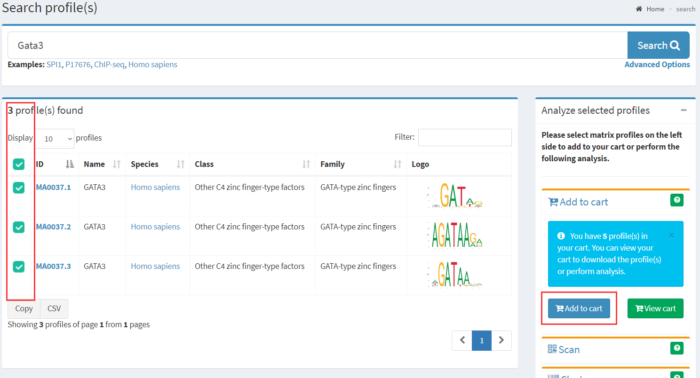
搜索结果被展示在一个列表中,可选择感兴趣的数据添加至右侧购物车中下载,然后自行分析。
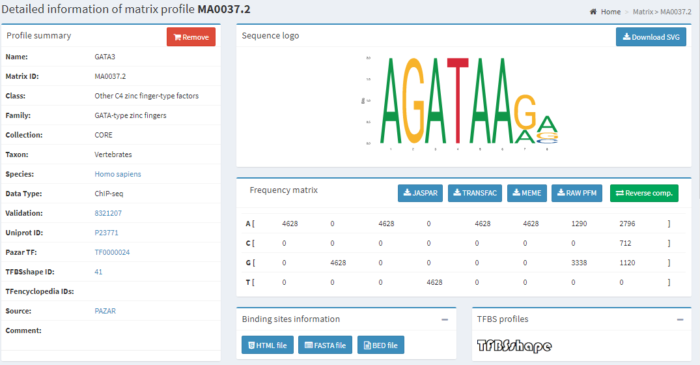
点击ID号,跳转至Gata3 motif详情,包括文件摘要、序列logo图、PFM矩阵(文件可下载)、TF-binding信息(FASTA格式为该motif在基因组范围的结合区域的序列,bed格式为该motif在基因组范围的结合位点,可点击下载)、外部链接、版本信息、ChIP-seq centrality、TFFM和其它信息。
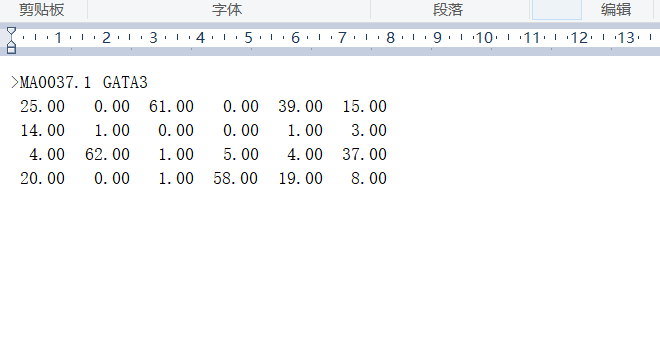
可在详情页Frequency matrix下载关于Gata3各类型矩阵信息,如下图。
jaspar tools 输入序列或矩阵搜索
- 点击
Profile Inference,可预测用户输入的蛋白序列的潜在结合motif。 - 点击
Matrix Align输入PFM矩阵,预测其相似性motif。常用于鉴定新发现的motif是否为某类转录因子的结合motif的变种。
蛋白序列的输入格式
MSDNDDIEVESDEEQPRFQSAADKRAHHNALERKRRDHIKDSFHSLRDSVPSLQGEKASRAQILDKATEYIQYMRRKNHTHQQDIDDLKRQNALLEQQVRALEKARSSAQLQTNYPSSDNSLYTNAKGSTISAFDGGSDSSSESEPEEPQSRKKLRMEAS
PFM矩阵的输入格式
A [13 13 3 1 54 1 1 1 0 3 2 5]C [13 39 5 53 0 1 50 1 0 37 0 17]G [17 2 37 0 0 52 3 0 53 8 37 12]T [11 0 9 0 0 0 0 52 1 6 15 20]
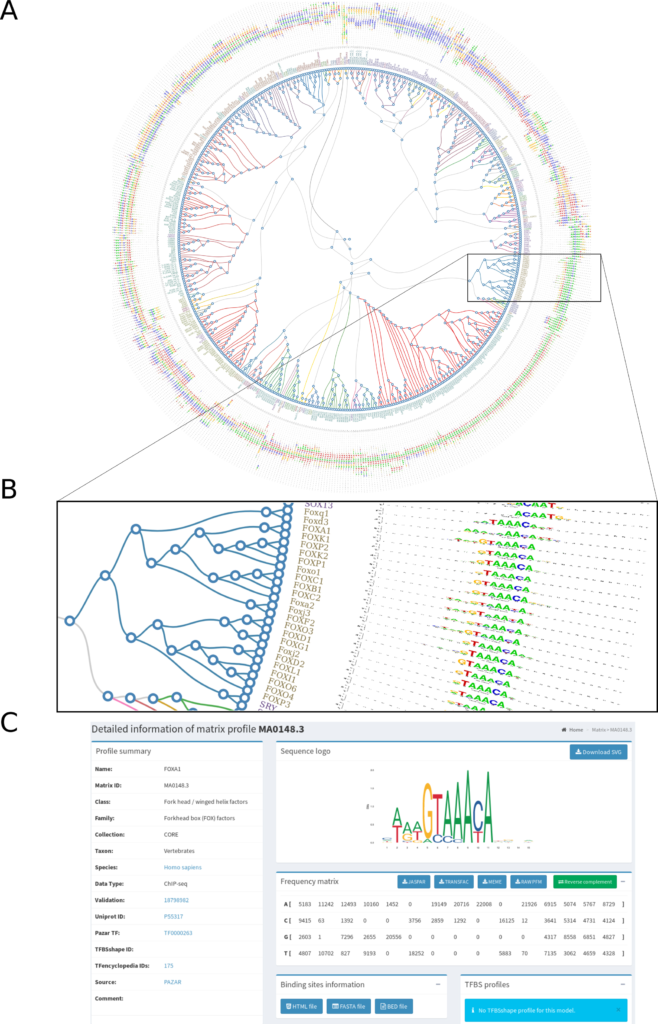
矩阵聚类工具
具有类似DNA-binding domains的TFs具有相似的序列偏好性,为了便于在Jaspar数据库中寻找相似的TFs,该数据库使用矩阵聚类工具对PFM进行了层级聚类分析。聚类结果以径向树的形式展示,可以点击每个logo图做进一步研究。