在做完16S、18S或ITS等微生物多样性研究后,我们常常还会想进一步了解微生物群落的功能。通常情况下,会采用宏基因组、宏转录组或宏代谢组等方法深入分析,但相对于扩增子测序,宏基因组等测序手段的价格还是相对较高,因此需要从已测完的样本中再挑选合适的样本进行宏基因组测序。那么,这个时候就遇到一个问题——我们该如何挑选样本呢?
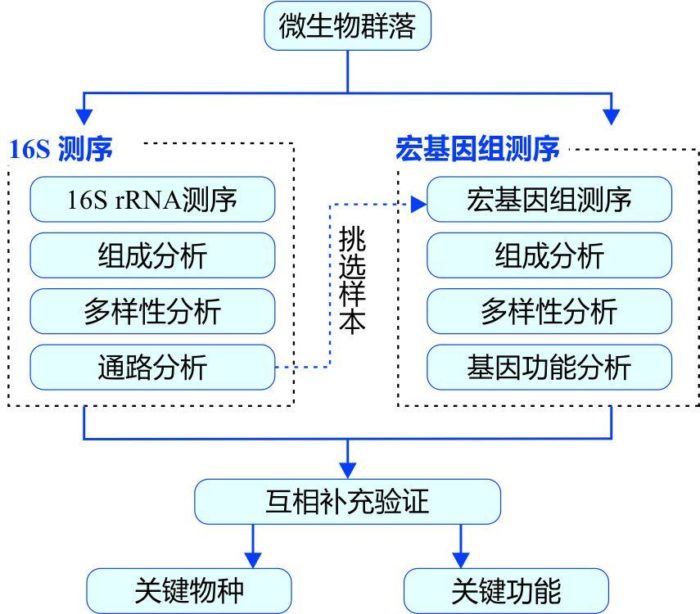
图1.16s+宏基因组关联分析思路
1.What——什么是microPITA
在人类微生物组项目(HMP)中,有5516个16s的样本,但其中只有682个样本进行了宏基因组测序,后续样本随机选择[1];在Yatsunenko等人的研究中,从532个16s样本中挑选110个样本进行宏基因组测序,没有明确的选择标准[2]。
而样本选择的方法,最好还是需要有一个可参考并且能提供给后人进行验证的方法,因此,microPITA软件应运而生[3]。microPITA(Microbiomes: Picking Interesting Taxa for Analysis),是由麻省理工学院的Tickle 等人研发,并且可以免费使用。
当我们的研究目标不同时,感兴趣的微生物群落特征也就随之发生改变。因此microPITA提供了4种无监督的方法和2种有监督的方法帮助大家进行样本挑选。
| 4种无监督方法 | 含义 | 样本特点 |
| diverse | 选择α多样性最高的样本 | 生态多样性高 |
| features | 根据目标物种挑选样本 | 针对特定物种 |
| extreme | 选择β多样性距离最远的样本 | 极端样本 |
| representative | 最能反映整体距离差异的样本 | 核心样本 |
| 2种有监督方法 | 含义 | 样本特点 |
| Distinct | 根据表型/分组特征, 挑选组间β多样性距离最大的样本 | 依据表型/分组特征,选择极端样本 |
| Discriminant | 根据表型/分组特征, 挑选离分组中心最近的样本 | 依据表型/分组特征,挑选核心样本 |
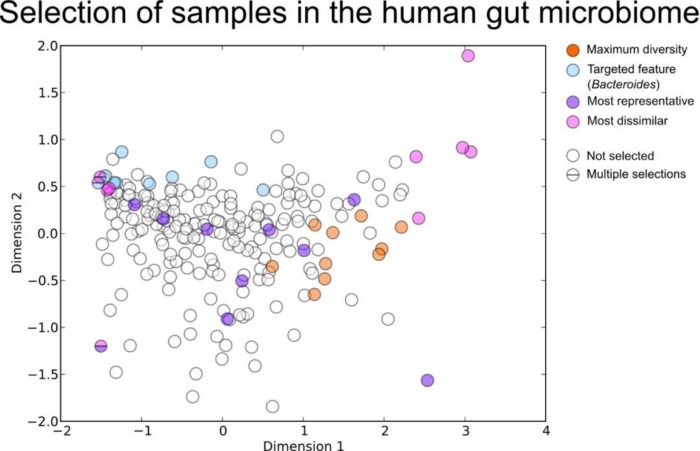
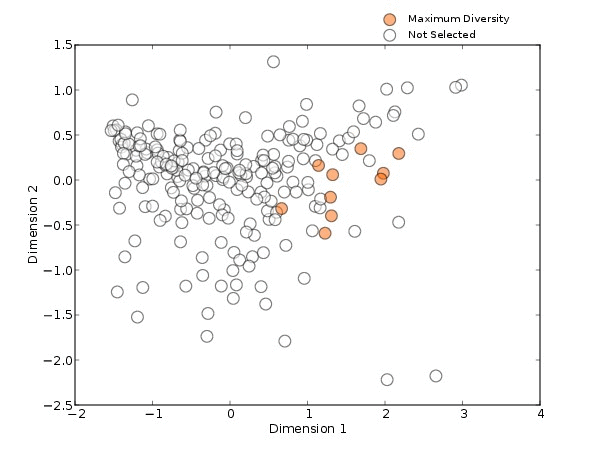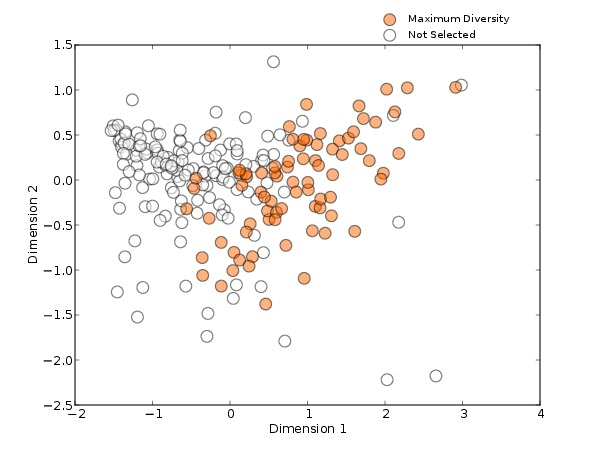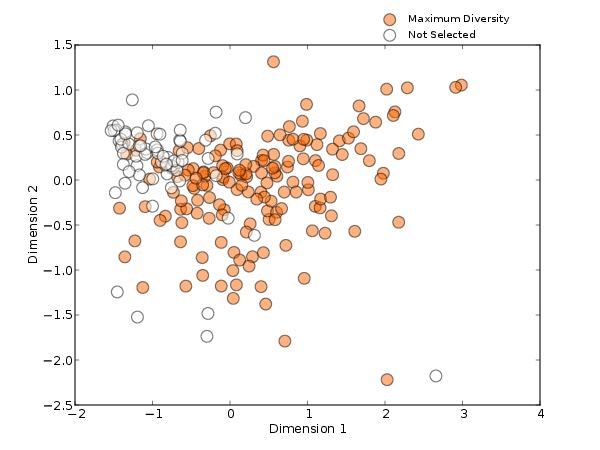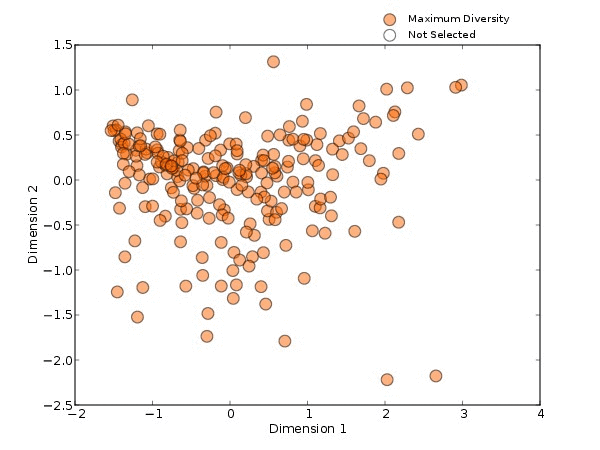
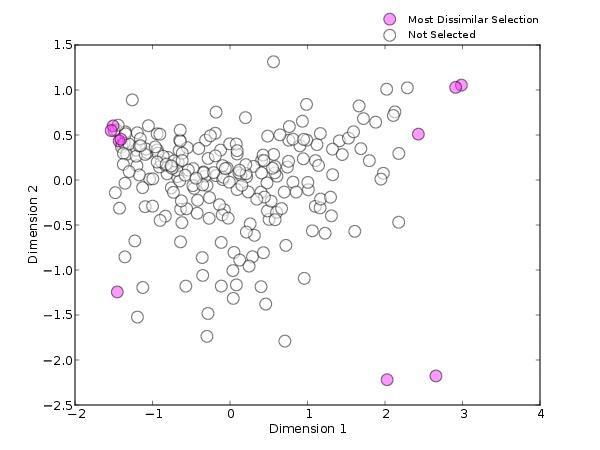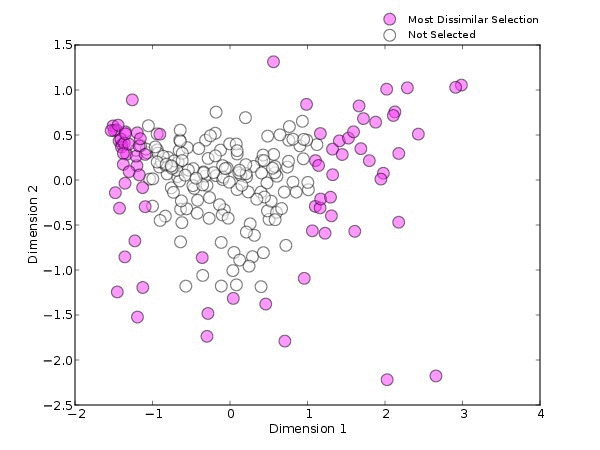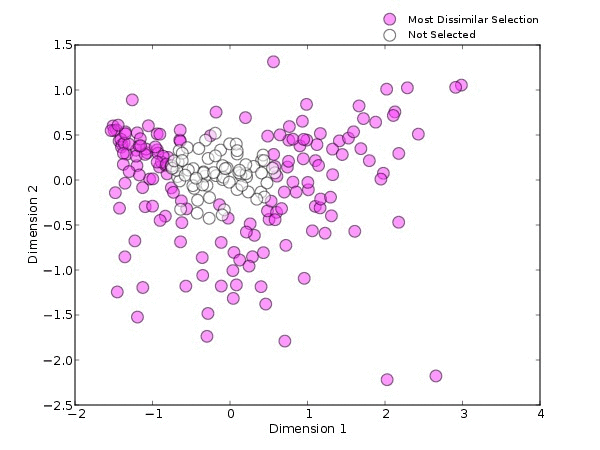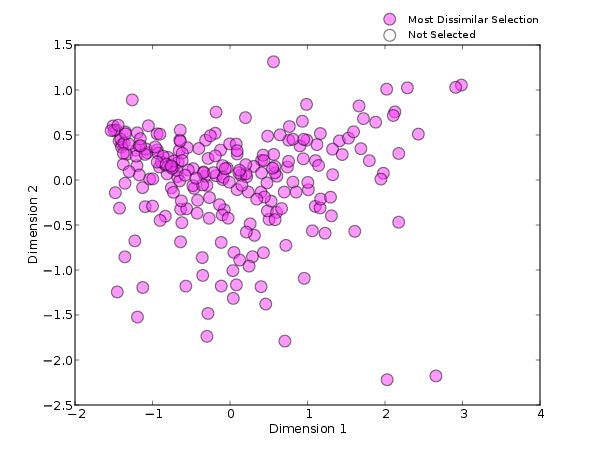
以HMP的16s肠道样本为例(图2),用不同的方法进行选择时,结果也会不同。分别使用目标物种为拟杆菌(蓝色)、最大多样性(橙色、代表性差异(紫色)和最不相似(粉红色)这四种方法筛选出10个样本。
可以看出,目标特征为拟杆菌为主的样本大多集中在PCOA中左上的位置;最大多样性则会选择更多样化的样本;代表性差异的方法会涵盖PCOA范围中大部分的样本,集中在密度较高的中心区域;而最不相似方法选择出的样本会在PCOA的外围。由此可见,当我们选择的方法不一样时,筛选出的样本也会随之发生改变。
图2.不同方法挑选的样本
2.How——microPITA如何保证结果的稳定性
当我们做了16s测序后,从中挑选出的样本需要保持与之前的特征一致,或者说更具有我们关注的特征。但有的时候,我们可能不太确定应该选择多少个样本用于后续的研究,并且选择出来的样品是否稳定,是否还依然保持着和16s结果一样的特征,如果是随机挑选的话,我们无法回答这些问题。
而microPITA能帮助我们解决这些问题。当采用不同的方法进行连续选择样品时(图3),目标特征为拟杆菌和最大多样性的方法,都是从PCOA的一端蔓延到另一端,代表性差异的方法会从整个PCOA范围内进行选择,最不相似方法则是由PCOA的外围到核心。
无论是哪种方法,在连续挑选时都具有非常明显的变化趋势,说明在选择的过程中,microPITA是有规可循,而不是随机选择,并具有一定的稳定性。microPITA的稳定性可以帮助我们在连续不断的选择过程中,找到最适合的样本数目。
图3. 不同方法挑选连续的样本
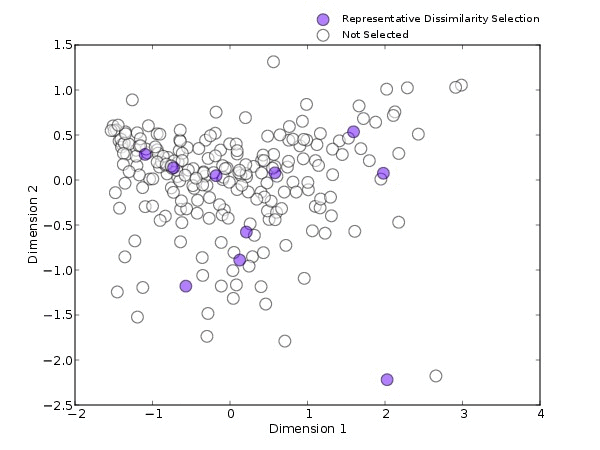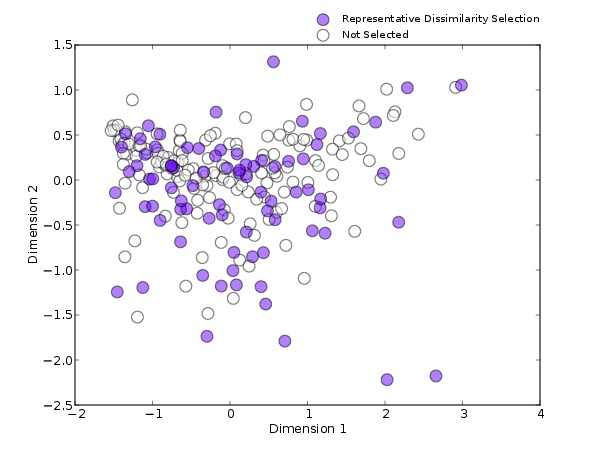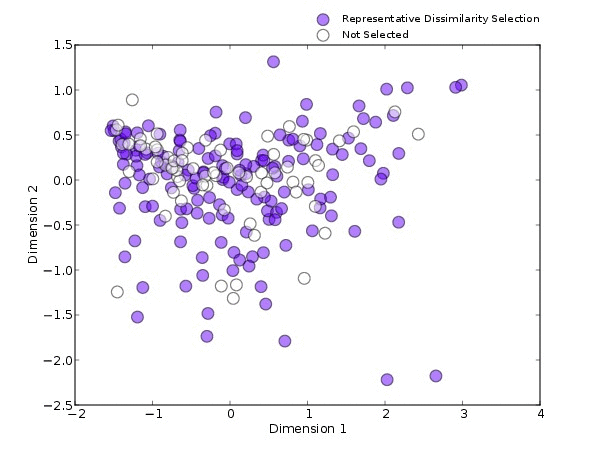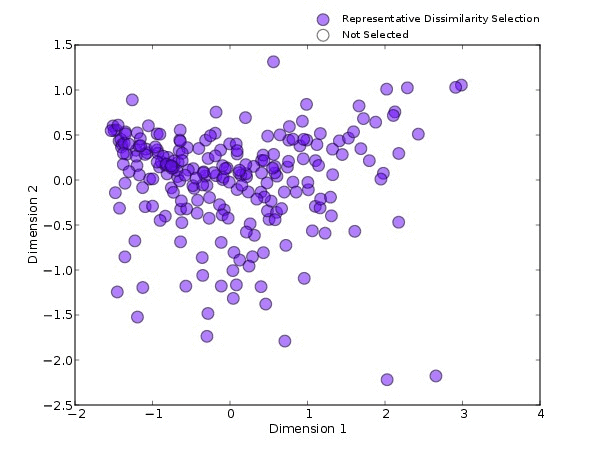
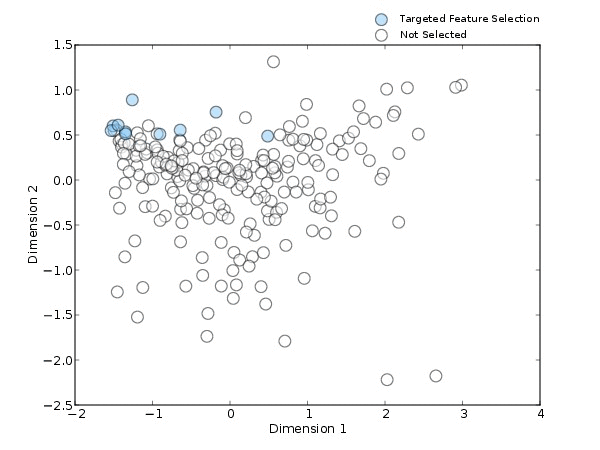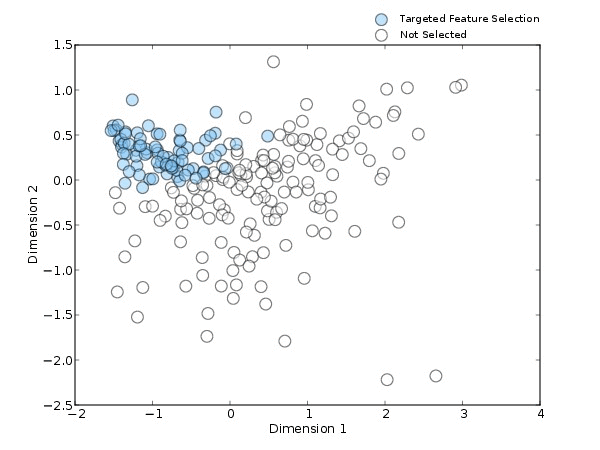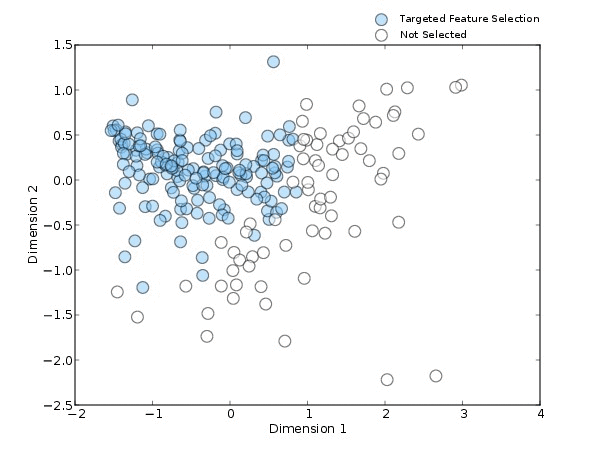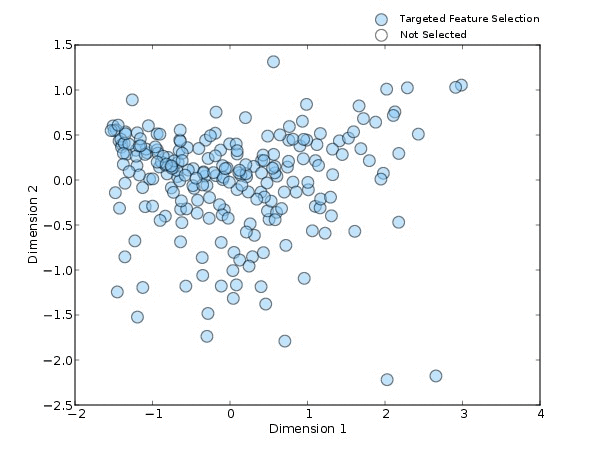
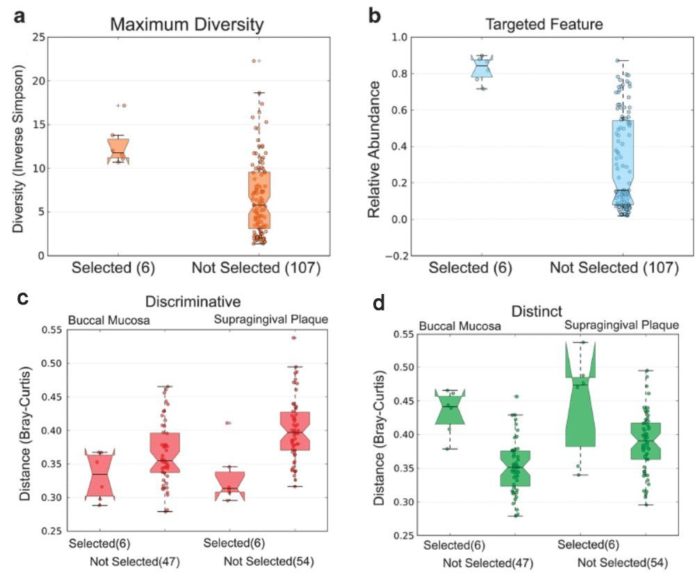
除此之外,我们还需要确认选择后的样本是否保持着原先样本的特性,这一点,可以通过选择样本前后特性的柱状图进行比较。当我们选择生态多样性最丰富的样本进行分析时,选择出的样本仍然保持着多样性(图4a);采用feature的方法时,关心的特征物种的丰度会明显高于之前的样本(图4b)。
当使用有监督的方法时,挑选后结果的差异也很明显,选择Discriminative即分组中心最近的方法时,会挑选具有组间区分度的“同类”的样本(图4c),而选择Distinct即组间β多样性距离最大的方法时,会挑选具有组间区分度“极端”的样本(图4d)。可以看出,不管是哪种方法,最终选出的样本保持着原先的特性,并且更加符合我们的预期。
图4.挑选后的结果,selceted代表利用不同方法所挑选的目标样本,not selected表示所有样本。a,b,c,d图分别是利用不同的挑选方法进行挑选。
3.Application——microPITA如何应用
最后,我们可以来看一篇文章案例,看看高分文章是如何利用microPITA软件进行样本选择[4]。
图5.多组学研究肠道微生物与自身免疫的形成机制
根据卫生假说,在婴儿期早期暴露于特定微生物和寄生虫有利于免疫系统发育,并对过敏性疾病和自身免疫性疾病提供保护。因此作者跟踪了北欧的222名婴儿,观察从出生到三岁的发育情况,发现芬兰和爱沙尼亚(高过敏症人群)的婴儿普遍患有早期自身免疫性疾病,但在俄罗斯(低过敏症人群)相对较少。
因此对这三个国家的婴儿肠道微生物进行了研究,挑选了1548个样本进行16s测序,研究物种组成、多样性和差异分析,接着利用microPITA挑选出785个用于后续研究的样本,进行物种组成和功能分析。
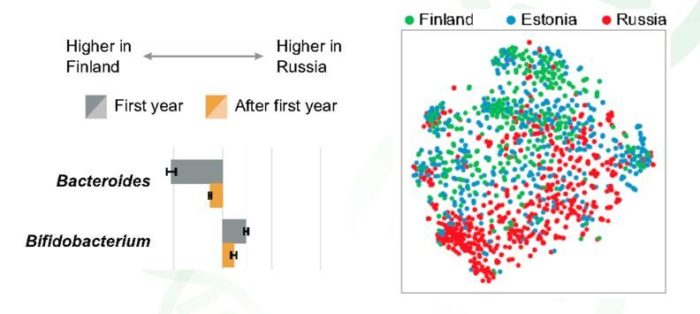
作者通过16s测序发现,芬兰和爱沙尼亚婴儿的肠道中存在大量拟杆菌门的细菌,而俄罗斯婴儿的肠道则为大肠杆菌,表明拟杆菌很有可能驱动人群的自身免疫差异;通过PCA分析,结果显示芬兰和爱沙尼亚人的群落结构更为接近,表示芬兰和爱沙尼亚的自身免疫效果类似(图6)。
图6.16s结果,物种组成(左)和PCA结果(右)
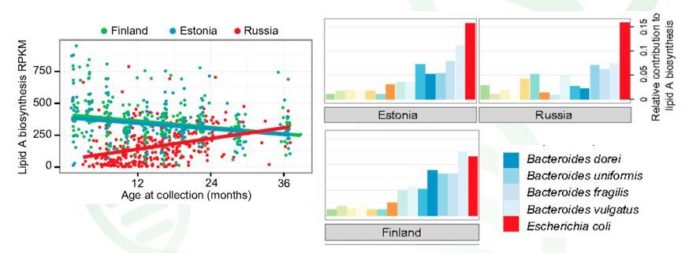
通过microPITA挑选样本后进行宏基因组测序,结果发现芬兰、爱沙尼亚与俄罗斯人群LPS(脂多糖)的合成存在差异,芬兰和爱沙尼亚肠道LPS的合成主要来源于拟杆菌,而俄罗斯则相反,这表示拟杆菌有可能通过LPS合成参与人群的自身免疫调节。
图7.宏基因组结果,不同人群肠道微生物LPS合成模式(左)和贡献物种(右)都存在差异
接着作者针对两种不同菌产生的LPS进行了分子结构、免疫刺激性属性等后续实验,表明拟杆菌LPS在结构上与大肠杆菌的LPS不同,并抑制先天免疫信号和内毒素耐受性,并且发现拟杆菌LPS不能降低非肥胖糖尿病小鼠自身免疫性糖尿病的发病率。从而得出最终结论,在对自身免疫敏感性高的国家的儿童菌群中,拟杆菌产生一种具有免疫抑制特性的脂多糖(LPS),其特性可能会妨碍早期免疫训练并导致I型糖尿病的发生。
这是一篇非常典型的16s+宏基因组关联的案例,文章先进行了16s测序,从中发现了肠道微生物存在物种上的差异(16s侧重于物种组成),接着进一步挑选出样本进行宏基因组测序,对肠道微生物的功能进行研究(宏基因组侧重于功能研究),最后加上功能试验,得出了最终的结论。
想要尝试这两个组学关联分析的老师同学们,microPITA是个非常好的帮手。如果想要进行分析的话,请联系我们哦~基迪奥会为您量身定制专业的个性化实验分析方案。
参考文献:
1.Gevers D, Knight R, Petrosino JF, Huang K,McGuire AL,Birren BW et al. (2012). The human microbiome project: a communityresource for the healthy human microbiome. PLoS Biol 10: e1001377.
2.Yatsunenko T, Rey FE, Manary MJ, Trehan I, DominguezBelloMG, Contreras M et al. (2012). Human gut microbiome viewed across age andgeography. Nature 486: 222–227.
3.Tickle T L, Segata N, Waldron L, et al. Two-stagemicrobial community experimental design[J]. The ISME journal, 2013, 7(12):2330.
4.VatanenT, Kostic A D, d’Hennezel E, et al. Variation in microbiome LPS immunogenicitycontributes to autoimmunity in humans[J]. Cell, 2016, 165(4): 842-853.