氨基酸的疏水性(Hydrophobicity)反映蛋白质的折叠情况,在潜在的跨膜区域会出现疏水区,并且在保持蛋白质的三级结构上(比如维持生物膜的结构)起重要作用。蛋白的亲疏水性(Hydropathicity/ Hydrophobicity)图谱可为蛋白跨膜区域的鉴定提供参考。下面为大家介绍一个绘制蛋白亲疏水性图谱的在线工具:ProtScale。
网址:
https://web.expasy.org/protscale/
序列输入
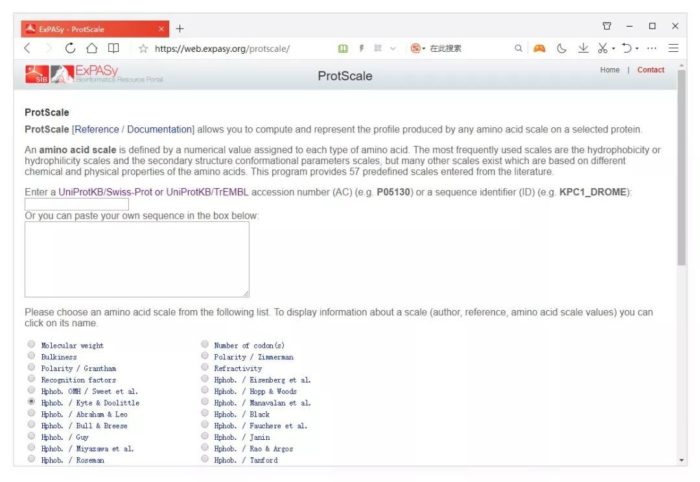
它的首页是这样子的:
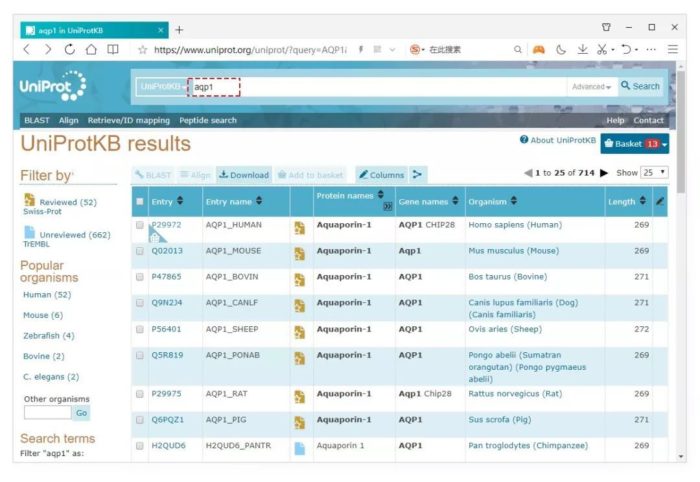
接下来,以AQP1蛋白序列为例看下如何用它分析蛋白的疏水性,只需要提供UniProt的登录号或序列ID即可,可直接点页面中的链接到Uniport数据库查找,如下图。关于Uniprot的使用方法可参考《听说画好看的进化树需要知道这2个数据库》一文,当然你也可以直接把蛋白序列粘贴到序列框中。
参数设置
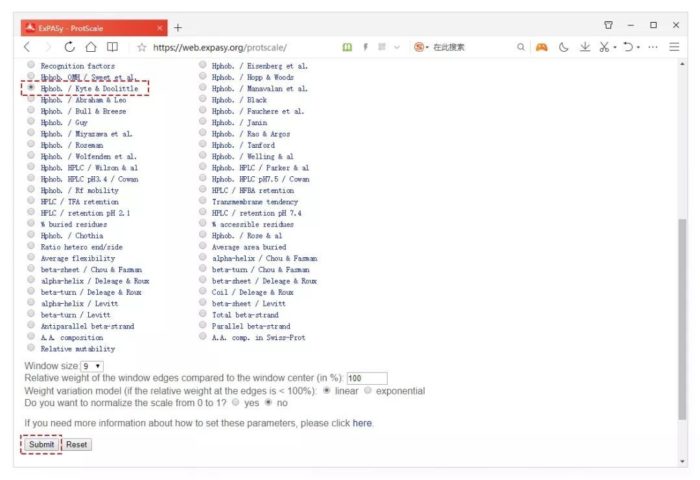
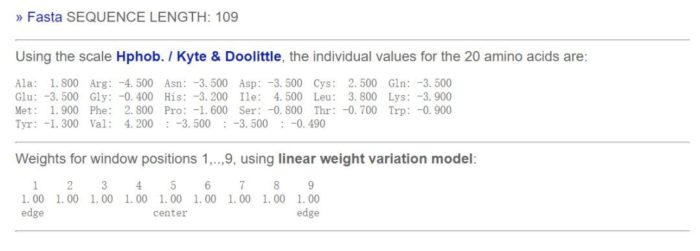
Amino acid scale选择默认的Hphob. / Kyte & Doolittle,滑窗大小为9,线性加权模型,如下图。
ProtScale可计算和展示所选蛋白的50多种氨基酸标度(Amino acid scale)的图谱,氨基酸标度是给每种氨基酸指定的数值,比如Molecular weight(分子质量)、Number of codon(密码子数)、Bulkiness(膨胀度)等等。最常用的标度是疏水性标度,主要来源于多肽在非极性和极性溶剂中的分配实验数据。
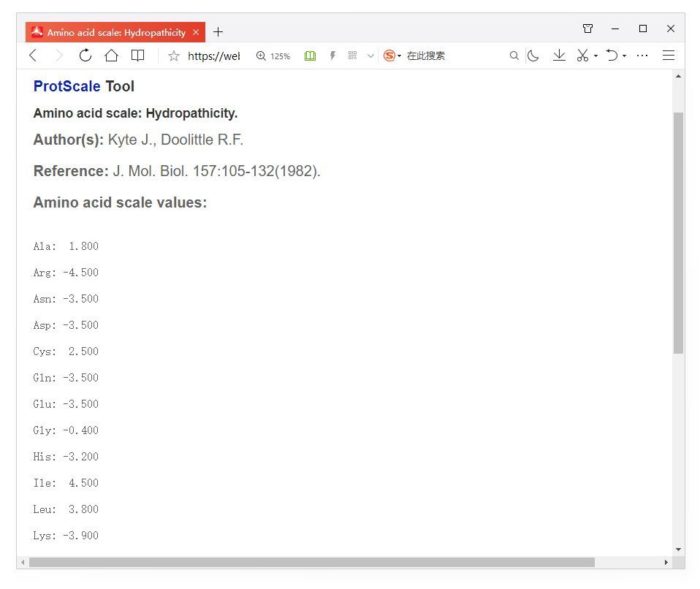
如果你对所选标度感兴趣可点击标度名称可进入详情页面,查看具体的氨基酸的标度数值和参考文献,如下图,这里正值的氨基酸具有更大的疏水性,负值越小的氨基酸则更加亲水。
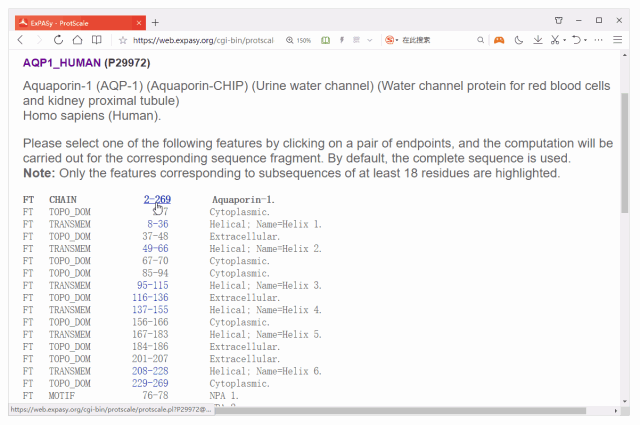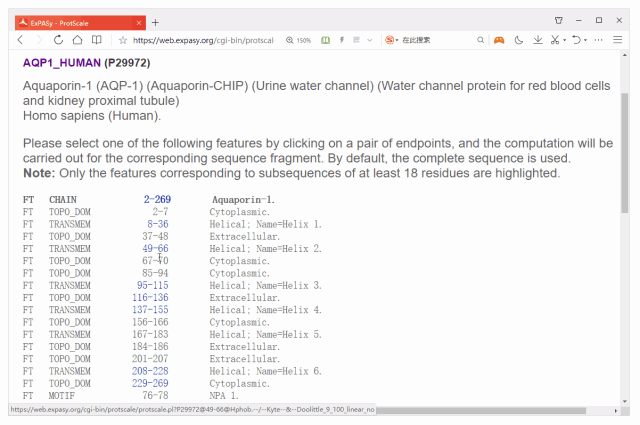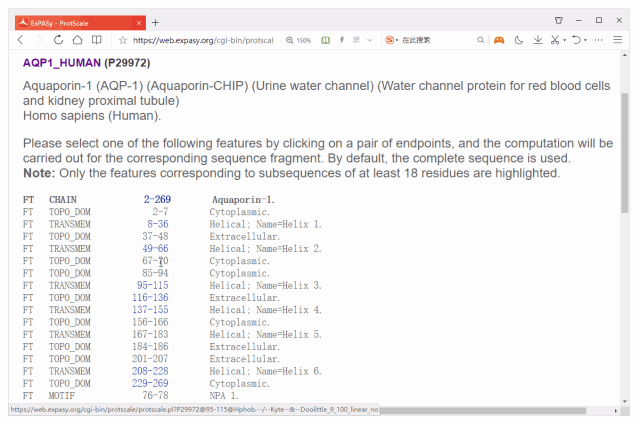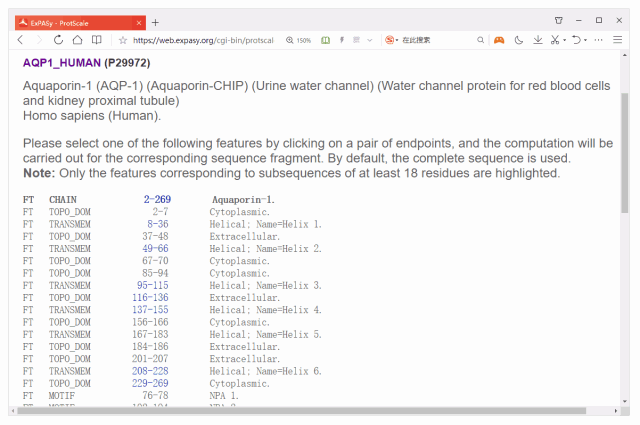
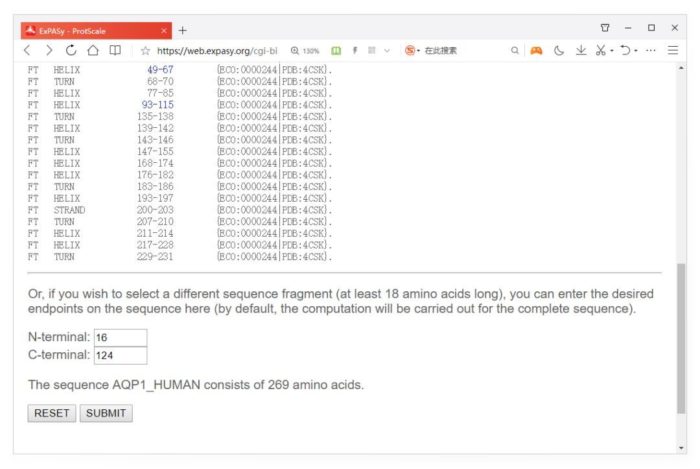
点击Submit按钮后,会给出蛋白的结构注释信息,你可通过单击蓝色数字选择分析区域,如下图。
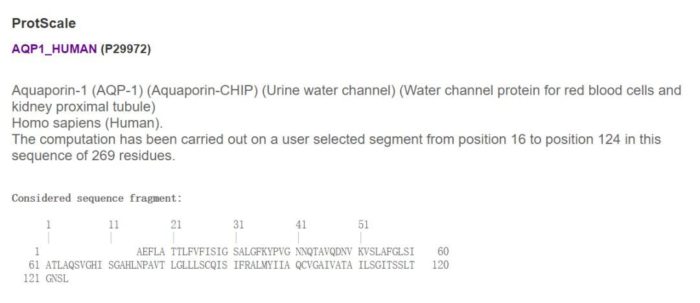
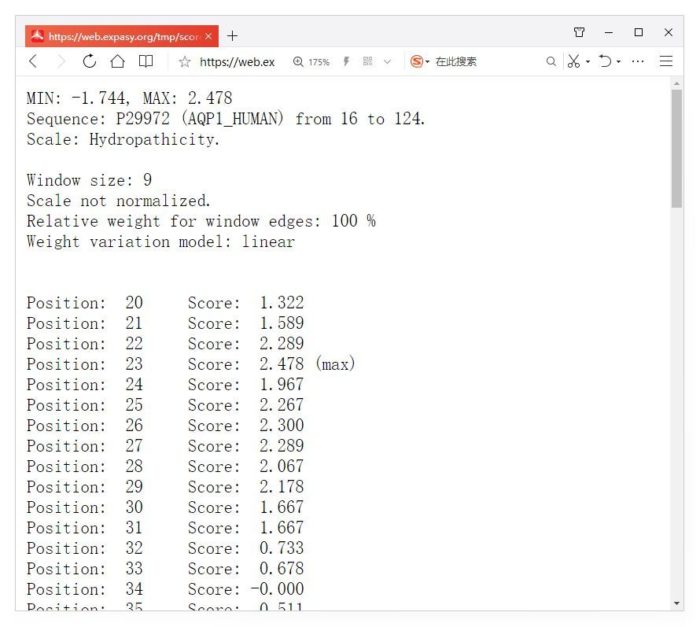
若不选则默认是对整个序列进行分析,当然你也可以自定义区域,因为滑窗的大小为9个氨基酸,下图输入的16-124,而点SUBMIT得到是20-120这个区域的结果。
结果分析
在结果页面首先给出蛋白序列信息和所选区域的氨基酸序列长度、滑窗的大小等,如下。
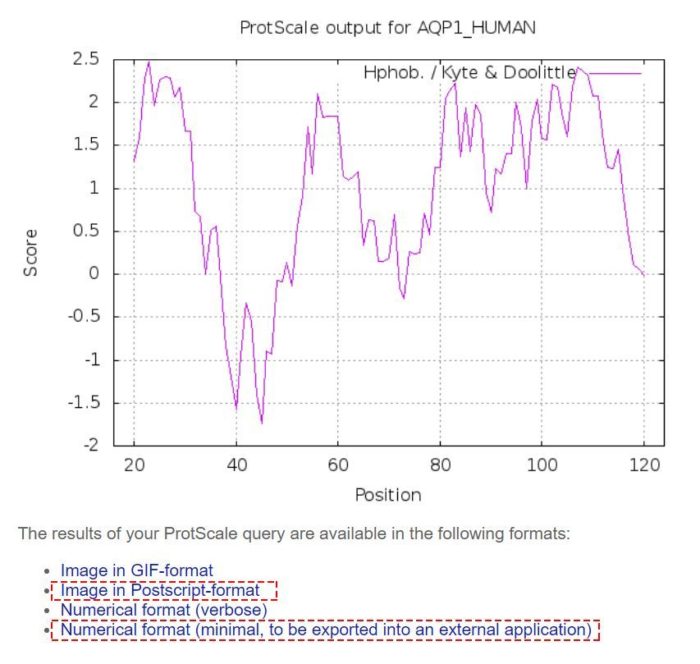
下面是最终的结果,图形的高峰值(正值)的区域表示疏水的区域,而负值的“低谷”区域是亲水区域。你可点下面的链接下载gif和PDF(会自动转换)图片和作图数据。
数据的保存方法是在相应网页(如下图)右键,另存为txt文件。
这里的数据是用空格分隔的,可用文本编辑期的查找替换功能将空格替换为制表符“t”,这样就可以直接粘贴到Orgin或Excel中自定义作图了,注意,结果中minimal版的数据更方便于作图。
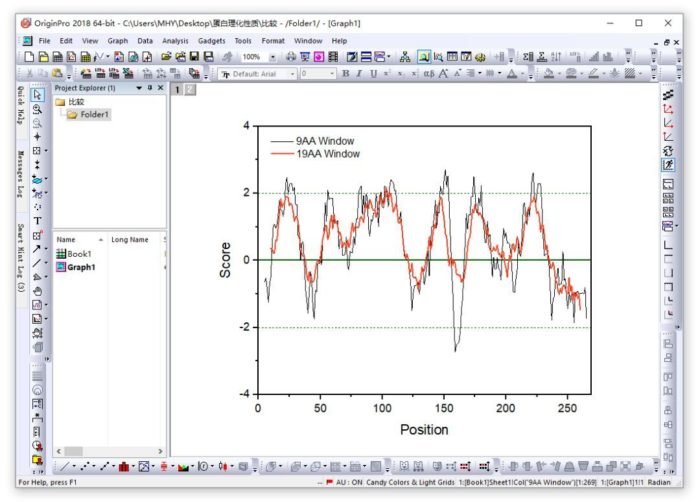
下图是对整个蛋白序列进行分析,滑窗大小分别选9和19的分析结果。对导出的数据用Origin作图更好看一点,滑窗选择19时杂峰较少,可明显看出整个序列有6个高峰(疏水区)。
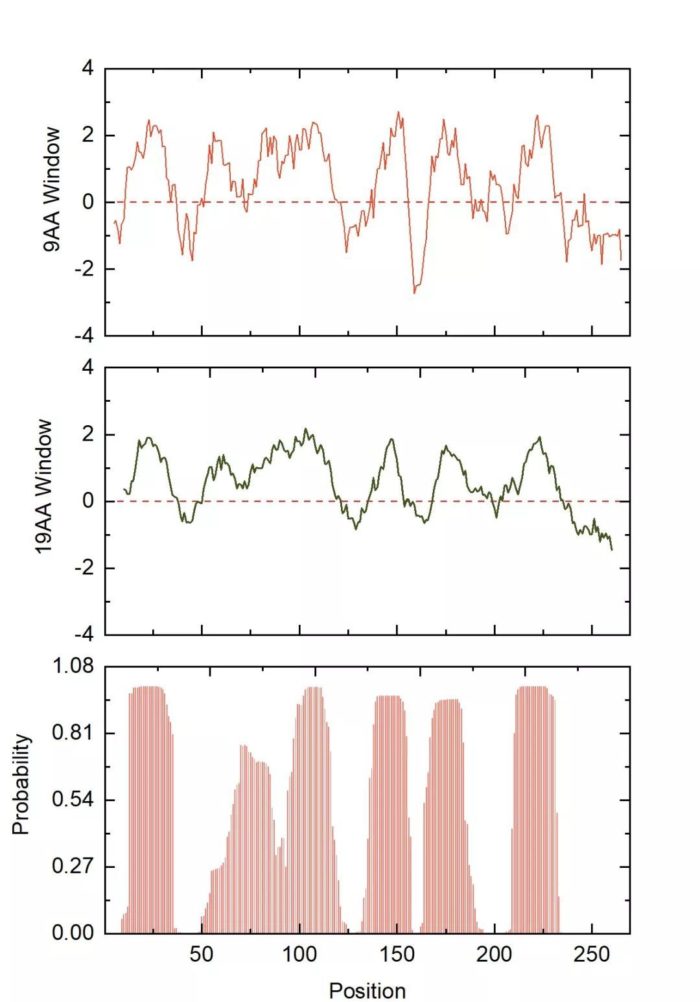
为了进一步确认更精确的区域,你也可以结合TMHMM 对跨膜区域的预测结果进行分析,一般来说,蛋白的跨膜区域为疏水区,而膜外的部分为亲水区。TMHMM的使用教程参见《如何预测蛋白的跨膜螺旋?》一文,为了便于比较预测的结果这里用Origin的“堆叠”图展示,如下图,表明TMHMM和ProtScale的预测结果非常一致。
参考文献
Kyte J,Doolittle R F. A simple method for displaying the hydropathic character of aprotein.[J]. Journal of Molecular Biology, 1982, 157(1):105-132.
http://blanco.biomol.uci.edu/hydrophobicity_scales.html
https://en.wikipedia.org/wiki/Hydrophobicity_scales