在之前的3篇关于EvolView文章中已介绍过进化树的调整和注释,并重点介绍了进化树与条形图、热图、蛋白结构域的组合操作。这次为大家介绍另一种常见的组合图:进化树+点状图。
由于内容上有一定的关联,如果是初次接触EvolView小伙伴建议大家先看一下《进化树+条形图:“组合图”的绘制》的内容。
点状图(Dot plots)是条形图(Barchart)的一种“变体”,条形图用“条”的“长短”展示数据,而,点状图用“点”的大小展示数据。因此,本文用到数据与《进化树+条形图:“组合图”的绘制》一文的数据完全一样。
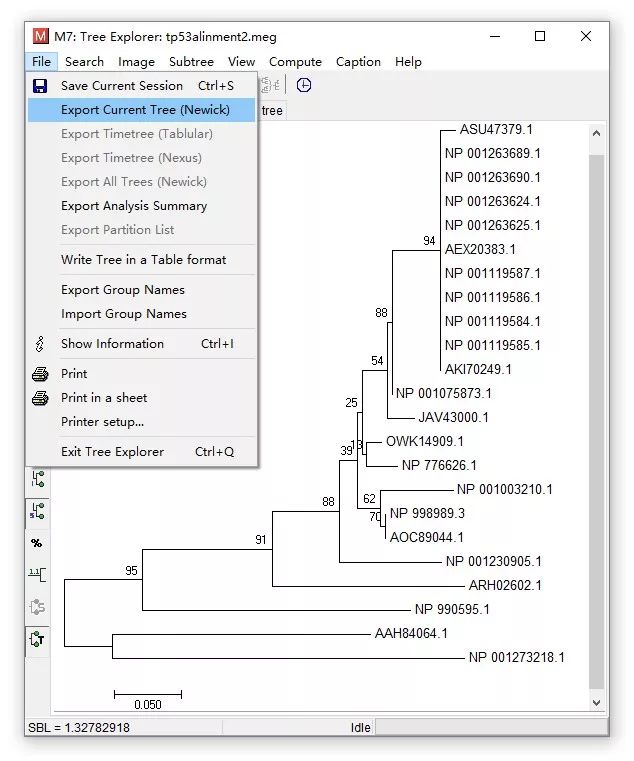
这里的进化树用MEGA构建,将进化树导出为Newick格式,方法如下(已介绍过多次)。
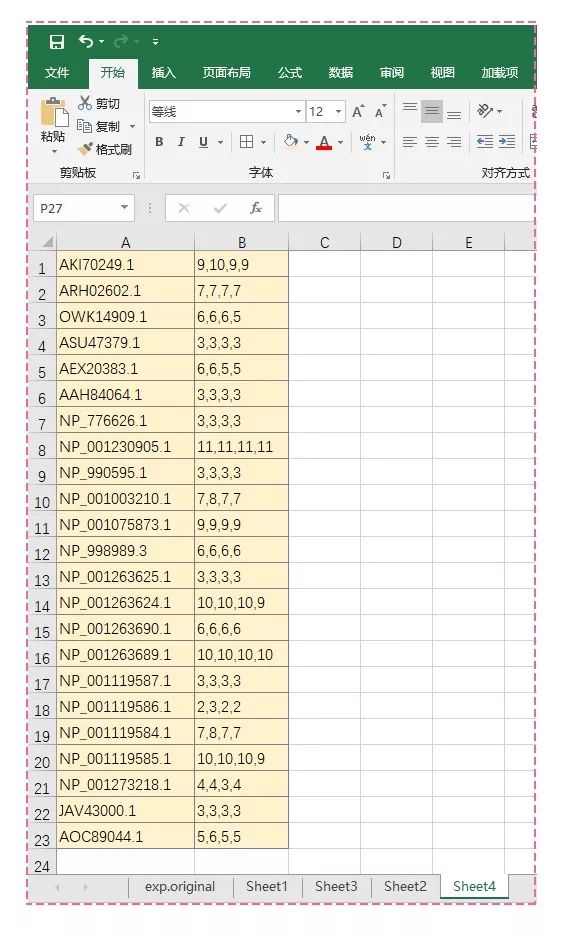
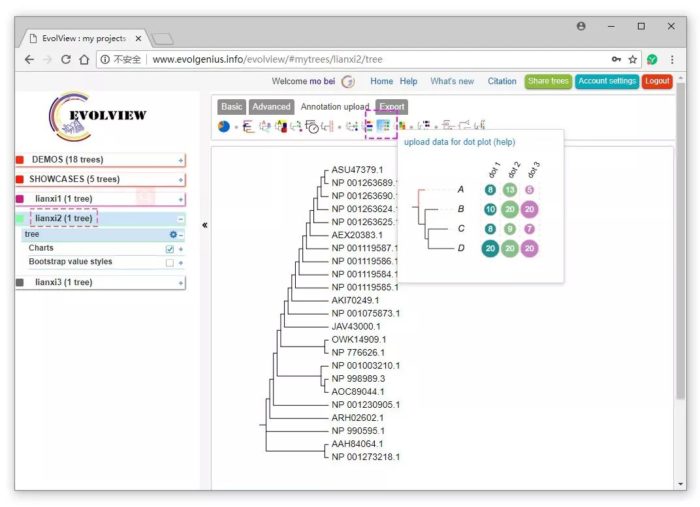
另一个需要准备的是用来表示“点”大小的数据,每一个gene ID(或基因名称)对应所需展示的数据(用英文“逗号”隔开),不需要考虑gene ID聚类顺序,如下图。
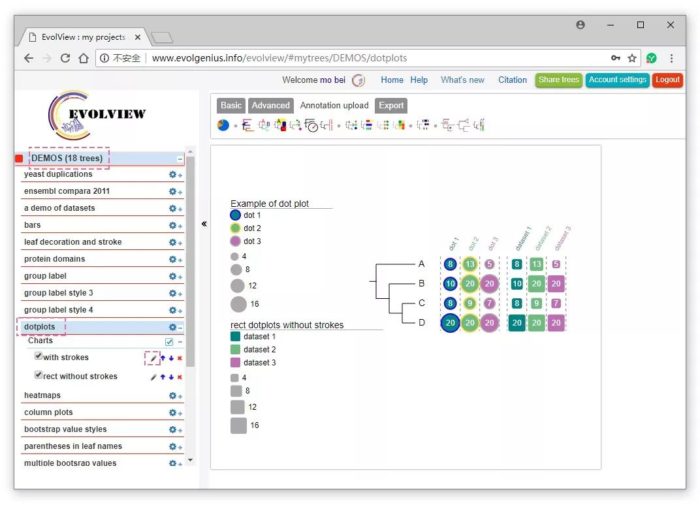
首先,在Basic选项下,载入从MEGA中导出的Newick树,方法参见《进化树+条形图:“组合图”的绘制》,这里仍以之前的lianxi2工程文件为例(注册账号后工程文件可反复使用)。然后点DEMOS,点“✏“图标查看EvolView自带的点状图的示例文件。
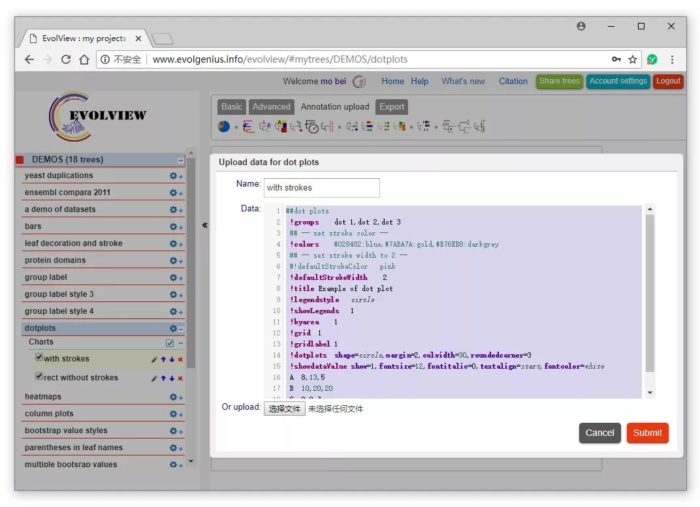
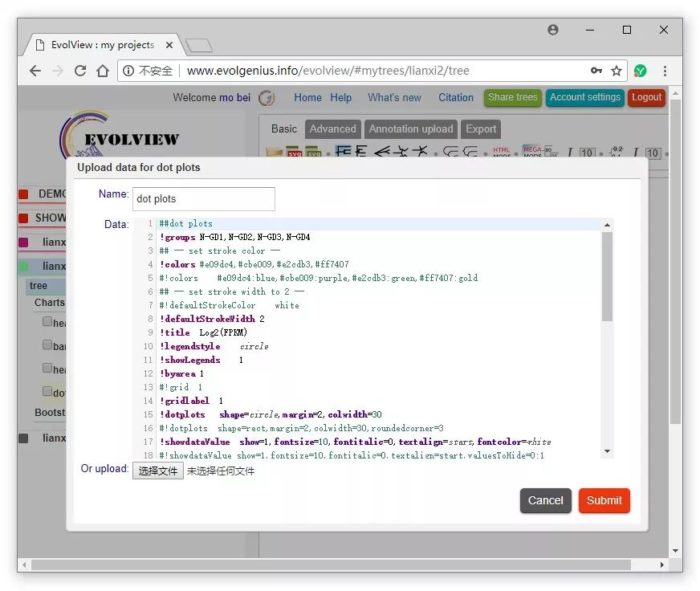
我们然后“Ctrl+a”全选“注释文件”,如下图,“Ctrl+c”将“注释代码“复制到剪贴板,点Cancel按钮退出编辑,然后粘贴到记事本、Notepad等文本编器中进行编辑。
接下来需要根据自己的数据,在文本编辑器中更改示例的注释文件,我将需要改动的部分标记为绿色。为了方便大家使用,我在示例的“注释文件”的基础上由补充了一些参数。
Tips:
通过在句首添加/删除“#”号来控制相应参数是否有效来实现更丰富的个性化调整。“!”表示可执行的“参数”,“#”表示备注、注释不被执行。
- ##dot plots
- ##设置列名称;
- !groups N-GD1,N-GD2,N-GD3,N-GD4
- ## 设置“点”的颜色和点的轮廓颜色,冒号后的为轮廓颜色,这里仅执行上一句,不显示轮廓颜色;
- !colors #e09dc4,#cbe009,#e2cdb3,#ff7407
- #!colors #e09dc4:blue,#cbe009:purple,#e2cdb3:green,#ff7407:gold
- ##设置轮廓默认颜色,这里不执行;
- #!defaultStrokeColor white
- ##设置轮廓粗细
- !defaultStrokeWidth 2
- ##设置图例的标题、形状、是否显示(0为不显示);
- !title Log2(FPKM)
- !legendstyle circle
- !showLegends 1
- ##设置数据与点的面积的对应关系;
- !byarea 1
- ##是否显示参考线;
- #!grid 1
- #!gridlabel 1
- ##“点”的形状、大小设置;
- !dotplotsshape=circle,margin=2,colwidth=30
- #!dotplots shape=rect,margin=2,colwidth=30,roundedcorner=3
- ##“点”上是否显示数据、字体设置、隐藏数据范围设置;
- !showdataValue show=1,fontsize=10,fontitalic=0,textalign=start,fontcolor=white
- #!showdataValueshow=1,fontsize=10,fontitalic=0,textalign=start,valuesToHide=0:1
- ##列标签的字体、加粗、角度设置;
- !columLabelStyleshow=1,fontsize=14,fontbold=1,textangle=-60
- ##将准备的数据粘贴到这里:
- AKI70249.1 9,10,9,9
- ARH02602.1 7,7,7,7
- OWK14909.1 6,6,6,5
- ASU47379.1 3,3,3,3
- AEX20383.1 6,6,5,5
- AAH84064.1 3,3,3,3
- NP_776626.1 3,3,3,3
- ……
接下来,在Annotation upload 选项下单击相应按钮(如下图)新建一个注释文件。
Name这里命名为dot plots,将刚才编辑的内容全选后复制的“代码”粘贴到data区,如下图。
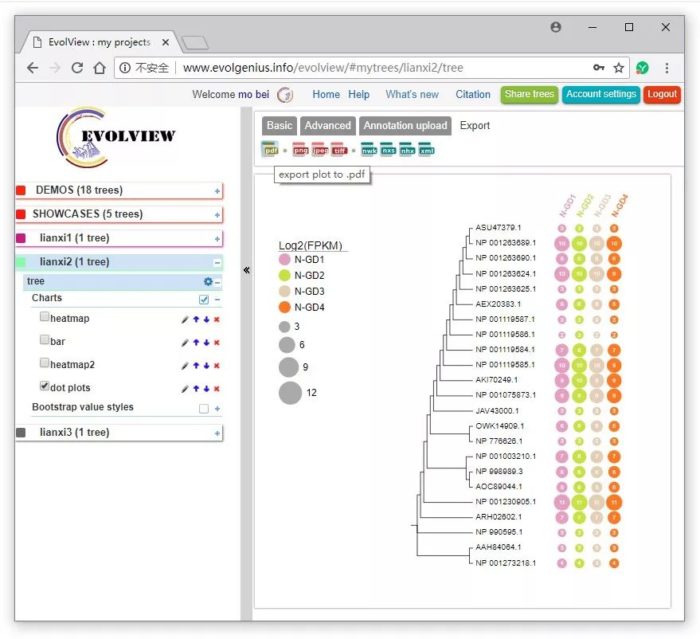
然后点Submit按钮,即可完成“注释”,点“✏”图标,可对热图的注释文件进行反复编辑,进而达到精细调整,最后通过Export菜单(如下图)导出所需格式的图片。
这里导出PDF格式的矢量图,点击下载链接,在新的网页预览、下载pdf图片。
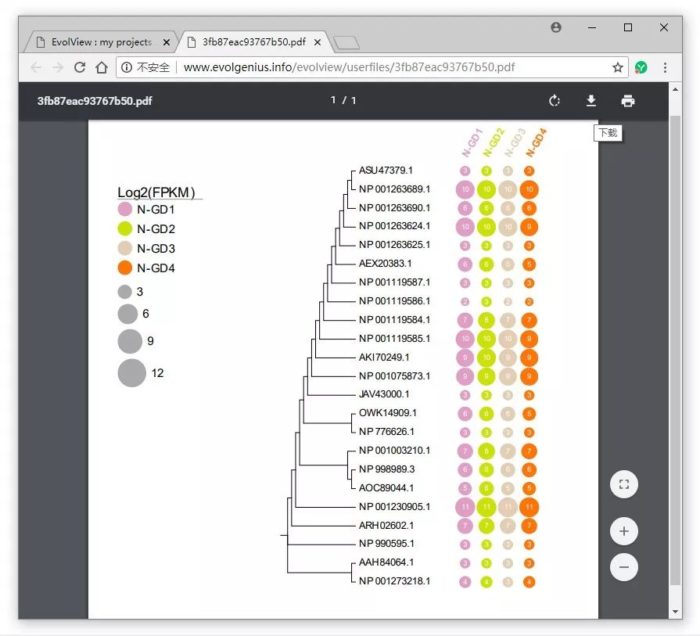
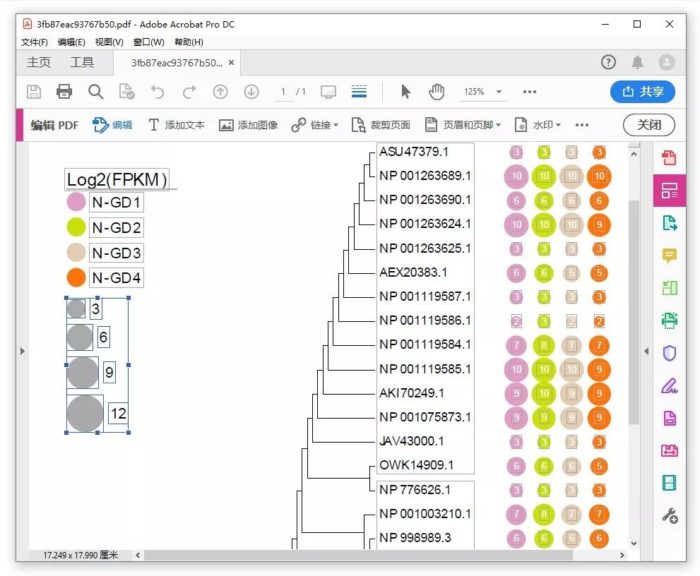
如果你还对图片不满意,可以用Adobe Illustrator或Adobe Acrobat中进一步编辑pdf文件。比如这里简单在Acrobat中框选图例的灰色部分,然后按Delete键删掉,然后选中图例部分做位置的移动。
最终的效果如下图: