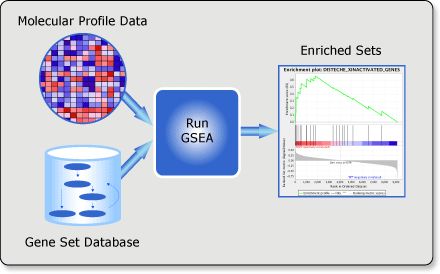
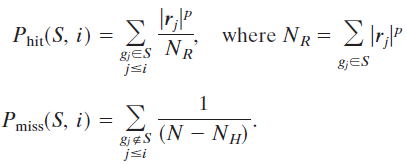Gene Set Enrichment Analysis (GSEA) is a computational method that determines whether an a priori defined set of genes shows statistically significant, concordant differences between two biological states (e.g. phenotypes).
用GSEA做富集分析是非常简单的,结果也很详细,并且直接出图;这个软件发表于2005年,一直都在不断更新和增加新的功能;软件基于的数据库Molecular Signatures Database也会根据新发表的文章进行完善。
GSEA软件版本了解
- GSEA设计了操作比较简单的桌面软件;
- GSEA也提供在无网络情况下的一个命令操作版本;
- 基于R的版本,但是2005后不再提供更新;
- GenePattern平台也有GSEA模块。
GSEA软件下载与安装
GSEA download
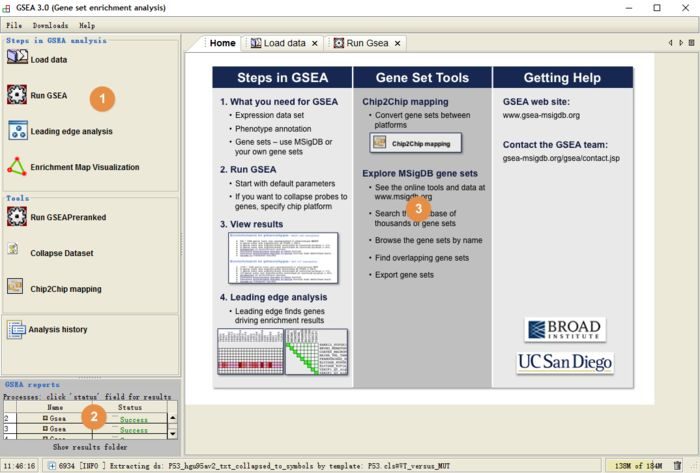
GSEA界面
1).圈1所在是导航栏,展示主要操作;
2).圈2是进度栏;当你进行分析时,查看分析进程与成功与否;成功后在此处可以查看网页版结果;
3).圈3是主页面,在此进行各种操作与分析;
GSEA运行
官网也准备了例子:Example Datasets(http://software.broadinstitute.org/gsea/datasets.jsp)
这儿使用P53这个例子:
- p53+ 与P53突变癌细胞系的表达谱
- Molecular Signatures Database C2数据基因集合
1. 下载数据
P53.cls #表型文档定义了表达文档中样品的表型标签,使用空格或tab隔开;
P53_collapsed_symbols.gct #基因表达谱数据
P53_hgu95av2.gct #基因芯片表达谱数据
GSEA软件需要的数据格式可参考:GSEA软件支持的数据格式
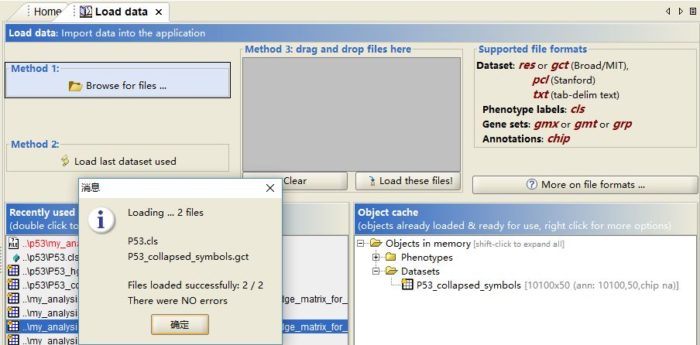
2. 点击导航栏Load data导入数据
3种不同的方法均可以导入数据:
- Method 1: Browse for files #上传各种文件;
- Method 2: Load last dataset used #使用最近用过的数据;
- Method 3: Drag-and-drop the files hereke #把文件拖曳至此处上传;
导入例子数据(p53)
P53_collapsed_symbols.gct #基因表达谱数据
P53.cls #表型文档定义了表达文档中样品的表型标签,使用空格或tab隔开;
导入数据需要没有报错: There were NO errors
3. Run GSEA
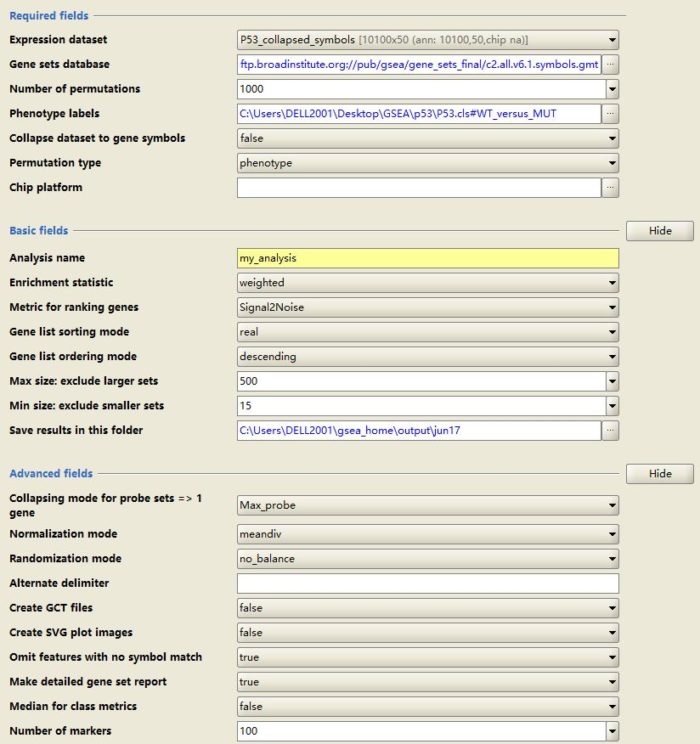
点击软件导航栏Run GSEA,选择数据并进行参数设定;
参数主要分为三部分:
- Required fields: #必须设置的参数
Expression dataset: #选择要分析的表达数据,P53_collapsed_symbols.gct。
Gene sets database: #选择基因集 ,Molecular Signatures Database,MSigDB
Number of permutations: #样品用于置换检验检验重复次数,一般1000。
Phenetype labels: #选择表型数据。
collapsed to gene symbols: #默认true,表达数据中探针名转换成gene symbols;
P53_collapsed_symbols.gct中是已经转换为基因名字,不需要这一步,选择false;
Permutation type: #phenotype用于每个表型组至少7个样本的实验;Gene_set用于表型组样本数少于7个的时候。
Chip platform: #选择Chip注释文件,用于collapsed to gene symbols这一步;
- Basic fields: #可选参数
Analysis name: 设定分析结果前缀
Metric for ranking genes:选定对基因打分和排序的模式;
Gene list sorting mode:基因排序可以选择使用原值(default)和绝对值。
Gene list ordering mode:基因排序是递增还是递减。
Max size:基因集基因数目上限。
Min size:基因集基因数目下限。
Save results in this folder:结果保存路径
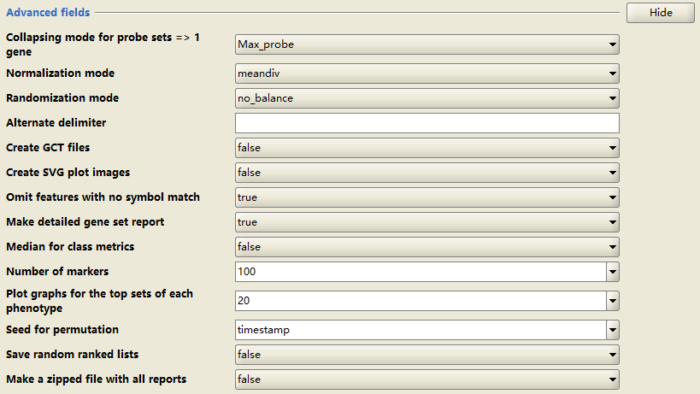
- Advanced fields: # 高级参数
建议使用默认,不要随意改动。
Collapsing mode for probe sets => 1 gene:#使用芯片数据时,基因表达值的计算;
max_probe (default):#芯片集中最大值作为基因表达值;
median_of_probes: #芯片集均值作为基因表达值
Normalization mode: #富集分数( Enrichment scores,ES)的标准化方法;
Normalized Enrichment Score (NES)方法:![]()
Randomization mode:
no_balance (default):完全随机抽样
equalize_and_balance:分别从不同表型组抽取相同数目样本;
4. 运行及处理进程观察
参数设置完成之后,点击run开始运行;左下角GSEA reports板块可以检测运行情况;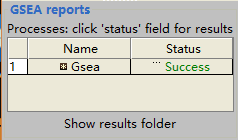
Running:正在分析,可以暂停;Success:分析成功,点击Success,可以查看网页报告;Error:分析出错,点击Error,查看出错详情;
5. 结果查看
5.1 GSEA结果中的统计量:
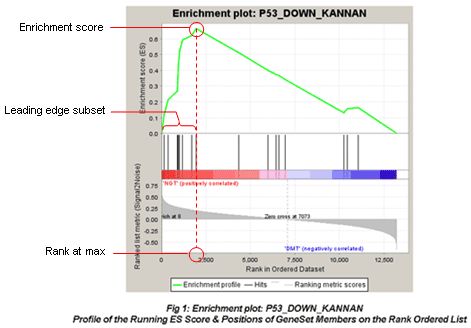
● Enrichment Score (ES)
● Normalized Enrichment Score (NES)
● False Discovery Rate (FDR)
● Nominal P Value
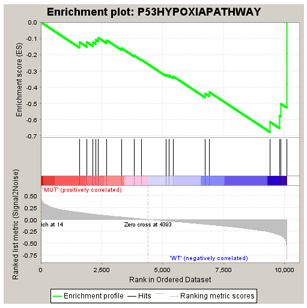
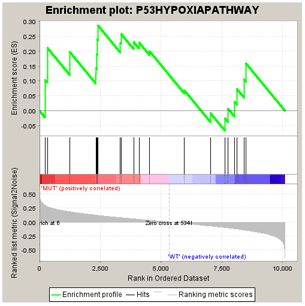
Enrichment Score (ES)
- 最上面的绿线是遍历排好序的基因列表是计算ES值的过程:遍历基因集L ,当基因出现在S中加分,反之减分;加减分值由基因与表型的相关性决定。当分值累积到最大时就是富集分数。
ES值:Phit -Pmiss最大值
预先定义的基因集S;待分析基因列表L;指数P的选择用来控制ES分布;r(gj)=rj 是定义的基因与表型的相关性系数。
L中第i个基因前有基因j也属于基因集S,Phit(S,i)=Phit(S,i)+|rj|p /NR ;与之相反,L中第i个基因前有基因j不属于属于基因集S时,Pmiss(S,i)增加。
- 中间黑线位置表示预定义基因集中基因在排好序的基因列表中的位置;
- 底部展示基因排列的一个度量分数,正数表示与第一个表型相关,负数表示与第二个表型相关;对于连续性表型的话,正数表示相关,负数表示不相关;
Normalized Enrichment Score (NES)
NES是基于样本的置换检验π,样本重新抽样使得基因表达值变化从而影响到基因排序和ES(S, π)。
False Discovery Rate (FDR)
一般情况下可用FDR<0.25;如果样本较少以至于Permutation type使用了 gene_set,FDR<0.05更合适。
这儿,FDR有两种分布:
Nominal P Value
置换检验中ES(S)统计分布中无效假设成立时ES的比率。
5.2 设置的结果生成路径下会有结果生成:
基因列表排序:例如P53_collapsed_symbols.P53.cls_WT_versus_MUT.rnk
基因集结果网页版:例如AMUNDSON_DNA_DAMAGE_RESPONSE_TP53.html
基因集结果统计表:例如AMUNDSON_DNA_DAMAGE_RESPONSE_TP53.xls
以及一些图。。。。。。
6. Running the Leading Edge Analysis
After running a gene set enrichment analysis, you can use the leading edge analysis to examine the genes in the leading edge subsets of selected enriched gene sets. Genes that appear in multiple subsets are more likely to be of interest than those that appear in only one.
6.1 左边导航栏点击Leading Edge Analysis;
6.2 导入数据:点击Load GSEA Results导入刚才分析完的P53的结果;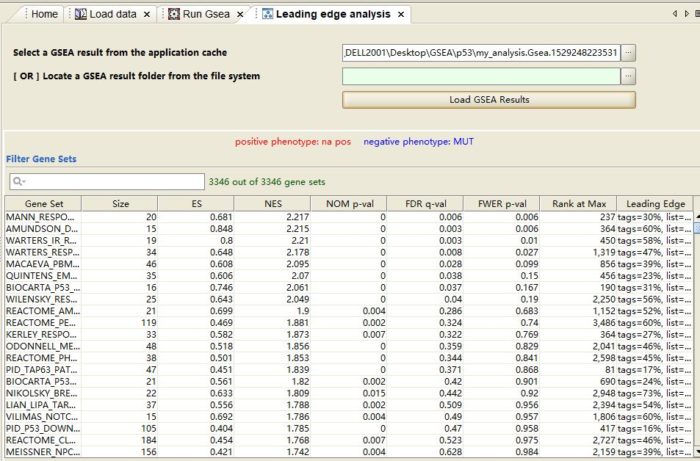
6.3 选择基因集:点击数据每列列名,调整数据排列顺序,选择基因集(FDR < 0.05);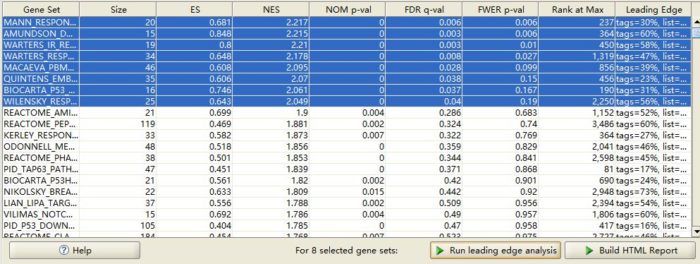
6.5 结果输出
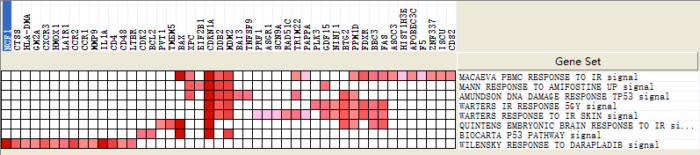
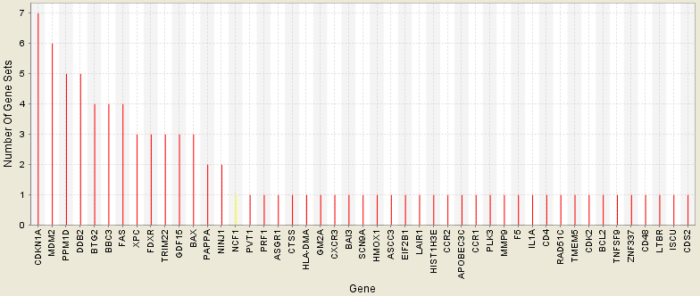
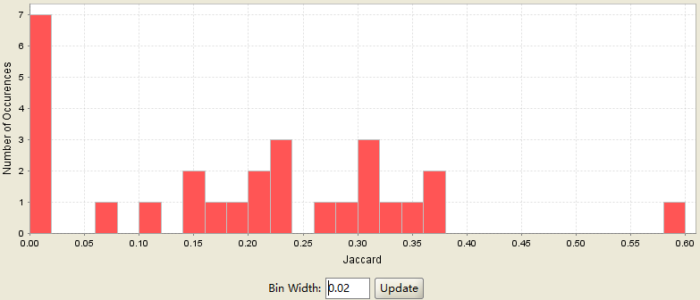
结果是四幅图,解读可参考( Interpreting Leading Edge Analysis Results)
Heat Map
不同基因集中富集基因表达情况:颜色 (red, pink, light blue, dark blue) 表示着表达值高低 (high, moderate, low, lowest)。
Set-to-Set
Gene in Subsets
Histogram
参考:
Quick Tour of the GSEA Java Desktop Application(http://software.broadinstitute.org/gsea/doc/desktop_tutorial.jsp)
GSEA User Guide(http://software.broadinstitute.org/gsea/doc/GSEAUserGuideFrame.html)
Molecular Signatures Database v6.1(http://software.broadinstitute.org/gsea/msigdb/index.jsp)
Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles.