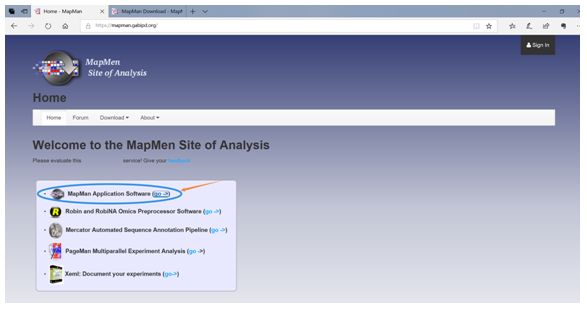
第一步:打开下载链接,点击Mapman Application Software (go)
下载地址: https://mapman.gabipd.org/home
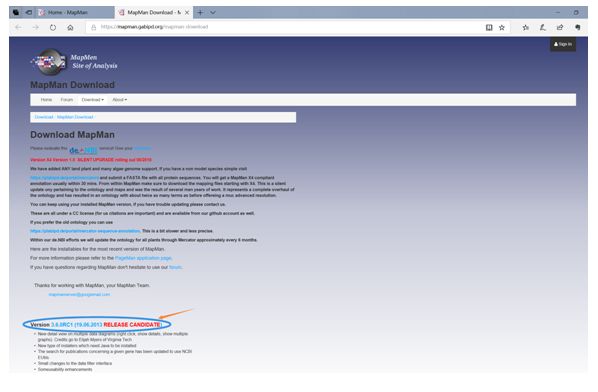
第二步:选择下载版本—Version 3.6.0RC1
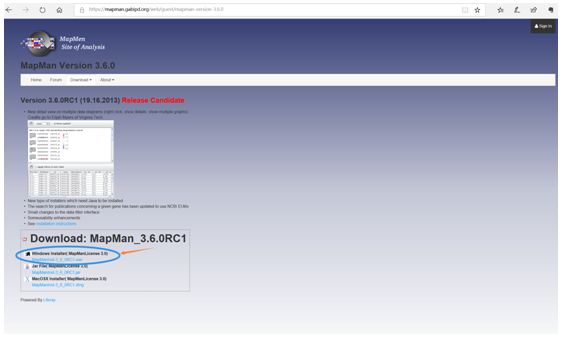
第三步:选择Windows版本或者Mac版本,本文以Windows版本进行演示;
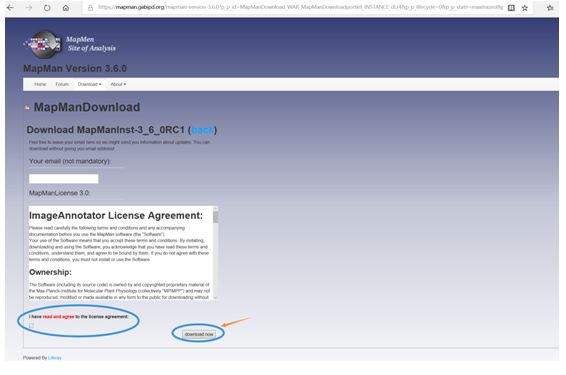
第四步:勾选上(I have read and agree),点击download now,即可开始下载;
第五步:下载完成后,双击文件进行安装;
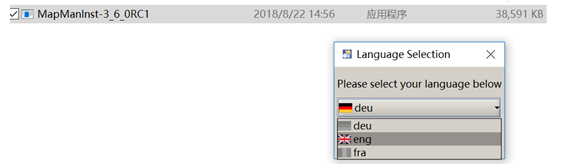
1、 选择语言,常用英文,如果擅长别的语言则可选择其他;
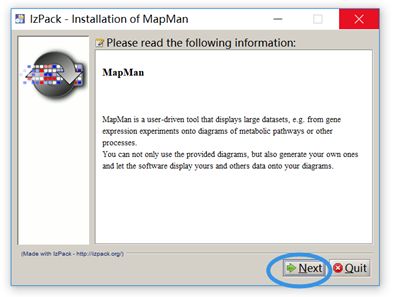
2、点击Next
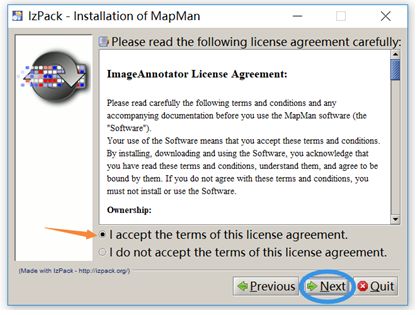
3、选择I accept the terms of this licenses agreement,点击Next
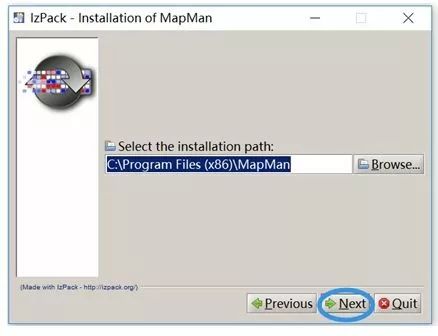
4、选择安装位置,点击Next
5、 安装成功后,即可开始使用
到这里,第一步软件安装终于完成了~
开始正式使用
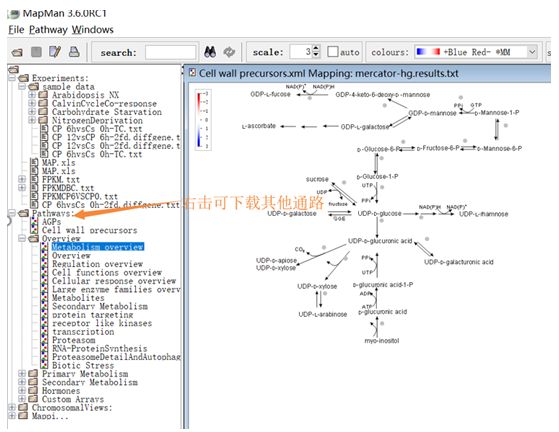
第一步:正式进入界面,左侧分为:Experiments,Pathways,Chromosomal Views,Mappings;
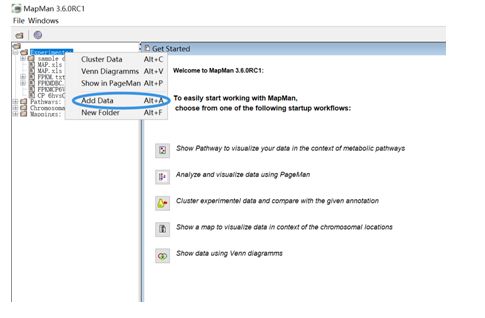
第二步:导入实验数据
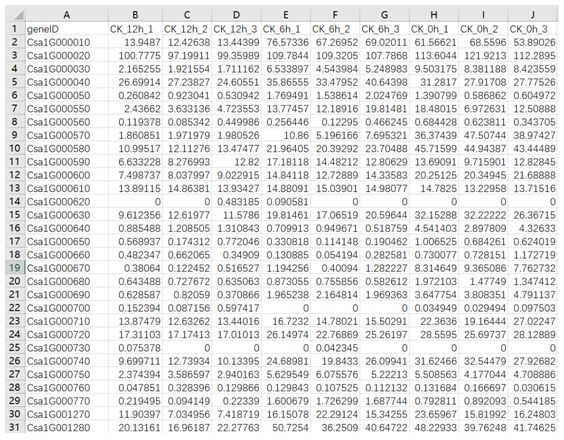
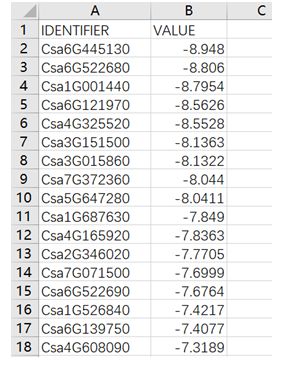
数据类型包括转录组测序得到的基因表达量列表、差异比较分析列表,表格内容如下:
基因表达量:
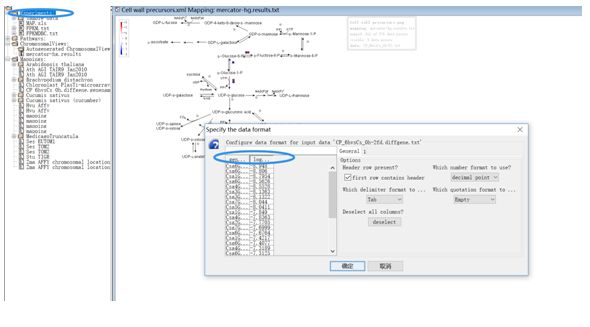
差异基因及差异倍数(数据中不允许出现NA或者INF等字符,如出现则需要修改为字母X):
成功导入数据如下:
第三步:通路图准备
系统中自带的通路图基本可满足需求,可直接选择或者右击选择download,当然也可导入自己准备的通路图;
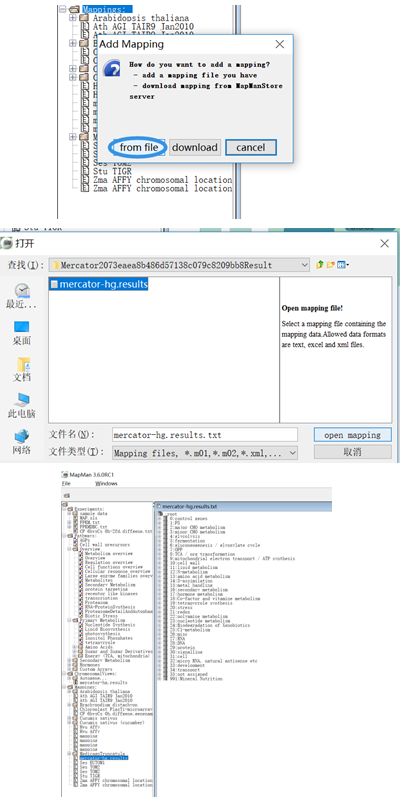
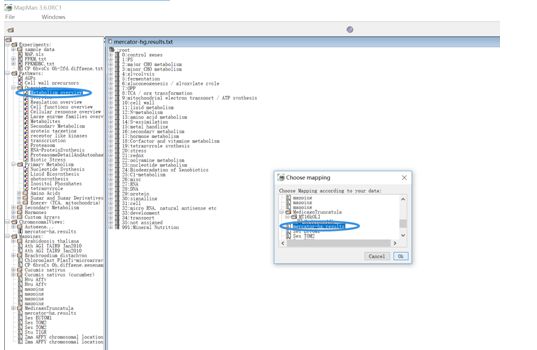
第四步:准备mapping文件
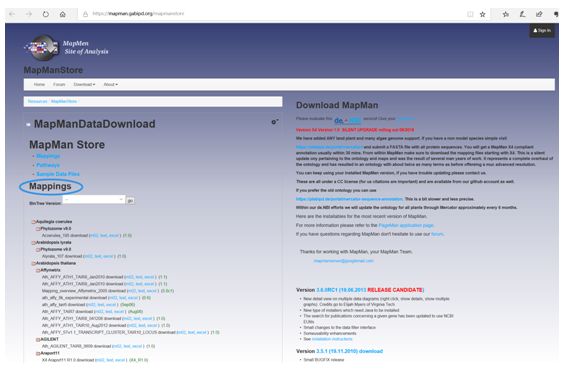
1、数据库中下载模式物种,链接为:https://Mapman.gabipd.org/Mapmanstore
注意:mapping文件中的ID与导入的数据ID必须是配套的,否则无法使用;
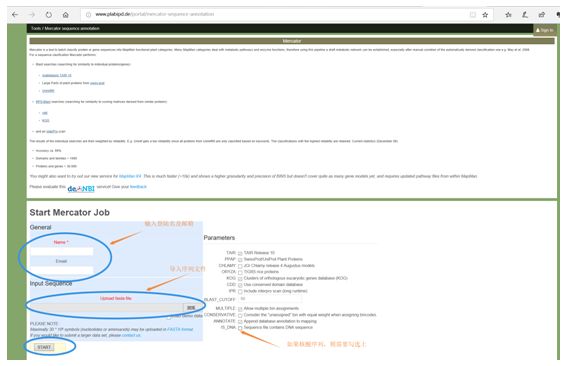
2、数据库中如无对应的文件,则需要自行准备,如果这样则使用到了注释网站:http://www.plabipd.de/portal/mercator-sequence-annotation,步骤如下:
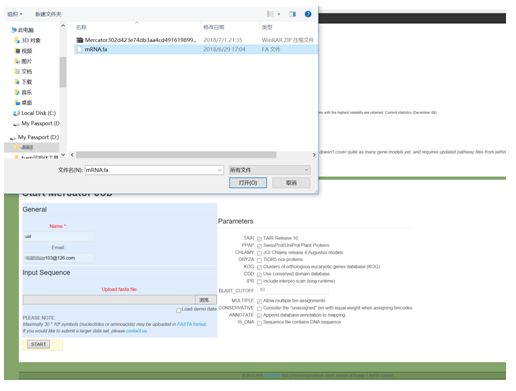
2.1 准备序列文件,格式为fasta的核酸序列或其他序列;
2.2 进入网站界面,输入登陆名及邮箱;
2.3导入序列文件,选择对应参数,核酸序列勾选上contains DNA,其它默认;
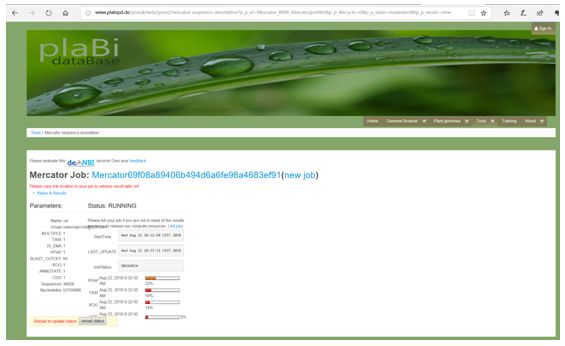
2.4 最后点击START,即开始进行注释;
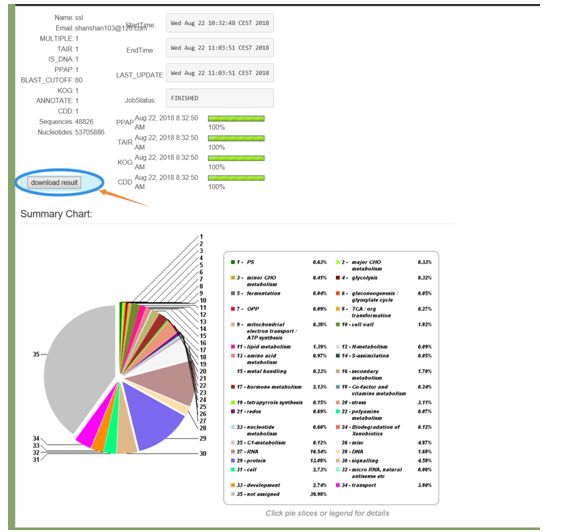
2.5注释结束,点击download result则可下载注释文件;
第五步:将选择好的mapping文件或者注释好的文件直接导入到 mapping文件中
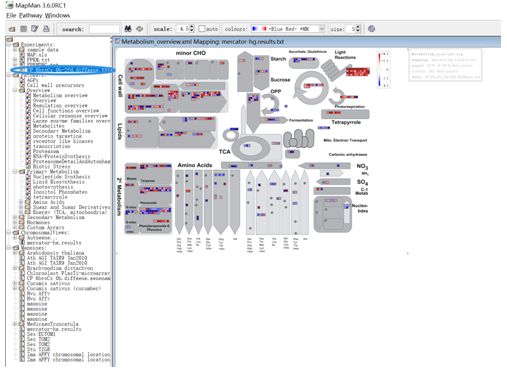
第六步:选择感兴趣的通路
点击导入的数据,即可将差异基因展示到通路图中,上调为红色,下调为蓝色,后续选择导出图片即可;