在原核生物中,mRNA 只占全部 RNA 的 1-5%,其余绝大部分是 ribosomal RNA,因此若要测序 mRNA 首先必须先将 mRNA 纯化出来,然而,原核生物并不像真核生物 mRNA 具有 poly A 的结构,因此,无法直接利用 oligo T 将 mRNA 纯化出来,如果拿 total RNA 进行测序,那么定序的效率一定会非常差,因为大部分的序列都来自 rRNA;如何提升原核生物 mRNA 的量,已成为 NGS 系统的重要议题,目前,提升原核生物 mRNA 的量,最主要的方式不外乎分为两大类,第一类是减少 rRNA 的量(下图 a 与 b ),图 a 的方式是利用 rRNA 的 probe 将 rRNA 去除,这也是最常用的方式,然而,去除 rRNA 的效率会与 probe 序列的设计与生物物种有关;图 b 的方式是利用针对 rRNA 作用的酵素将 rRNA 去除,主要是利用原核生物的 mRNA 5’端有三个磷酸,而此酵素无法对三个磷酸的结构作用,因此,能够有效去除 rRNA。
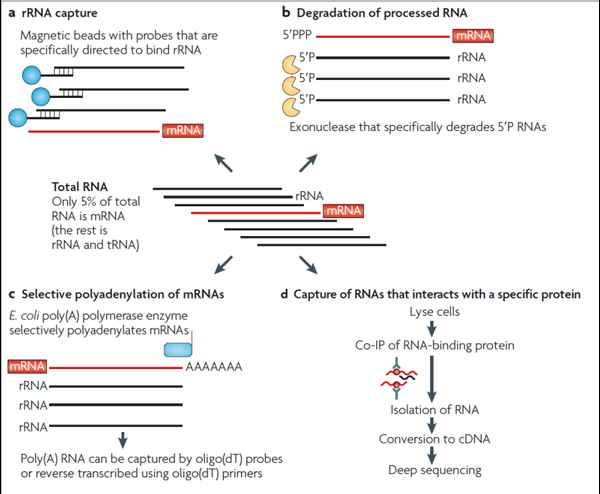
另外一大类则是将 mRNA 分离纯化出来(上图 c 与 d ),图 c 的方式是利用 E. coli 的 poly A polymerase 将 mRNA 3’端接上 poly A,再利用 oligo T 将 mRNA 纯化出来;图 d 的方式是利用 Co-IP 的方式针对 RNA binding protein 进行 IP,最后再将 mRNA 纯化出来。
以上四种方式是目前比较常见的方式,各有其优缺点,主要还是依不同研究的需求,找出最适当的方式。



1F
学习了,不过每种方法获得mRNA的比例是多少呢?这个比较直观
B1
@ tianyantammy 呵呵,这里面只是分享的实验策略,具体比例目前还没有看到相关的数据。
2F
请问怎样联系站长?