HiChIP(in situ Hi-C followed by chromatin immunoprecipitation)是一种利用原位Hi-C原理和转座酶介导构建文库来解析染色质构象的方法。该方法由Howard Chang实验室建立,相关方法论文在2016年11月份发表于 Nature Methods[1]。
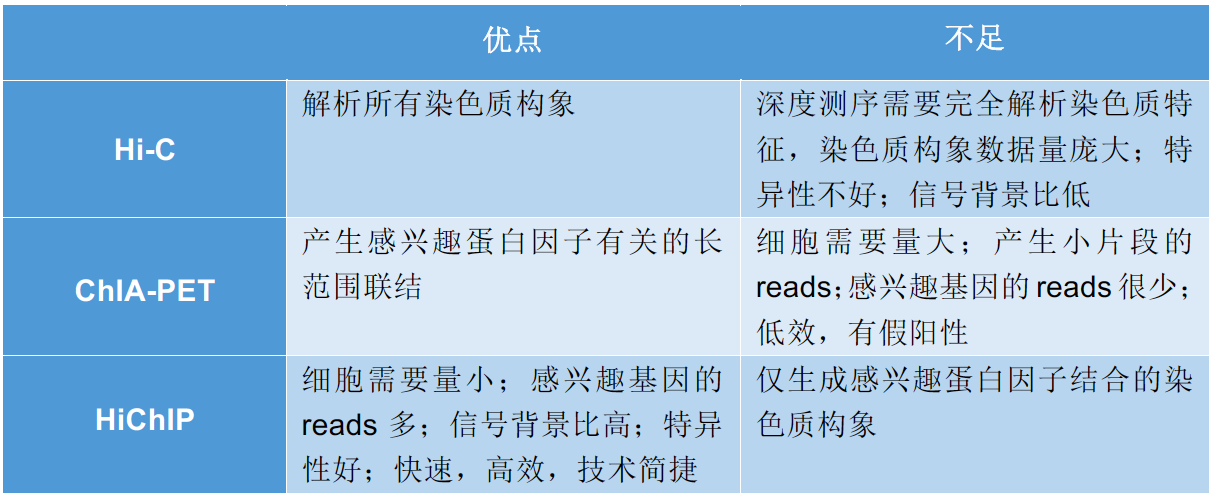
与Hi-C(Chromosome conformation capture (3C) coupled with sequencing)技术和ChIA-PET(Chromatin interaction analysis by paired-end tag sequencing)相比,Hi-C的优缺点如下:
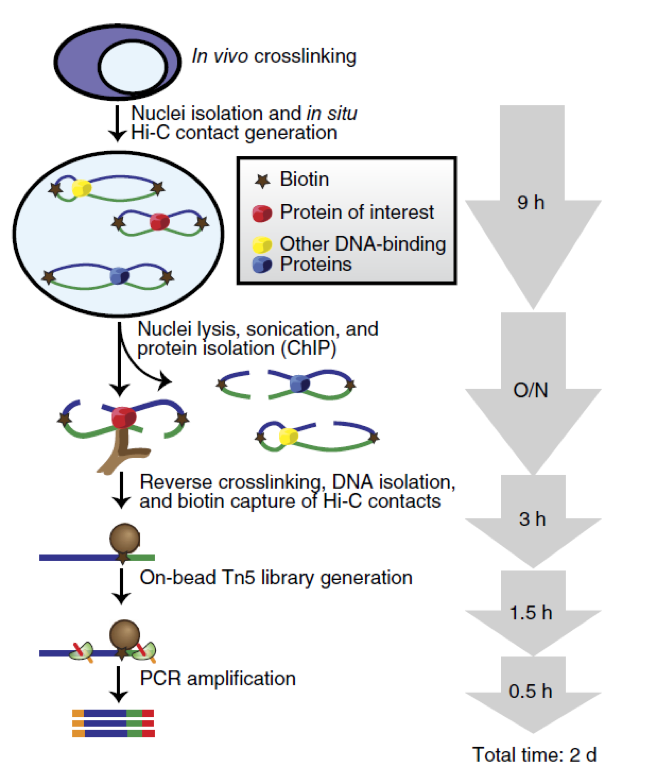
HiChIP结合了Hi-C技术和ChIA-PET技术,用更小的数据量获取更高分辨率的染色质三维结构信息。通过技术革新,表观生物在国内首次推出最新的HiChIP技术服务。HiChIP通过裂解细胞前就在细胞核中交联,从而降低假阳性,最大化提高DNA contact的捕获效率。然后收集细胞核,在原位产生Hi-C交联,利用生物素标记DNA末端。接着进行细胞核的裂解,超声打断DNA,后续用特异性的抗体进行ChIP实验。得到DNA蛋白复合物后,进行DNA洗脱和反向交联。随后,进行生物素捕获Hi-C交联和文库制备,上机测序。HiChIP的流程如图1所示。
图1 HiChIP技术流程图
技术应用
- 针对特定蛋白的染色质互作构象分析
- 转录因子作用机制研究
- 表观修饰对基因调控的机制研究
技术优势
- 构象信息读取的产量提高了10倍以上
- 相对于ChIA-PET,样本要求降低了100倍以上
- 所需细胞数量低于Hi-C
- 比Hi-C具有更大的信噪比
样本材料要求
样本类型:1. 活细胞样本,细胞数量≥1.5×107;
样本物种:仅限人、大小鼠,其他物种需评估。
其他注意事项:
- 前期准备需要固定细胞,提供细胞沉淀,通过干冰运输。
生物信息分析内容
- 数据产出统计,与参考基因组比对获得有效的reads;
- 用Fit-Hi-C得到有意义的交互矩阵;
- 统计Hi-ChIP数据富集区域 (Peak) 的信息;
- 维恩图工具分析重叠loop;
- 交互矩阵和虚拟4C可视化分析;
- 散点图和相关性的再现;
- Loop 4C热图绘制和Loop元图绘制。
参考案例:进化保守原则预测染色质的3D结构[2]
转录抑制因子CTCF也称为11-锌指蛋白或CCCTC结合因子,是一种转录因子,由CTCF基因编码。CTCF参与许多细胞过程,包括转录调节、绝缘子活性、重组和调控染色质结构等。其最主要的功能被认为是调节染色质的3D结构。CTCF环结构域、拓扑缔合结构域(TAD)和A / B区室已被确定为染色质3D结构的重要结构和功能组件,但这些特征之间的关系尚不清楚。
方法:根据进化保守原则利用高分辨的Hi-C、ChIA-PET以及HiChIP技术,获取更高分辨率的染色质3D结构。
细胞类型及数量:108 Kc167细胞和GM12878细胞
结果:转录因子结合位点是预测几种真核生物Hi-C联结的工具。A / B区室是由转录状态决定的小区域。在转录和CTCF中,环是最重要的染色质结构组成部分。在大多数真核生物中有CTCF,转录状态决定了3D染色质组织。转录活性是整个真核生物中染色质组织的主要预测子,而CTCF在哺乳动物中起着重要作用。
意义:迄今为止,在所有生物中,将基因组分离成基因大小的活性和非活性组件解释了染色质组成的结构方面。近端基因结构域共同缔合以形成进一步相互作用以形成区室的结构域。与CTCF介导的点对点相互作用一起,这些短程和长程相互作用产生TAD (Topologically associating domains, 拓扑结构域)。总之,转录状态和区室结构域之间的相关性提示了整个真核生物中染色质组织的基本和保守原理。
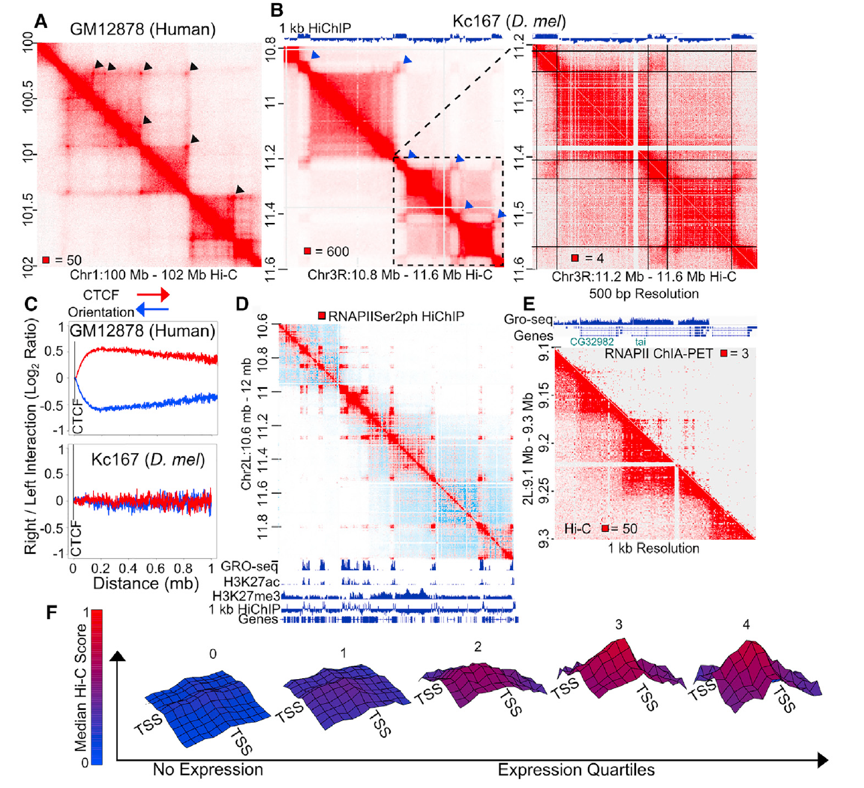
高分辨率的Hi-C contact图显示,哺乳动物存在强烈的点对点相互作用,表现为Hi-C热图中的亮点,对应于接触结构域角处的CTCF loop (A)。果蝇Hi-C contact图也显示,类似点的存在似乎与域的边界之间的交互是相对应的(B)。人类右侧CTCF位点优先与染色体右侧的其它基因组序列相互作用,左侧CTCF位点与左侧相互作用,这与果蝇是相反的 (C)。进一步发现,小的活跃的区室高度富集在RNAPII Ser2ph位点 (丝氨酸磷酸化) (D)。为了确定这些结果,进行了RNAPII的ChIA-PET以及Hi-C (E)。通过GRO-seq发现,发生在A区室的基因与基因之间的交互是与表达量相关的 (F)。
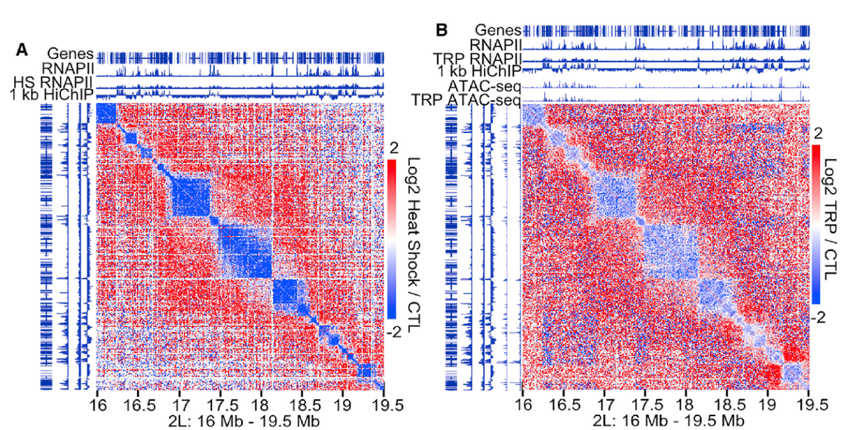
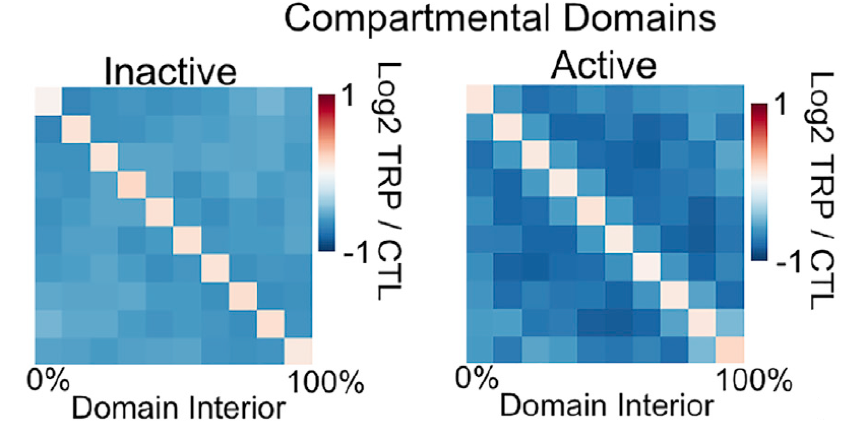
由于转录状态和结构域组织高度相关,所以测试了转录的抑制是否影响区室结构域的形成。用雷公藤内酯和热休克抑制转录,前者抑制转录起始,后者抑制整个转录。来自雷公藤内酯醇处理的细胞的10kb分辨率的Hi-C热图显示,区室结构域内的信号减小 (A)。尽管两者都导致大多数或所有基因的转录沉默,但受热休克的细胞中结构域结构的减少似乎比雷公藤内酯治疗更明显 (B)。Hi-C元图比较发现,活跃结构域比失活结构域交互减少的更多 (图4)。
参考文献
[1] Mumbach MR, Rubin AJ, Flynn RA, et al. HiChIP: Efficient and sensitive analysis of protein-directed genome architecture. Nature methods. 2016;13(11):919-922. doi:10.1038/nmeth.3999.
[2] Rowley M J, Nichols M H, Lyu X, et al. Evolutionarily conserved principles predict 3D chromatin organization[J]. Molecular cell, 2017, 67(5): 837-852. e7.