一、什么是单细胞测序?
如果简单地说,单细胞测序就是获取单个细胞遗传信息的测序技术,似乎没有多大的帮助。为了理解这个问题,咱们不妨先来了解一下测序技术到底可以做些什么。
目前,测序可以回答以下6类问题:
1. DNA的序列:ATCG怎么排列,以及各序列的丰度;
2. DNA的表观遗传修饰:比如甲基化、羟甲基化,以及组蛋白的各种修饰;
3. RNA的序列:AUCG怎么排列,以及各序列的丰度;
4. RNA的表观遗传修饰:比如近年很火的m6A修饰;
5. 染色质的结构:3C、4C、5C等各种C;
6. 其他魔性应用:比如DNA损伤位置、蛋白-蛋白相互作用等。
单细胞测序,就是想办法在单细胞层面去回答以上6类问题。
二、为什么要使用单细胞测序?
如果把这个问题换个姿势来问,那就变成,为什么非用单细胞测序不可?
世界上没有两片相同的叶子。对于多细胞生物来说,细胞与细胞之间是有差异的。当然了,这个差异可大可小。
比如说,受精卵从一个细胞开始分裂,并逐渐形成囊胚,最终发育成个体的时候,细胞与细胞之间的差异会越来越大:有的分化成神经元,有的分化成骨骼肌,各自表达着不同的遗传信息,承担着不同的生理功能。
又比如在肿瘤组织中,肿块中心的细胞,肿块周围的细胞,淋巴转移灶的细胞,以及远端转移的细胞,其基因组和转录组等遗传信息,是存在差异的。而这种差异,在临床上,可以决定该肿瘤对某种疗法是否有效。
这就是所谓的遗传信息的异质性。
传统的研究方法,是在多细胞水平进行的。因此,最终得到的信号值,其实是多个细胞的平均,丢失了异质性的信息。为了让大家能够更加直观地理解这个问题,我们不妨来看下面这张图:
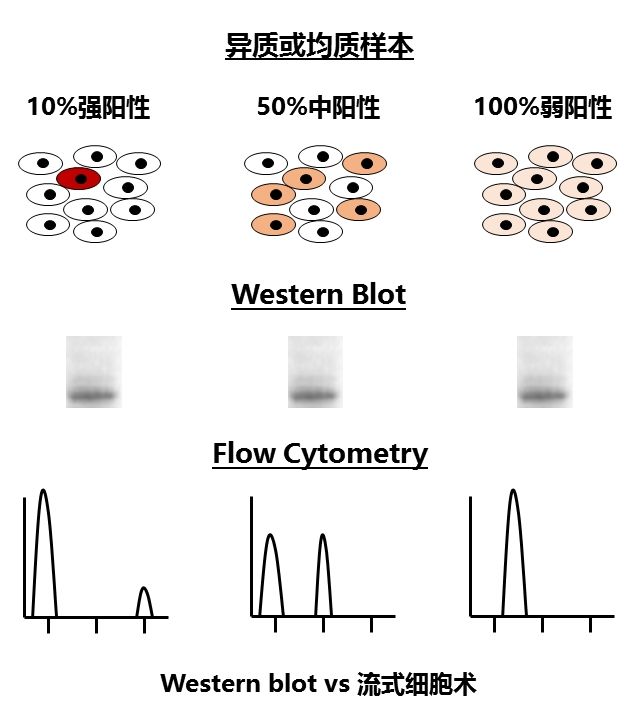
为了检测某个蛋白质的表达量,我们可以用Western blot和流式细胞术来实现。但是,用Western blot的话,我们并没有办法区分上述的情况:目的蛋白只在10%的细胞中强表达,还是在50%的细胞里中等表达,还是在所有细胞中弱表达呢?因为最终电泳跑出来,就是一条差不多强度的带。但如果用流式细胞术这种在单细胞水平对荧光强度加以测定的技术,就能区分上述的情况了。
同样道理,单细胞测序能够检出混杂样品测序所无法得到的异质性信息。而这将带领整个遗传学领域进入新的次元。
三、如何实现单细胞测序?
目前主要有两种策略来实现单细胞测序。
第一种,也就是目前大多数人所想象的那样,将单个细胞分离出来,并独立构建测序文库,最终进行测序的路线。我们可以通过流式细胞术(含微流体芯片),或者激光捕获显微切割(LCM)来实现。流式细胞术估计大家比较熟悉,就不多讲了,它主要运用于细胞样品。对于组织切片样品来说,主要是通过LCM来获取单细胞,原理可以见下面的示意图。
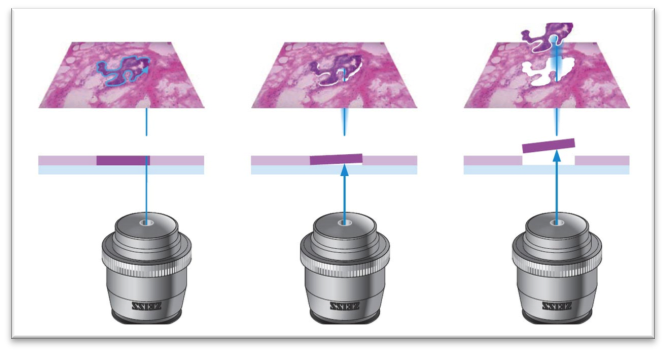
不过,将单细胞挨个分离出来再分别建库测序,通量非常低,这主要受成本的限制。随着待测单细胞的个数的增长,测序的成本也会几乎呈线性提升。通常做十几二十来个细胞,就要烧掉很多钱了。然而,这数十个细胞,就足够说明问题了吗?
为了克服这个困难,近年来多采取第二种策略:基于标签(barcode)的单细胞识别。它的主要思想是,给每个细胞加上独一无二的DNA序列,这样在测序的时候,就把携带相同barcode的序列视为来自同一个细胞了。这种策略,可以通过一次建库,测得数百上千个单细胞的信息。
不过,针对具体的测序类型,给细胞加barcode的方案是有不小的区别的。对于RNA(转录组mRNA)来说,会比较容易理解一些。由于mRNA测序前需要做逆转录,那么我们只需要在poly T引物的5’端加入barcode即可。具体可见下面的示意图(来自文献doi:10.1038/nprot.2016.154):
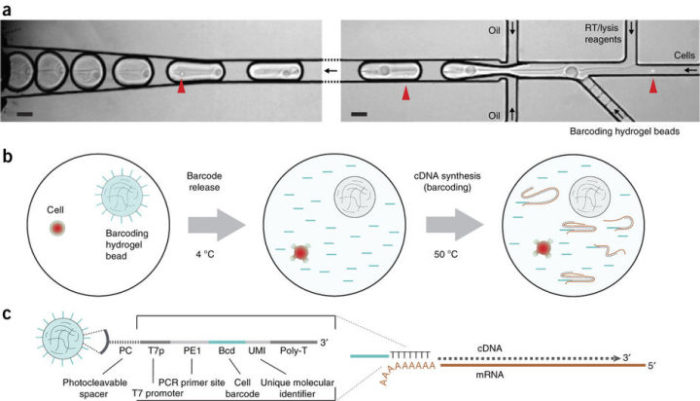
首先将单细胞悬液样品和带有barcode的水凝胶珠子,通过微流体芯片,包裹在一个油滴之中。在油滴中进行逆转录之后,每一个单细胞的cDNA文库,就带上了独一无二的barcode了(蓝色部分)。最后,我们再将所有的单细胞cDNA文库混在一起测序,再通过程序识别barcode,区分单细胞。
如果测序对象是DNA,比如全基因组,就需要用别的方式来加barcode。目前主要是通过一种经过改造的高效转座酶(transposase)Tn5来实现。
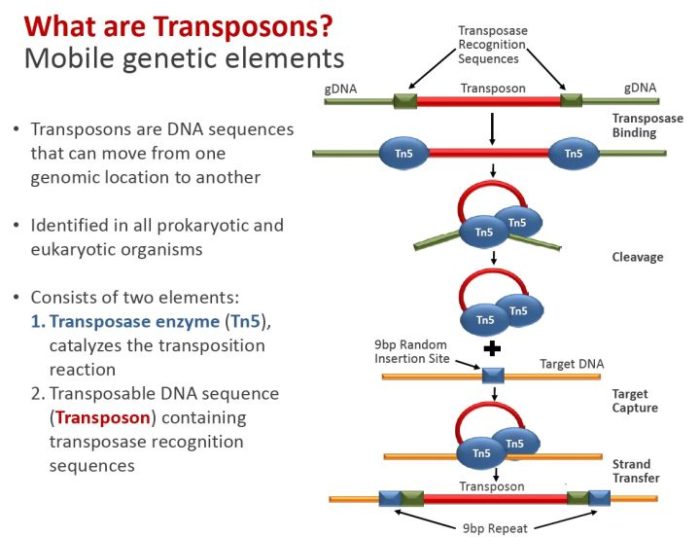
基因转座是指转座子DNA从一个染色体座位“跳跃”到另外一个座位的过程。在这个过程中,有转座酶的参与。单细胞的DNA测序就利用了这个特性,将barcode DNA预先和转座酶Tn5组装好,再通过上述的微流体技术,将细胞和转座复合物包裹在一个油滴之中。随后,转座酶会把barcode插入到基因组DNA之中。这个过程在文献中也被成为tagmentation。
不过,基于Tn5的barcode复杂度(即能有多少独一无二的barcode)还是比较有限的。为了保证tagmentation的效率,上图中红色的barcode区域不可以过长。同时,为了避免测序错误带来的误识别(如偶尔测错了一个碱基,但却被当成另外一个barcode),barcode的复杂度也不是4的n次方那么高,需要引入校正机制。具体就不展开讲了。总地来说,仅靠Tn5来做单细胞,一次往往仅能识别数十到数百个单细胞。
为了提高复杂度,即一次能够捕获的单细胞数目,目前的解决方案是走组合索引(combinatorial indexing)路线。(见下图,来自文献doi:10.1038/nmeth.4154)
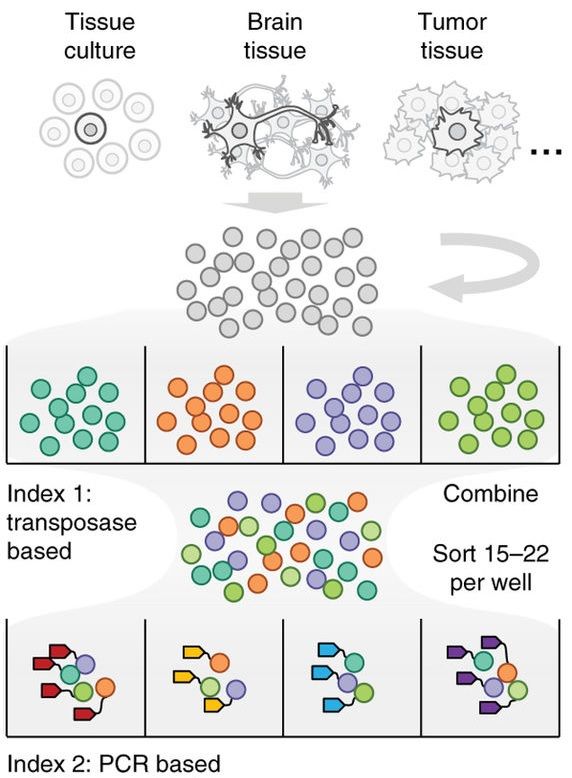
它的主要思路是,通过两步反应,加两次标签。首先,将单细胞悬液放在多孔板中,并用转座酶Tn5给细胞加第一个barcode,这里每个孔中的barcode是不同的。然后,再将样品混合起来,通过流式细胞术,将少量的细胞分选到含有建库PCR引物的多孔板中。而这些引物是带有第二轮barcode的。因此,经过Tn5的转座,和PCR加标签,绝大部分的细胞就能带上独一无二的barcode了。
读到这里,肯定有人发现这个方案存在的问题。举个例子,万一在流式分选时,在第一个孔里分了两个或以上橙色细胞,然后又通过PCR被加上了红色的标签,那这两个单细胞就无法被区分开来了。
确实如此,combinatorial indexing大概会有10%的撞车率(collision rate),即约有10%的机会把两个单细胞被误认为是同一个。这个数值的高低,取决于第一步tagmentation的复杂度(复杂度越高,撞车率越低),以及在分选时,分到每一个孔里的细胞数量(数量越低,撞车率越低)。但是,combinatorial indexing却能一次识别数千个单细胞,将通量提升数十至上百倍。鱼与熊掌,就看实验者的取舍了。



1F
您好,请问可以转载吗?
B1
@ aasdf 可以