主成份分析方法可以对基因芯片的样本聚类情况进行可视化,可获得样本在实验组和对照组之间的直观分布情况,从而便于对异常样本进行检测和去除,否则异常样本的存在将会对差异基因的鉴定等后续分析造成不利影响。下面我将演示在R语言中如何对芯片样本进行PCA分析及可视化。
首先,需要下载R语言的程序,R是一个很好的免费统计语言,官方下载网址为:www.r-project.org/
为了便于演示,我首先用在R中建立一个模拟的芯片数据矩阵,该矩阵为10000行(10000个基因),30列(30个样本):
- chip.dat<-matrix(rnorm(10000*30,mean=0),ncol=30,nrow=10000)
我把30个样本分为两组,前15列和后15列各为一组,给它们定义不同的颜色:
- colour<-c(rep(2,15),rep(3,15))
在10000个基因中,我们假定有100个基因在两个组间是有差异的,我们假设其中有50个在前一组是上调的,另50个在前一组中是下调的:
- diff.ind<-sample(1:10000,100)
- chip.dat[diff.ind[1:50],1:15]<-rnorm(50*15,mean=2)
- chip.dat[diff.ind[51:100],1:15]<-rnorm(50*15,mean=-2)
如此,我们就构造好了这个模拟数据,当然,这个数据中两组间的样本是没有问题的,也就是说没有异常值的,下面我们可以对其进行PCA分析:
- chip.dat.pca<-princomp(chip.dat)
然后我们可以对前三个主成份进行可视化,看看这30个样本在前三个主成份的空间中的分布,这需要用到rgl包中的plot3d的函数:
- library(rgl)
- plot3d(chip.dat.pca$loadings[,1:3],col=colour,type="s",radius=0.025)
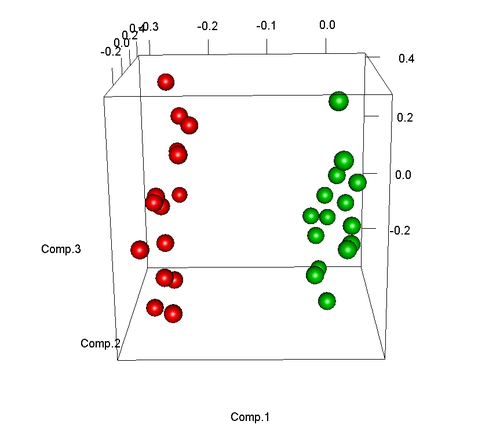
这样我们就可以得到了三维的样本分布图如下:
但是,如果样本中有异常样本,那么PCA也可以很好的发现,下面将会模拟样本15为异常样本(如实验未成功而其实因该属于对照组):
- chip.dat<-matrix(rnorm(10000*30,mean=0),ncol=30,nrow=10000)
- chip.dat[diff.ind[1:50],1:14]<-rnorm(50*14,mean=2)
- chip.dat[diff.ind[51:100],1:14]<-rnorm(50*14,mean=-2)
对该数据进行PCA分析及可视化:
- chip.dat.pca<-princomp(chip.dat)
- plot3d(chip.dat.pca$loadings[,1:3],col=colour,type="s",radius=0.025)
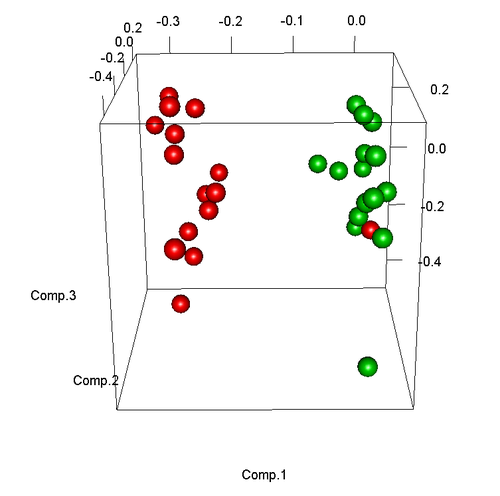
出现下图:
从图中即可清晰的看出有一个样本(红色)是异常的,出现在了绿色组中,在后续分析时应予以去除。
From:http://blog.sina.com.cn/u/1944457820