利用转录组测序如何研究选择性剪接规律?
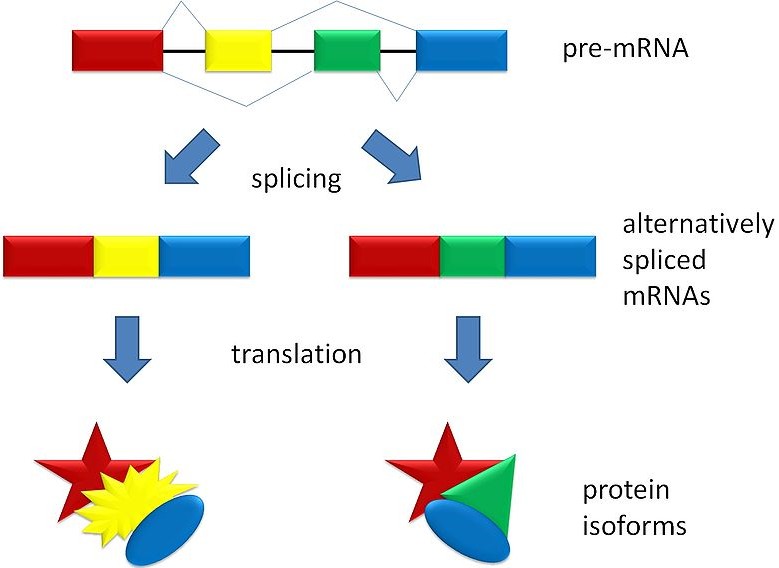
答:在真核生物中,选择性剪接现象普遍存在。基因转录形成的mRNA前体(pre-mRNA)在剪接过程中因去掉不同的内含子区域或保留不同的外显子区域,可形成不同的剪接异构体. 根据RNA-seq原理,只要测序深度足够深,就能检测到所有转录本的全部序列,包括来自剪接接合区的序列。利用考虑到接合区的reads定位方法,就有可能系统地研究某一组织或某一条件下的基因选择性剪接事件。
Tophat等软件定位剪接接合区reads的策略能标定出剪接事件中的两个剪接位点:供体位点和受体位点. 通过比较供体位点和受体位点的组合,就能识别选择性剪接事件。进一步,通过对供体和受体位点的reads计数,结合外显子其他区域的reads数据,还能定量地计算选择性剪接事件之间的比例。
对于每一个剪接异构体,RNA-seq数据能在一定程度上推断其表达水平。比如,可以根据已知外显子组成和各外显子长度对剪接异构体建立数学模型,在测序reads在转录本上均匀分布的假设下,利用各外显子上的reads数和接合区reads数求解异构体的表达值。软件IsoInfer和cufflinks都采用了这种思路来实现剪接异构体的表达推断。



1F
外显子延长
外显子遗漏
内含子保留
可变剪接
互斥剪接
Cell 126:37 (2006)
Nature Reviews: Genetics 5:773 (2004)