数据来自2018年9月的NC文章Acquired cancer resistance to combination immunotherapy from transcriptional loss of class I HLA
实验设计
共有两名患者:
- 患者2586-4:
- The primary patient (2586-4) received hypofractionated radiation for HLA upregulation to some but not all disease sites
- 利用10X 3' Chromium v2.0平台建库 + Hiseq2500 "rapid run"模式 GSE117988
- discovery tumor部分:After sequence alignment and filtering, 7431 tumor cells (2243 cells before and 5188 cells after T cell therapy)
- discovery PBMC部分:After sequence alignment and filtering, a total of 12,874 cells were analyzed [其中包含了四个时间点:治疗前(Pre),治疗后早期day +27(Early),治疗后反应期day+37(Resp),治疗后复发+614 (AR)]
ID Description GSM3330559 Tumor Disc Pre GSM3330560 Tumor Disc AR GSM3330561 PBMC Pre GSM3330562 PBMC Disc Early GSM3330563 PBMC Disc Resp GSM3330564 PBMC Disc AR - 患者9245-3:
- The second validation patient (9245-3) is a 59-year-old man with metastatic MCC that had initially presented as stage IIIB disease, now metastatic at multiple sites
- 利用10X 5' V(D)J 进行cell washing, barcoding and library prep+ NovaSeq 6000(gene expression) + Hiseq4000 (V(D)J) GSE118056
ID Description GSM3317833 PBMC Relapse - L001 GSM3317834 PBMC Relapse - L002 GSM3317835 Tumor Relapse - L001 GSM3317836 Tumor Relapse - L002
软件环境
原始数据一般是以SRR格式存放,这个文件一般都要几个G,于是下载器首选ascp,但是直接使用ascp下载又需要配置一些参数,对于新手来说,最好是能提供一个ID,然后直接就下载,这个就需要用到prefetch 与 ascp的组合了
prefetch是sratools中的一个小工具,因此直接用conda下载就好
- conda install -c daler sratoolkit
- prefetch -h # 可以显示帮助文档就说明安装成功
- # 如果要下载数据比如SRR文件,直接加ID号,指定输出目录就好
- prefetch SRRxxxxxxx -O PATH
默认情况下,prefetch是利用https方式去下载原始数据,这个就像直接从网页下载一样,速度有一定的限制。因此我们需要先安装一款叫做"aspera"的下载工具,它是IBM旗下的商业高速文件传输软件,与NCBI和EBI有协作合同
- wget http://download.asperasoft.com/download/sw/connect/3.7.4/aspera-connect-3.7.4.147727-linux-64.tar.gz
- tar zxvf aspera-connect-3.7.4.147727-linux-64.tar.gz
- #安装
- bash aspera-connect-3.7.4.147727-linux-64.sh
- # 然后cd到根目录下看看是不是存在了.aspera文件夹,有的话表示安装成功
- cd && ls -a
- # 将aspera软件加入环境变量,并激活
- echo 'export PATH=~/.aspera/connect/bin:$PATH' >> ~/.bashrc
- source ~/.bashrc
- # 最后检查ascp是不是能用了
- ascp --help
ascp安装成功后,prefetch就会默认将下载方式从https转移到fasp,说明开启加速模式
一般ascp没有什么问题,出问题主要是:
- ascp: Failed to open TCP connection for SSH, exiting.
- Session Stop (Error: Failed to open TCP connection for SSH)
- # 官网给出的解决办法是:https://support.asperasoft.com/hc/en-us/articles/216126918-Error-44-UDP-session-initiation-fatal-error
- On many Linux systems the default firewall can be configured with iptables. You will have to allow all incoming and outgoing traffic on UDP port 33001 (or whatever your Aspera UDP port is), which you can do with the following commands:
- # 使用下面这两个命令(但需要管理员权限)
- # iptables -I INPUT -p tcp --dport 33001 -j ACCEPT
- # iptables -I OUTPUT -p tcp --dport 33001 -j ACCEPT
数据下载
以患者2586-4为例,所有数据都存放在GEO中
- 打开https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE117988
(这里注意链接是有规律的,只需要改变最后的ID号就能获取其他的GEO数据) - 点击SRA这里的
SRP155988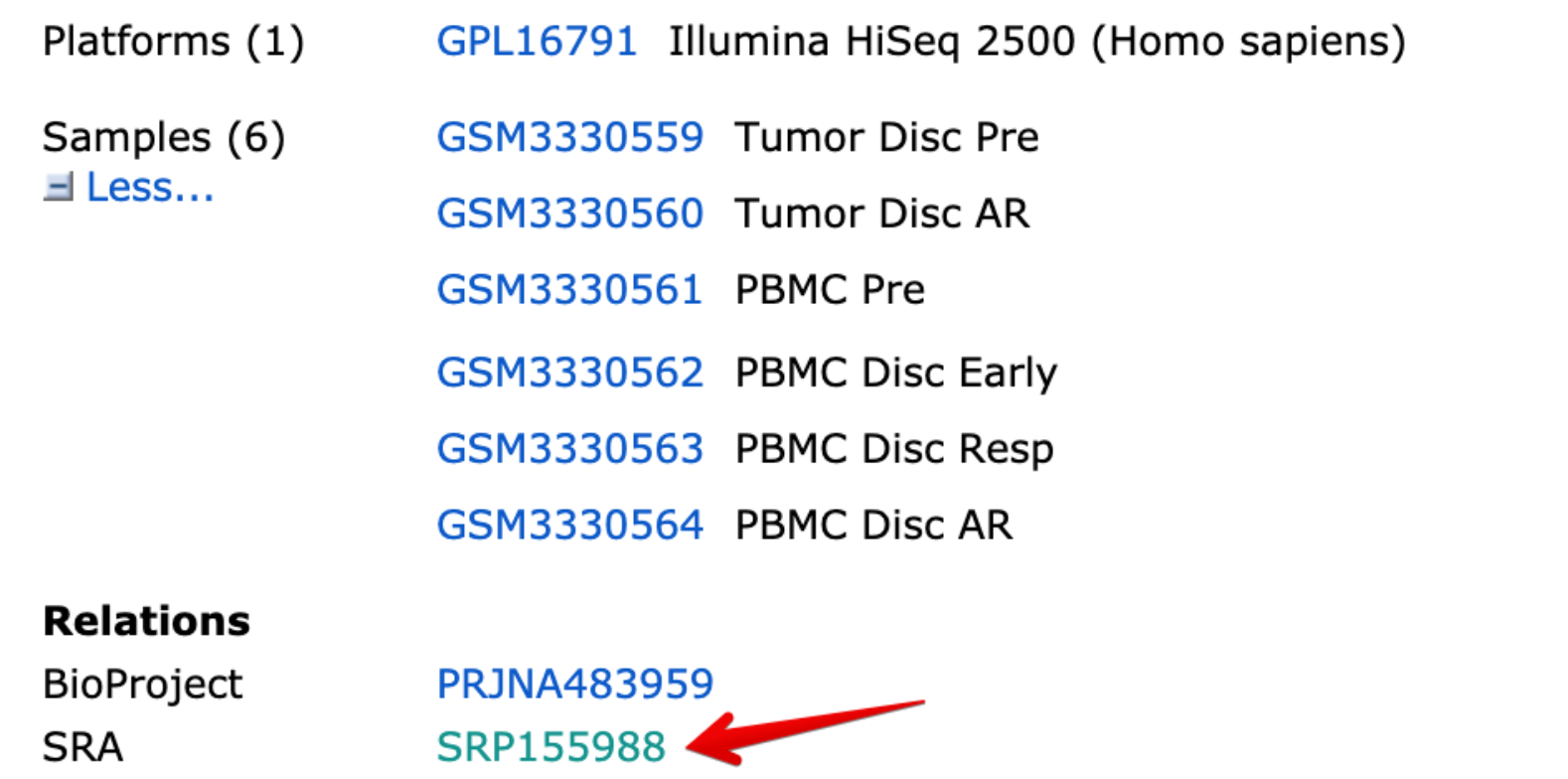
send to=>Run Selector=>Go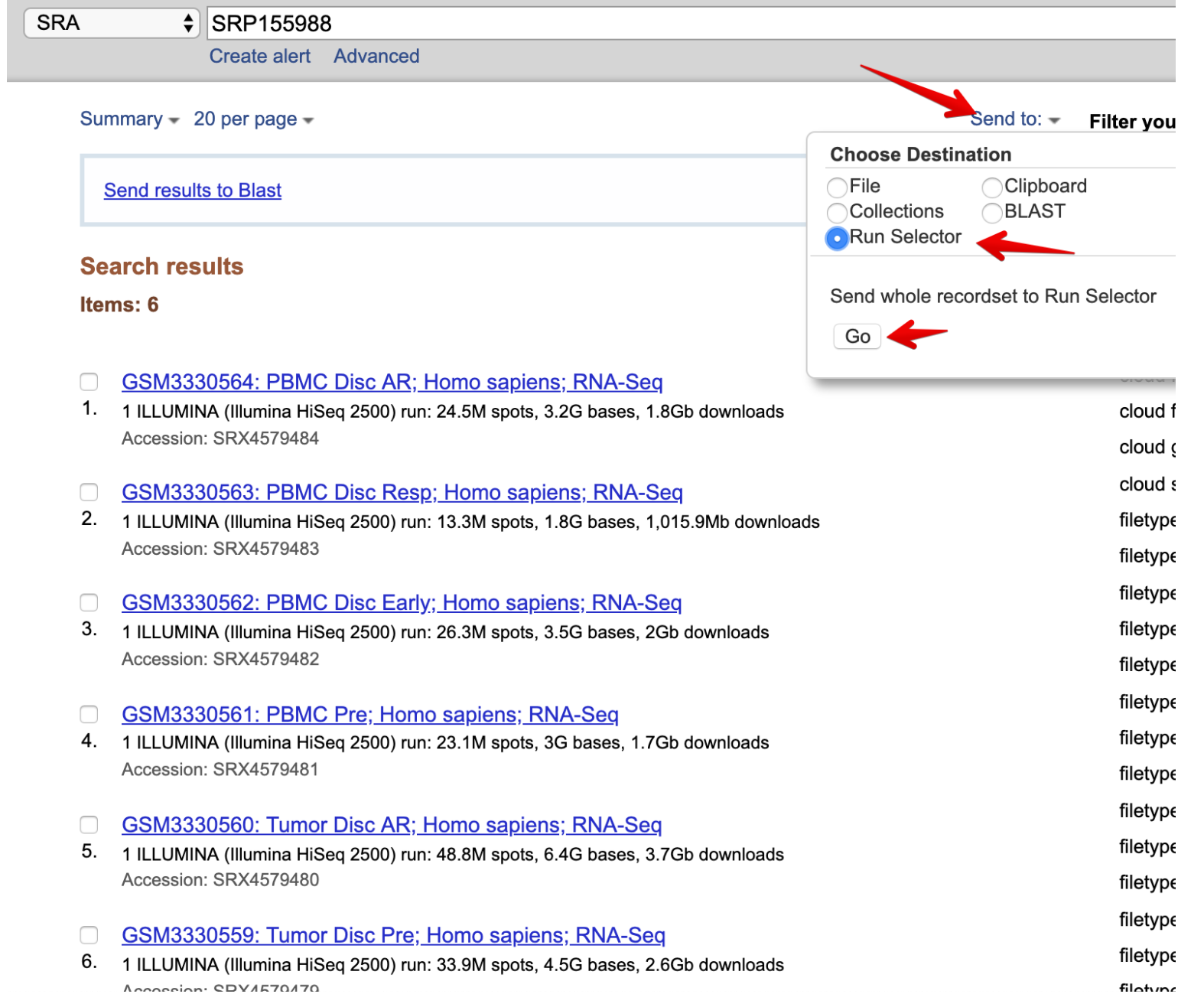
- 下载Accession List,然后就得到了一个文本文件,列出了6个SRR ID号
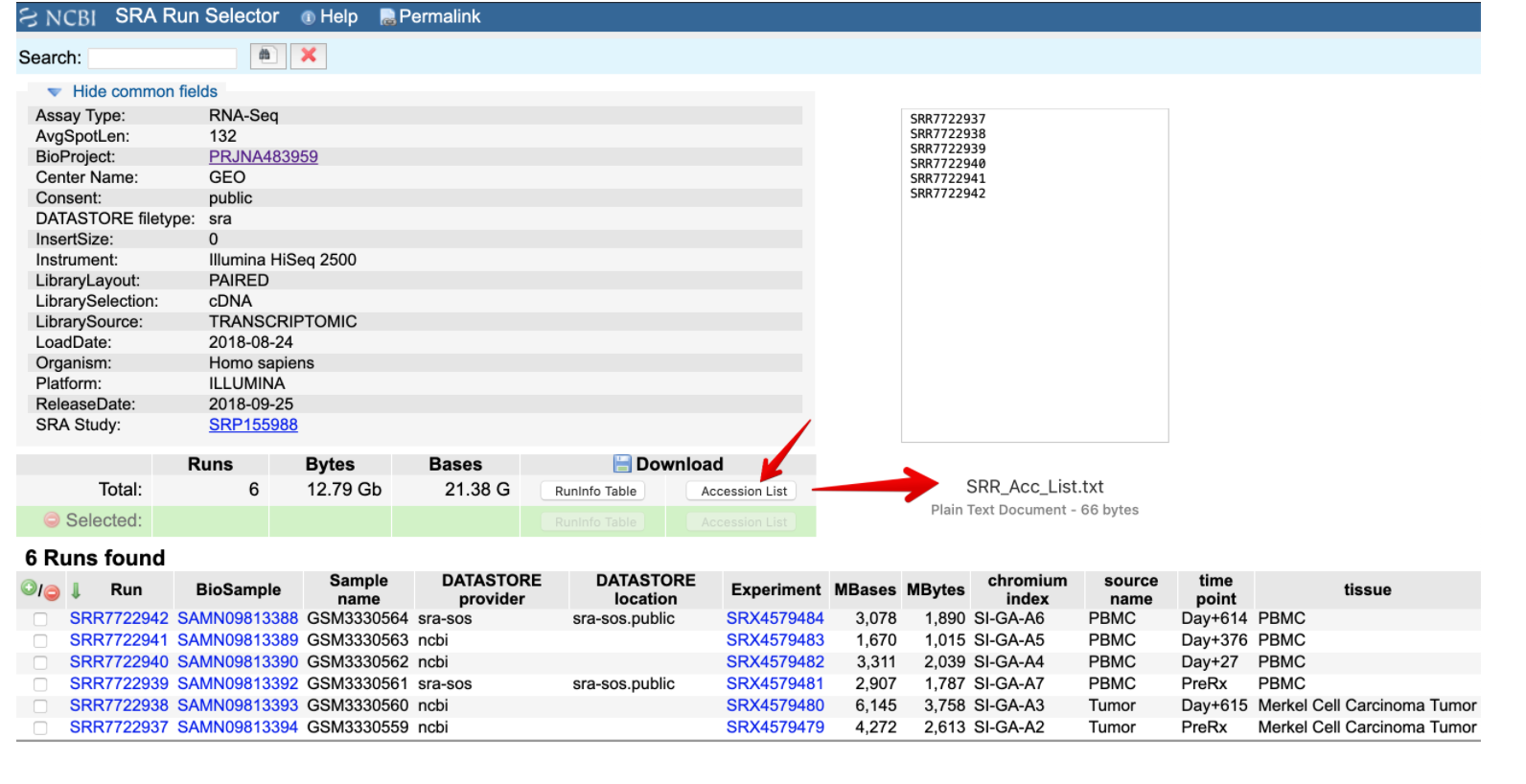
- 下载代码
- wkd=/home/project/single-cell/MCC
- cd $wkd/raw
- # for patient 2586-4
- cat >SRR_Acc_List-2586-4.txt
- SRR7722937
- SRR7722938
- SRR7722939
- SRR7722940
- SRR7722941
- SRR7722942
- cat SRR_Acc_List-2586-4.txt |while read i
- do prefetch $i -O `pwd` && echo "** ${i}.sra done **"
- done
- # 一般2.6G文件下载2分钟左右
- 下载成功会有提示
- 2019-xxxxxxxx prefetch.2.9.1: fasp download succeed
- 2019-xxxxxxxx prefetch.2.9.1: 1) 'SRR7722937' was downloaded successfully
- 2019-xxxxxxxx prefetch.2.9.1: 'SRR7722937' has 0 unresolved dependencies
- ** SRR7722937.sra done **
两个患者的十个样本数据下载结束后发现,SRR7722939和SRR7722942下载失败,看了一下数据源,这两个数据在sra-sos.public这个位置,而不是在ncbi
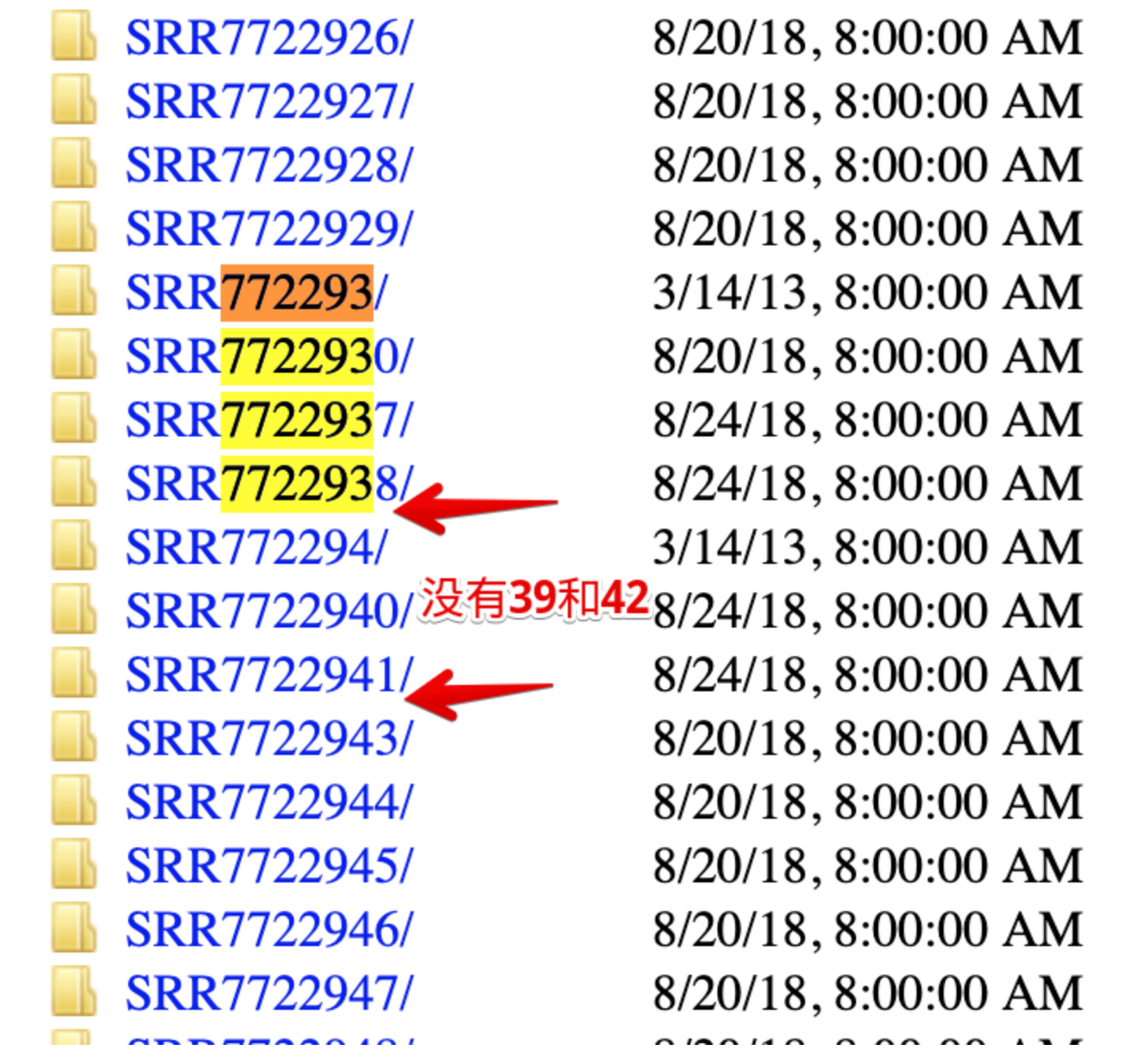
于是,可以选择另一个途径EBI下载
- 进入官网https://www.ebi.ac.uk/ena ,搜索想下载的SRA号
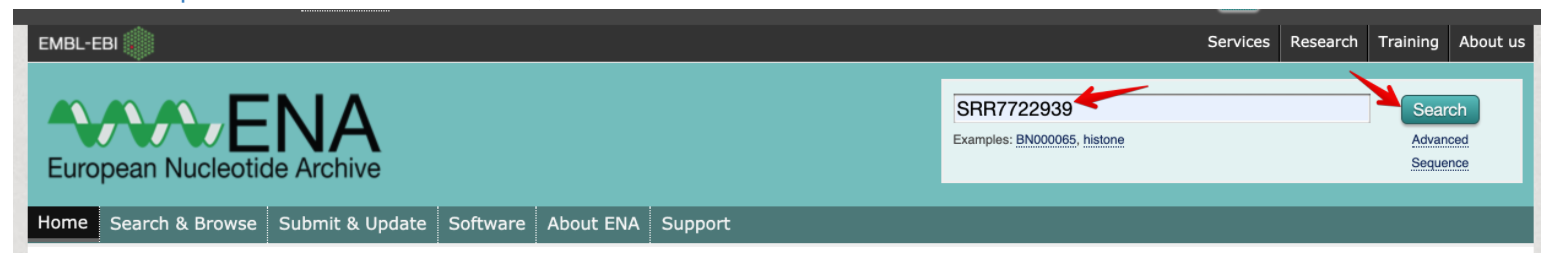
- 选择SRR这里[或者直接通过https://www.ebi.ac.uk/ena/data/view/SRR7722939修改ID]
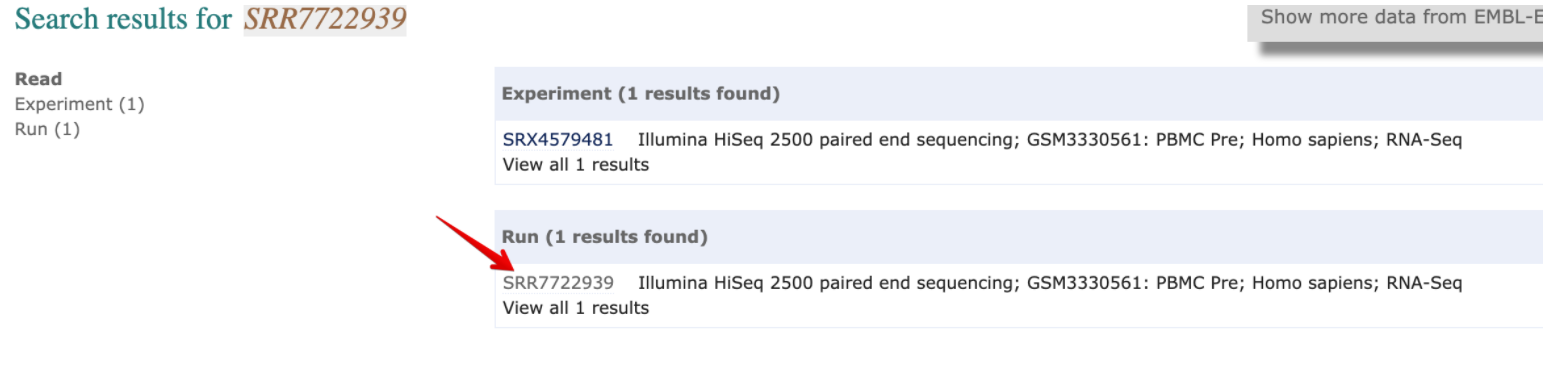
- EBI有个好处就是可以直接下载fastq格式文件(左边方框),如果要下载sra就复制右边红色方框中链接
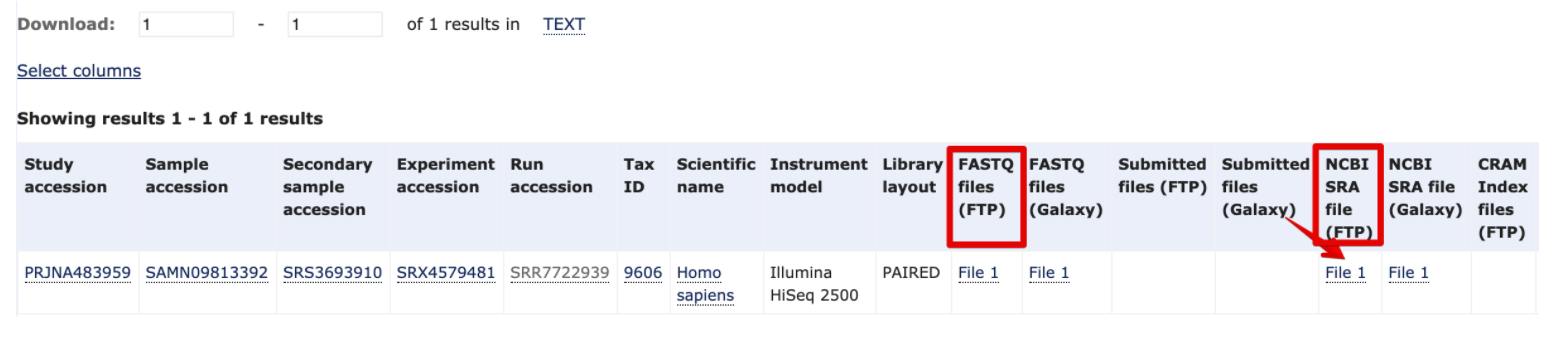
- 然后利用这个代码下载
- ascp -QT -l 300m -P33001 -i ~/.aspera/connect/etc/asperaweb_id_dsa.openssh era-fasp@fasp.sra.ebi.ac.uk:vol1/srr/SRR772/009/SRR7722939 ./


