01、PROGgeneV2
http://genomics.jefferson.edu/proggene/
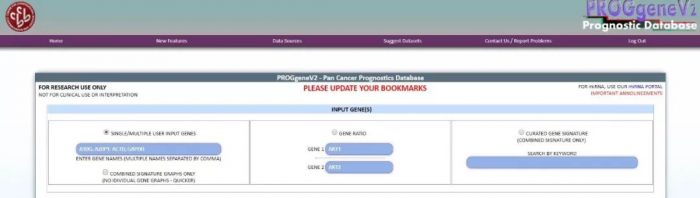
PROGgeneV2可以分析乳腺、结直肠、卵巢、皮肤、眼、胸腺、肾上腺、胰腺、胃、甲状腺、膀胱、前列腺、尿道、骨、肝、宫颈、神经内分泌系统、肺、脑、肾等器官系统各恶性肿瘤的预后,覆盖度非常广泛
分析类型可选择复发、转移、死亡、脑转移、肺转移等

可选择基因表达量的中位、25%、75%和平均值为界定义高表达和低表达
该网站还可以整合多个基因,即gene expression signature,即联合多基因的表达来分析预后,功能十分强大!但有个缺点,只有mRNA,无lncRNA,microRNA的数据。
02、lnCAR
https://lncar.renlab.org/

一款专门做lncRNA差异表达、kegg、共表达网络和生存分析的数据库,由中山大学任间教授开发,目前共收录了10种肿瘤的数据 ,膀胱癌、乳腺癌、宫颈癌、结直肠癌、食管癌、胃癌、肝癌、肺癌、卵巢癌、前列腺癌的lncRNA数据。
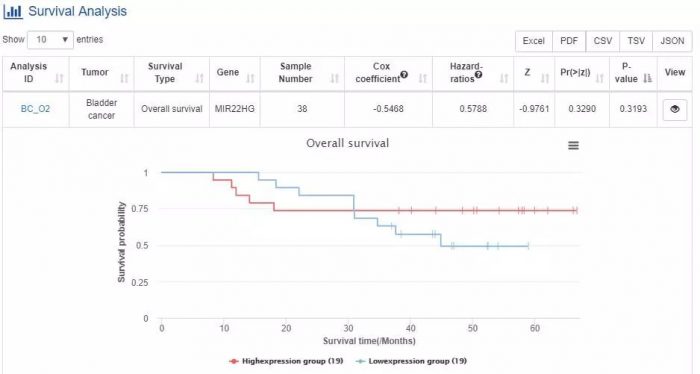
03、GEPIA
http://gepia.cancer-pku.cn/index.html

一款TCGA数据库的可视化在线网站,TCGA中包含的癌种GEPIA几乎都有,可分析mRNA、lncRNA、miRNA的数据,覆盖面广,大大方便了我们对TCGA数据库的挖掘
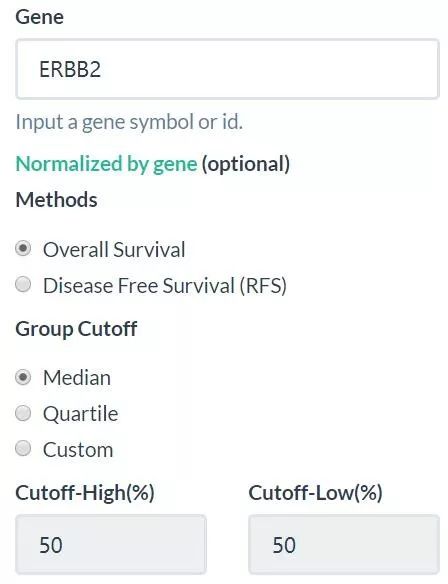
生存分析可选择总生存期和无病生存期,还可自由选择cutoff值(即定义高表达和低表达),不同cutoff值得到的结果可能不同。
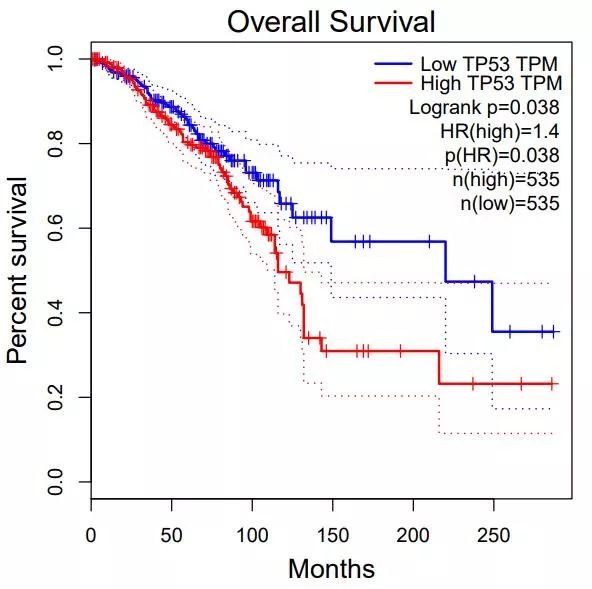
04、UALCAN
http://ualcan.path.uab.edu/analysis.html
UALCAN数据库的生存分析也是基于TCGA数据库,数据包括mRNA和部分lncRNA

点击survival,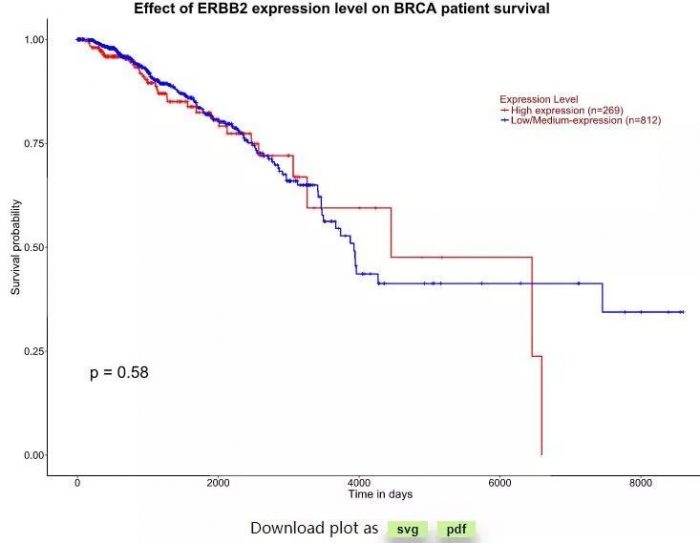
05、KM plotter
http://kmplot.com/analysis/index.php?p=background
因为有个auto select best cufoff选项(对cutoff值进行最优化以判定高表达低表达与预后的关系),该在线网站常能得到p<0.05的阳性结果,为生信分析文章中最常用的数据库之一。
survival 选项中可选择:
OS 总生存期
RFS(recurrence-free survival) 无复发生存期
PPS(post progression survival,进展后的生存期)
DMFS(distant metastasis free survival) 无远处转移生存期
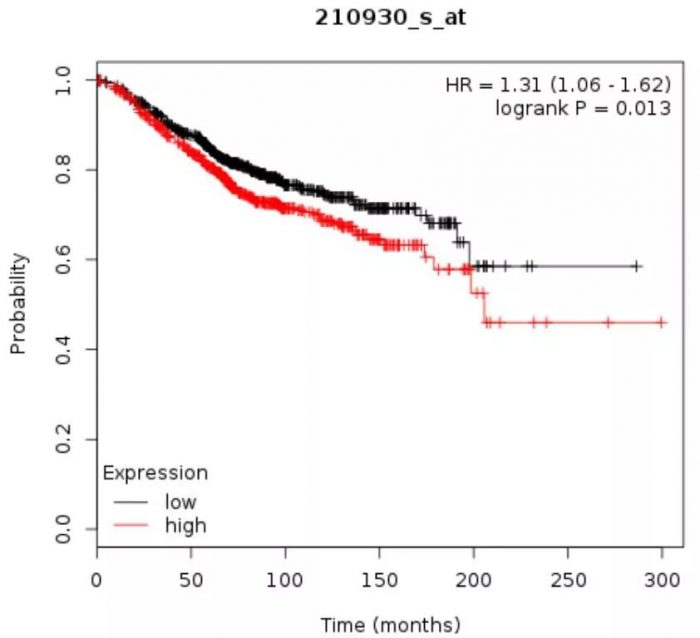
例如我们选择ERBB2后选择auto select best cufoff选项,得到的p=0.013,与前面的GEPIA和UALCAN得到的结果有所不同。
06、cbiportal
http://www.cbioportal.org/
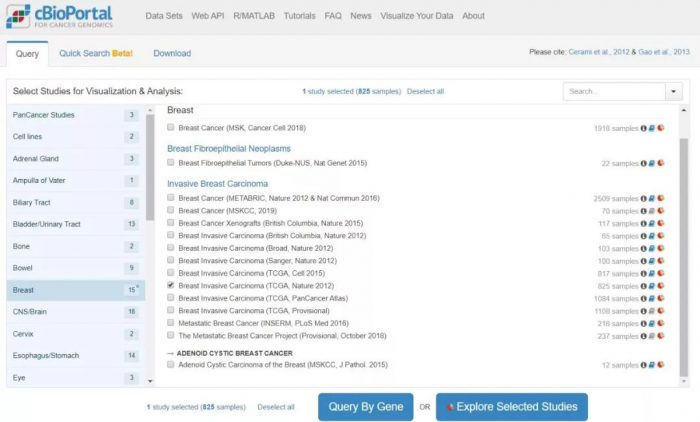
一款TCGA数据可视化的网站,并包含一部分细胞系和芯片数据,进入官网后再左侧勾选部位,在右侧选择数据集,点击query by gene,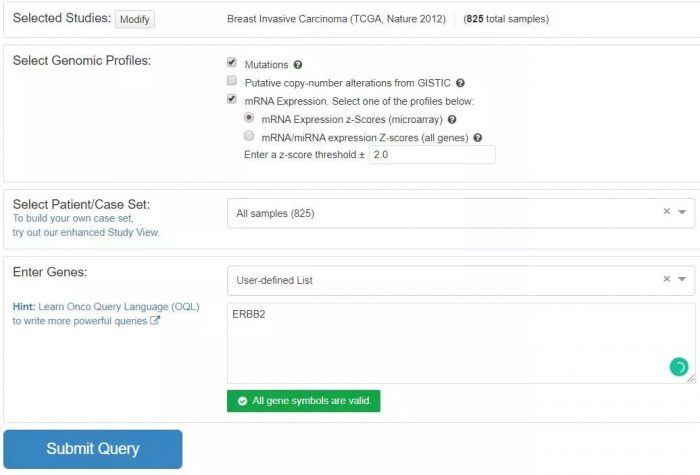
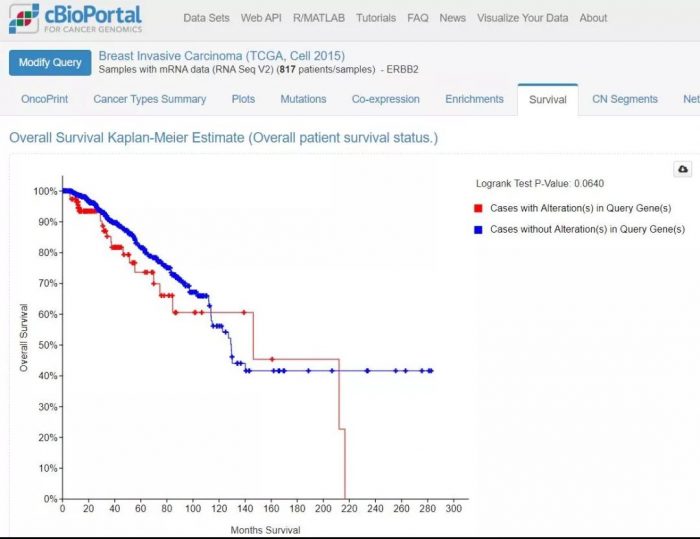
此页面可按需求选择病人亚组,这里我们选择all samples, 输入ERBB2,生存分析结果如下
07、Oncolnc
http://www.oncolnc.org/
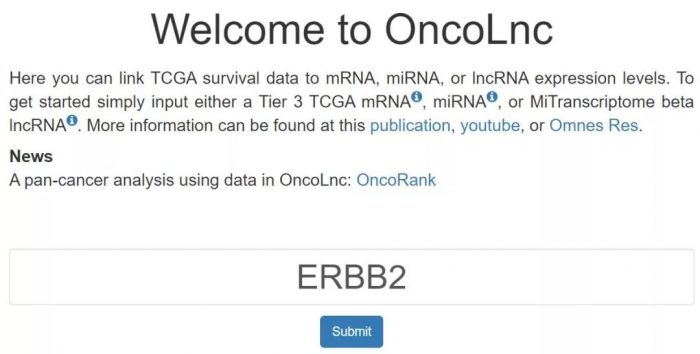
一款TCGAmiRNA/mRNA/lncRNA 生存数据分析的网站,优点是cutoff值可以自己随意设置。在首页直接输入感兴的分子
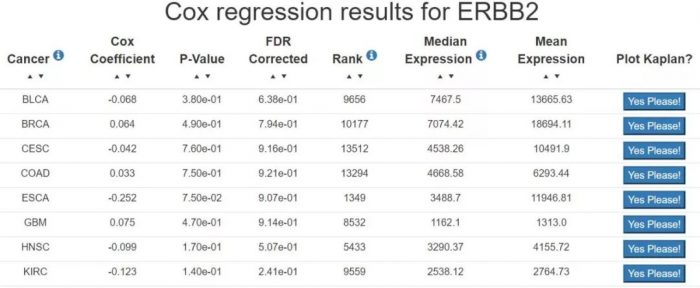
选择感兴趣的癌种,此处选择BRCA,点击Yes please

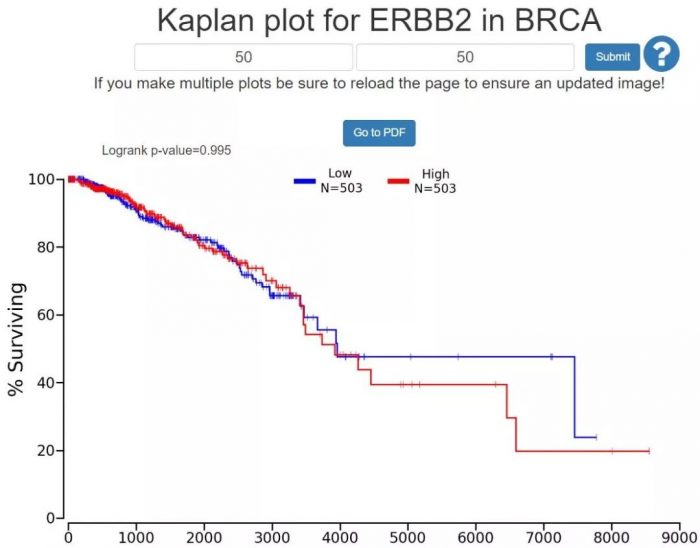
输入百分之多少定为低表达,百分之多少定为高表达,即设定cutoff值,例如输入50, 50
08、Oncomir
http://www.oncomir.org/
一款专注于miRNA生存分析的在线工具,在左侧选择custome signatures
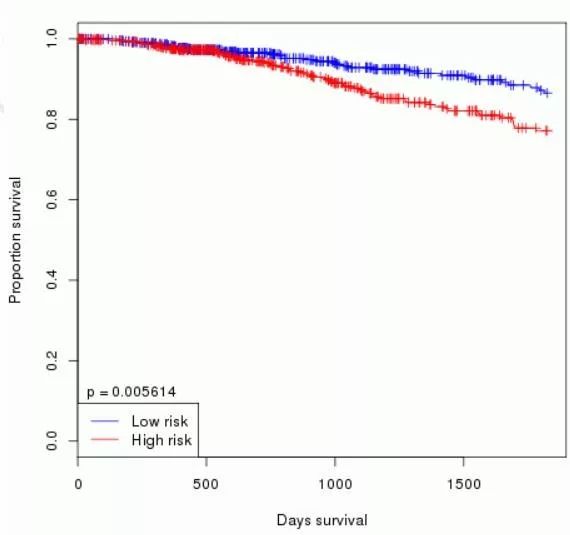
选择癌种,输入miRNA的全称,输入cutoff值,点击retrieve survival curve, 结果如下

09、Breast Cancer Gene‑ExpressionMiner v4.1
http://bcgenex.centregauducheau.fr/BC-GEM/GEM-Accueil.php?js=1
专注于分析乳腺癌数据,在首页的analysis 菜单下选择prognostic-targeted
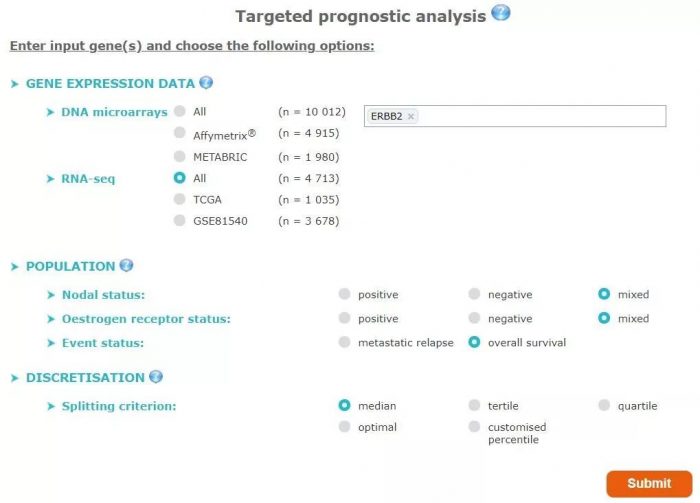
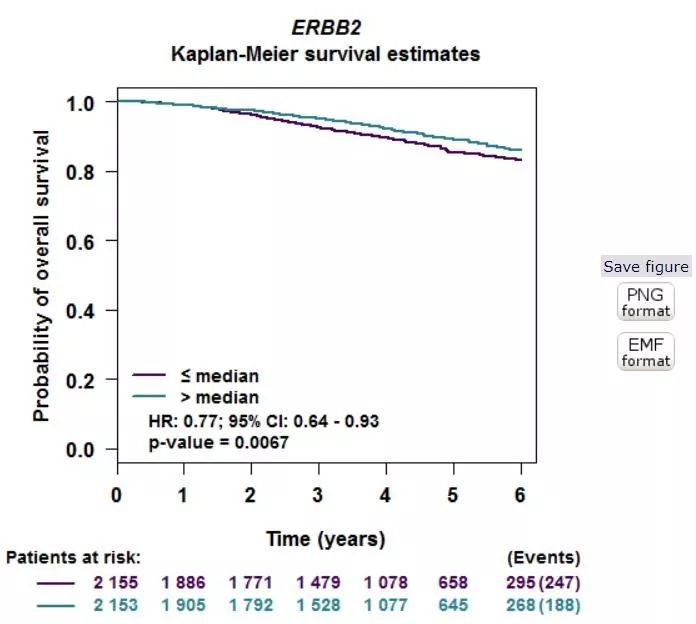
选择相应的数据来源和患者亚组后,点击submit
10、MethSurv
https://biit.cs.ut.ee/methsurv/
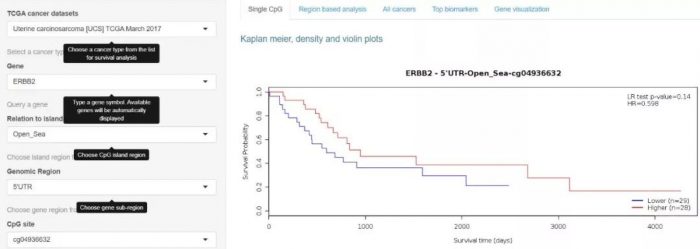
基于TCGA 甲基化数据开发的甲基化与肿瘤预后关系的在线分析网站,分析单个CpG位点与患者预后的关系
对于同一分子,这些数据库预测的结果可能不尽相同,主要原因是数据来源、样本量和cutoff值的区别导致的。限于时间和经费,没有收集手术标本的小伙伴可用这些工具尝试探讨一下目标分子的临床意义,个人觉得只要是阳性结果(与生存相关)都可以往SCI中放,多凑几个图。


