文章
这是一篇2017发表在Genome Medicine上的文章A practical guide to single-cell RNA-sequencing for biomedical research and clinical applications
背景
- 单细胞测序:《Nature Methods》2013年度技术;《Nature》2017年7月刊的封面推荐 ;《Science》2018十大科学突破榜首
- 研究细胞的方法:基因组DNA序列(碱基如何排列、各个序列的丰度)、染色质结构(常听说的3C、4C、5C、HiC等)、mRNA序列(排列与丰度)、非编码RNA、蛋白表达、蛋白修饰、细胞代谢【因此不只有scRNA,还有sc基因组、sc表观组】
- 一个细胞中的待研究分子是微量的,因此我们一般使用几千细胞或直接取组织(上千万甚至上亿),这样就可以积累足够的分子信息,可以开展 Genome-wide association studies (GWASs) 、鉴定SNPs
- 主要做什么:分离新细胞亚群、构建细胞间互作网络、体内与体外实验反应、细胞在不同器官的情况、不同人群比较、不同物种比较
- 总体≠个体:细胞异质性的存在(受精卵发育成个体,最终去向十分多样=》肌肉细胞、神经细胞等等,why?;肿块中心、周围、转移中的细胞各异,分离出来判断疗法有效性)
- 挑战:同时检查单个细胞中表达的数千种蛋白质(蛋白组研究范畴),这个完整性有待提高
名词
Barcoding
- 之前做单细胞,真的是一个个细胞取出来,然后独立构建文库测序(比如:流式细胞术、激光捕获显微切割LCM=》组织切片),但是这通量非常低(有点Sanger测序和二代测序对比的感觉)。
- 后来发展出高通量的方法,主要是给每个细胞加上独一无二的DNA序列(就是条形码barcode,就是为了识别),然后测序时将相同的barcode序列归为同一个细胞来源
- 单细胞转录组可以在polyT引物5'端加上barcode;单细胞基因组目前主要利用高效转座酶(transposase)Tn5实现
Spike-in
- Spike-ins can be used for assessing the level of technical variability and for identifying genes with a high degree of biological variability
- 每个细胞都是独特的,和普通的Bulk RNA-seq不同,材料不容易获得,不太好做重复,因此通过生物学重复来评价技术手段/数据质量的方法不靠谱。
- 但是数据质量还是需要评价的,那么就通过向每个细胞裂解液中加入已知序列与一定数量的合成mRNA,例如 external RNA control consortium (ERCC)【翻译的话,姑且翻译成:外源RNA对照联盟】开发的“内参”,可以根据RNA读数判断样本间差异
- 高ERCC含量与低质量数据相关
- 但是使用spike-in也有一些问题要注意:
- has to carefully calibrate the concentration that results in an optimal fraction of reads from the spike-ins
- spike-in mixes are sensitive to degradation
- captured less efficiently than endogenous transcripts
- Spike-in不适用于droplet-seq的方法
- UMI( Unique molecular identifier )
- barcoding的变体,待扩增的RNA分子用随机n-mer寡核苷酸标记。设计不同标签的数量,大大超过待扩增的转录本,产生独特标记的分子,并允许控制扩增偏差【例如10-mer的UMI,就会有 4的十次方 约等于100万种变化】
- UMI是一段随机序列,每一个DNA分子都有自己的UMI序列。可以大大降低PCR误差(比如:原来两个样本中某基因表达量相同,但是由于两个样本扩增效率不同,样本1为99%,样本2只有95%,那么同时扩增40个循环,这同一个基因就有了
0.99^40 / 0.95^40 = 5.2倍差异,因此本来没有差异也会因为外界因素扩增效率的影响而产生“假阳性”) - UMI只用在3'转录本测序的方法中,如CEL-seq2, Drop-seq, MARS-seq
Dropout
- 基因在一个细胞中有表达,但在另一个细胞中未检测到(按照道理,每个基因应该都可以检测到,只是表达量多少)
- 可能源于RNA总量少导致扩增建库丢失 或者 RNA表达随机性
Mass cytometry
- 基于流式细胞法和质谱,其中使用元素标签标记的抗体检测蛋白质表达 - 允许在一次实验中对数千个单细胞上的数十种蛋白质进行平行测定
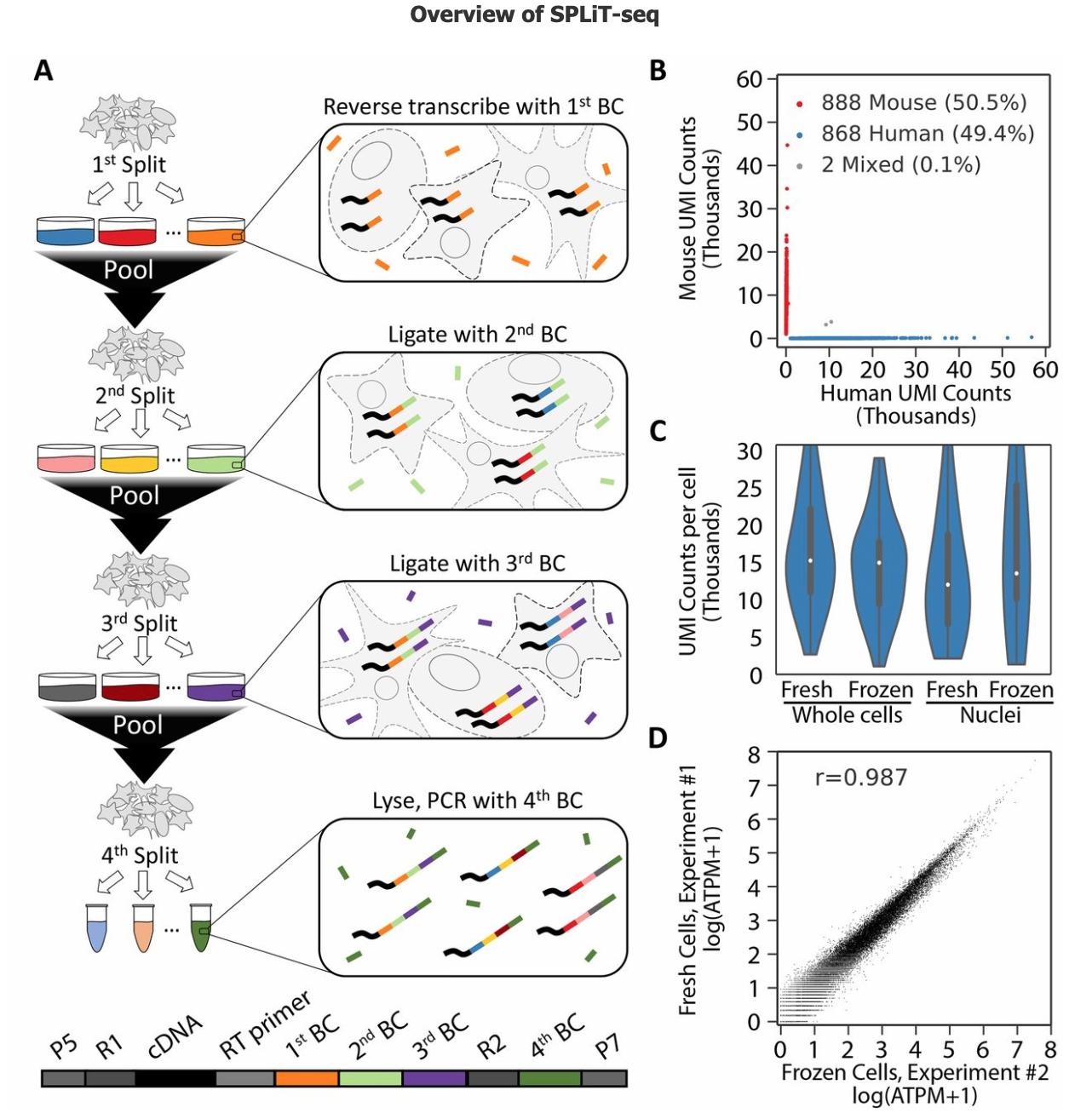
Split-pooling
- (Rosenberg et al. ) combinatorial barcoding to profile single-cell transcriptomes without requiring the physical isolation of each cell
- https://www.rna-seqblog.com/split-seq-single-cell-profiling-with-split-pool-barcoding/
Basic step
- The first, and most important, step in conducting scRNA-seq has been the effective isolation of viable, single cells from the tissue of interest
- Next, isolated individual cells are lysed to allow capture of as many RNA molecules as possible.
- Next, poly[T]-primed mRNA is converted to complementary DNA (cDNA) by a reverse transcriptase.
- Then, amplified and tagged cDNAfrom every cell is pooled and sequenced by NGS.
Types of material
- 理论上,任何真核生物细胞都可以
- Primary cells
- 胚胎 embryo
- 肿瘤 tumours
- 神经 nervous system
- 造血 haematopoietically derived cells
- The Human Cell Atlas
- 2017年启动,“媲美人类基因组计划”,核心技术=》单细胞组学
- 对人类37万亿个细胞进行细胞采集、分类和绘图,侧重描绘组织,而不是整个器官;后期阶段可以纳入器官及感兴趣的疾病小群体
- 2018.3.8,Sanger研究所宣布人类发育细胞图谱(Human Developmental Cell Atlas ,HDCA)的初步项目25万个发育细胞测序完成
补充:测序平台
- 10X Genomics
- 2016.2推出 Chromium;
- 通量高(7分钟内完成100~80,000个细胞的捕获),周期短,成本低,细胞捕获效率高(单个样本细胞捕获率高达65%);细胞活性要求>90% =》 适用于发现新细胞
- 横向孔道逐个导入凝胶微珠Gel beads =》 第一个纵向道输入细胞 =》Gel吸附细胞=》微流控技术送到第二个纵向通道(“油tube”)=》油滴GEMs 【因此,一个油滴就是一个Gel bead,也就是一个细胞】=》收集到EP管 =》每个Gel bead表明都放满了各不相同的Barcode和UMI序列+polyT =》细胞裂解,polyT抓取mRNA的3'polyA
- BD Rhapsody
- 分子标签技术(每个转录本标记特异性分子标签)=》单细胞水平上基因表达谱的绝对定量
- 单次实验可制备100-10000个单细胞文库
- CytoSeq特有的蜂窝板技术(20W+的微孔),避免了10X中存在的概率碰撞影响捕获效率问题
- 可以多样本混合捕获;成像系统;转录组-蛋白组联合分析
- Wafergen公司 ICELL8
- 基于微流控芯片,5184个反应孔
- 每次运行可分离500-1000个细胞
- 捕获效率为30%,成本相对较低
- Fluidigm公司C1
- 通量低、成本高(2000-3000细胞需要18000-100000美元)、周期慢
- 同时捕获96个细胞
- 全长转录组
- llumina Bio-Rad
- ddSEQ
- 一次性检测8个样本,每个样本可以得到500~10000个细胞
- 组织功能、病情进展和治疗反应方面的协同作用
- 捕获效率低,仅为3%;成本低
- 1CellBio => InDrop
- Dolomite => µEncapsulator


