一般在基因组组装中,我们常用的拼接方法是,对测序得到的二代数据直接用soapdenovo等软件进行基因组组装。但是这些方法一般只是适用于单一的基因组序列。
最近做的一个课题是从植物混合测序的数据中(核基因组、线粒体基因组、叶绿体基因组DNA混合测序的数据)分别组装出细胞器(叶绿体、线粒体)的基因组。这里面问题的难点在于,在叶绿体基因组、线粒体基因组、核基因组之间存在比较频繁的DNA迁移;所以虽然叶绿体基因组和线粒体基因组很小(叶绿体一般几十kb到100多kb,线粒体基因组一般几百kb),但是在组装的时候,有太多的干扰信息,很难将叶绿体或者线粒体基因组组装起来。
当然也有人认为,为什么不将叶绿体和线粒体基因组分别分离,当然这是一个策略之一,但是不可避免地存在三者之间相互污染;其二是,现在在做植物核基因组的过程中,有些时候细胞器的基因组没有完全去除或者未作去除,可以利用一下这个混合数据,组装一下细胞器的基因组,也可以节省一笔经费。
去年实验室一个师兄基于454的混合数据组装了牛耳草的细胞器基因组,基因组数据的已经发表在plos one,但是这里推荐的是其发表在plant method上的一篇方法学的文章。
An efficient procedure for plant organellar genome assembly, based on whole genome data from the 454 GS FLX sequencing platform
Motivation
Complete organellar genome sequences (chloroplasts and mitochondria) provide valuable resources and information for studying plant molecular ecology and evolution. As high-throughput sequencing technology advances, it becomes the norm that a shotgun approach is used to obtain complete genome sequences. Therefore, to assemble organellar sequences from the whole genome, shotgun reads are inevitable. However, associated techniques are often cumbersome, time-consuming, and difficult, because true organellar DNA is difficult to separate efficiently from nuclear copies, which have been transferred to the nucleus through the course of evolution.
Results
We report a new, rapid procedure for plant chloroplast and mitochondrial genome sequencing and assembly using the Roche/454 GS FLX platform. Plant cells can contain multiple copies of the organellar genomes, and there is a significant correlation between the depth of sequence reads in contigs and the number of copies of the genome. Without isolating organellar DNA from the mixture of nuclear and organellar DNA for sequencing, we retrospectively extracted assembled contigs of either chloroplast or mitochondrial sequences from the whole genome shotgun data. Moreover, the contig connection graph property of Newbler (a platform-specific sequence assembler) ensures an efficient final assembly. Using this procedure, we assembled both chloroplast and mitochondrial genomes of a resurrection plant, Boea hygrometrica, with high fidelity. We also present information and a minimal sequence dataset as a reference for the assembly of other plant organellar genomes.
基本的workflow如下:
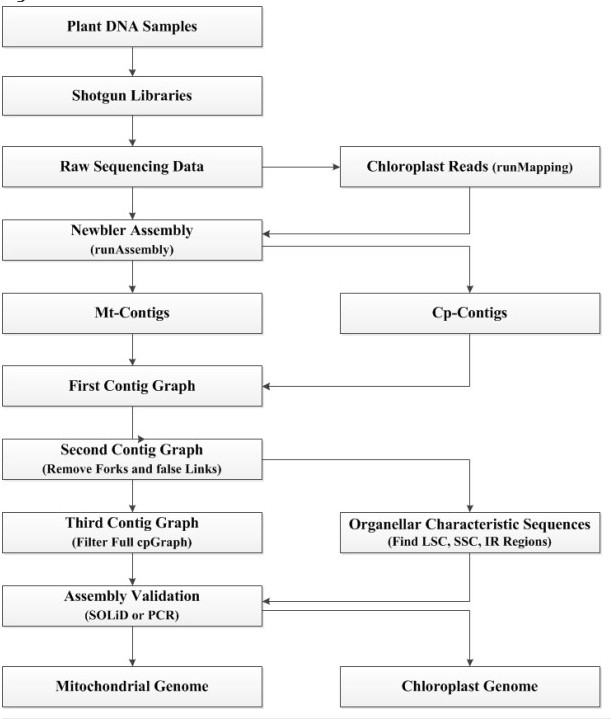
这篇文章的基本策略:
对于叶绿体基因组
一般叶绿体基因组学相对比较保守,而且现在已经测序的也有200多,可以用已经测序的叶绿体基因组作为reference,从454reads中调出叶绿体相关的reads,进行组装。
接着选出一个contig作为seed,然后用bb.454contignet这个脚本,会从newbler组装的结果中(主要是 454ContigGraph.txt)找出与seed相连的contig,然后将那些鱼原始seed相连的contig进一步作为seed,递归地找下去,当然还有一些其他的cutoff可以设置。最后画出一个从原始seed开始,找出来的contig连接图。去掉上面一些false 分支和contig就能得到完整的叶绿体contig连接图。如下图:
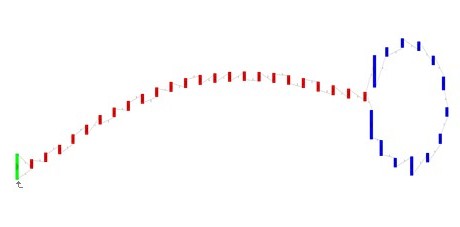
- 对于线粒体基因组
先用newbler对所有的454reads进行组装,然后基于叶绿体或者线粒体上保守的基因,选出能确定是线粒体的contig作为seed。然后用bb.454contignet这个脚本,会从newbler组装的结果中(主要是 454ContigGraph.txt)找出与seed相连的contig,然后将那些鱼原始seed相连的contig进一步作为seed,递归地找下去,当然还有一些其他的cutoff可以设置。
由于每个细胞中核基因组、叶绿体基因组、线粒体基因组的拷贝数各不相同,如下图对牛耳草混合基因组数据的统计分析,基于454reads组装(使用newbler进行组装)得到的contig的覆盖度也不相同,可以利用覆盖度信息对来自核基因组、叶绿体基因组、线粒体基因组的contig进行大致区分,然后去掉明显属于叶绿体基因组上的contig分支,以及其他从覆盖度上能明显区分不属于线粒体基因组的分支,经过几轮不断修正得到玩真过的线粒体contig连接图。
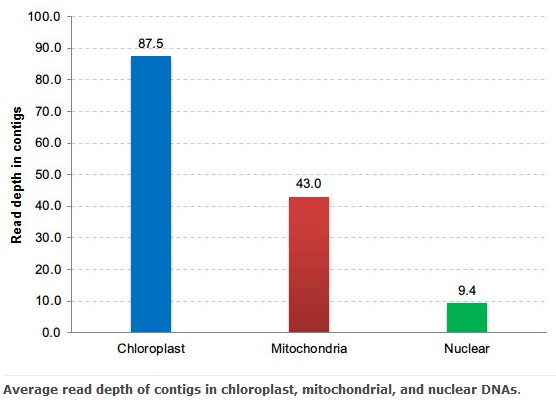
另外,文中提及的bb.454contignet这个脚本下载链接:http://www.vcru.wisc.edu/simonlab/sdata/software/
关于这个软件,近期软件作者发飙了一篇文章,大家也可以读一读:
Massimo Iorizzo, Douglas Senalik, Marek Szklarczyk, Dariusz Grzebelus, David Spooner and Philipp Simon.
De novo assembly of the carrot mitochondrial genome using next generation sequencing of whole genomic DNA provides first evidence of DNA transfer into an angiosperm plastid genome
BMC Plant Biology 2012, 12:61



1F
这样搞序列很容易出问题的,线粒体中存在有大量叶绿体和细胞核基因组的插入序列,如果提不到纯的线粒体基因组,就会造成其它基因组的混杂,如何判断liberies中的叶绿体和核基因组数据是混杂进入的还是真实属于线粒体基因组中的?454的读长太短,恐怕不好解决吧。
B1
@ Plant bio 其实用454的数据这样组装是没问题的,对于混合数据这种策略。当然这也仅是一种组装策略而已,不是一种软件和算法。我用这种策略组装叶绿体的时候,分别独立用454数据和hiseq的数据,拼出来的结果是一样的。但是线粒体的话,组装完之后,还是要补充mate pair、pair end 数据来确定重复序列的连接。
一般在做高等植物基因组的时候,都会有hiseq的数据吧,所以可以用来检查一下。我现在做的一个课题中,测了一些pacbio的数据,也可以用于校正454连接的contig是否正确。
B2
@ ybzhao 哦,用hiseq数据支持是个好办法。还想请教您一个问题,454测序后的poly碱基错误比较多,有没有比较快的解决办法啊?
B3
@ Plant bio 我的方法是先把基因组拼出来,然后再把hiseq的reads往上mapping,因为hiseq reads的准确性和覆盖度是非常高的,可以纠正454数据里面的一些测序错误。
B4
@ ybzhao 好的,谢谢!
2F
你好,请问bb.454contignet生成文件中recursion limit是什么意思啊?
B1
@ Plant bio bb.454contignet 中
–level 这个参数控制 recursion次数,contig graph上的 recursion limit 表示由于 –level 这个参数设定限制,该contig 不继续往下找与他相连的contig了
3F
请问这个recursion次数在拼接中有什么具体的意义吗?
4F
请问用newbler如何拼接contigs