SignalP是一个信号肽预测服务器,它的功能是预测给定的氨基酸序列中是否存在潜在的信号肽剪切位点及其所在,原核生物和真核生物都可以进行预测。目前服务器提供的是SignalP 4.0版本。在线服务器网址:http://www.cbs.dtu.dk/services/SignalP/
SignalP 4.0 server predicts the presence and location of signal peptide cleavage sites in amino acid sequences from different organisms: Gram-positive prokaryotes, Gram-negative prokaryotes, and eukaryotes. The method incorporates a prediction of cleavage sites and a signal peptide/non-signal peptide prediction based on a combination of several artificial neural networks.
下面简单介绍一下使用说明:
网页界面:
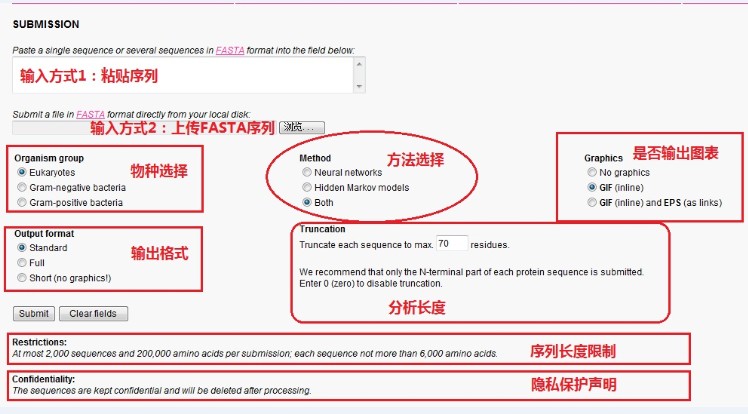
提交序列中的选项
A.序列输入方式:
有两种方式:一种是直接输入FASTA格式的氨基酸序列,可以是一条也可以是多条;另一种是本地上传包含含有FASTA格式序列的文件。
这里要注意我们必须输入氨基酸序列,看过有的朋友用核酸序列去预测的;输入的氨基酸序列为单字母简写形式。除了20中氨基酸外,仅允许使用X代表未知氨基酸。所有序列中的其他字母(非20种氨基酸简写和X的)被视为X;所有的非字母字符将会自动忽略。
还要注意的是对序列长度和多少的限制:每次最多提交2000条序列进行分析;总氨基酸残基不能超过20万;每条序列不能超过6000残基。其实一般来讲我们都不会有这么多序列一起去预测的O(∩_∩)O哈哈~
B.物种选择:
根据你预测的蛋白质来源选择革兰氏阳性细菌、革兰氏阴性细菌或真核生物。
C.方法选择:
可选神经网络法(neural network,结果中简写NN);隐马可夫模型(hidden markov models,结果中简写HMM)以及联合两种方式。默认为两种一起预测。
两种方法各自的优势:HMM主要在于预测是否含有信号肽;NN主要为预测信号肽位点。
D.是否输出图表:
图表方式显示的界面要直观一些,默认为GIF格式,不用改了。
E.输出格式:
默认为标准模式,输出的结果中含有预测的图表,序列的总体分析数据如是否含有信号肽及其位点;Full模式除Standard中含有的数据外还有每个氨基酸对应的相应分析数据;Short的结果仅有序列的总体分析数据。同样建议默认不要改变。因为full模式中数据对我们来说意义不大。
F.分析长度:
一般来讲,信号肽在蛋白质N端,极少超过45个氨基酸残基。所以在序列中仅N端部分序列对信号肽的分析预测具有重要作用。默认为分析N端70个氨基酸,后面的不做分析。如果你想要全部的分析将此值设置为0即可。
G.隐私声明:
分析结束后会删除你提交分析的数据。
结果显示
神经网络法NN中结果主要主要涉及3个值:C、S和Y。
S值:每个氨基酸对应1个S值,在结果显示的图表中有一个曲线显示S值的变化趋势,(在full模式中可以看见具体数值),信号肽区域的S值较高。
C值:剪切位点值。每个氨基酸会有一个C值,在剪切位点处C值是最高的。
Y值:Y-max综合考虑S值和C值的一个参数,其比单独考虑C值要更精确。因为在一条系列中C值可能有不止一个较高的位点,但是剪切位点只有一个;此时的剪切位点就由Y-max值来推测的,为S值是陡峭的位置和具有高C值的位点。
在现在的3.0版本中还有两个值S-mean和D值。
S-mean是从N端氨基酸开始到剪切位点处各氨基酸的平均S值。
D值是S-mean和Y-max的平均值,对区分是否为分泌蛋白具有重要作用。
隐马可夫模型(HMM)主要计算序列中是否含有信号肽,在真核生物的预测中还有signal anchor的一个参数(相当于信号肽),并进一步分为n-region、h-region和c-region三个部分。
这里我就直接用服务器中提供的结果说明为例。
在分泌蛋白的预测结果中,NN法Signal peptide列中结果为yes,并根据C值、S值和Y值等给出潜在的剪切位点;图表右上角处有C值、S值和Y值的曲线颜色指示,图表中有各值的变化趋势曲线。
HMM法文本结果显示其含有信号肽的可能性以及潜在的剪切位点;图表中给出信号肽不同区域划分的预测。
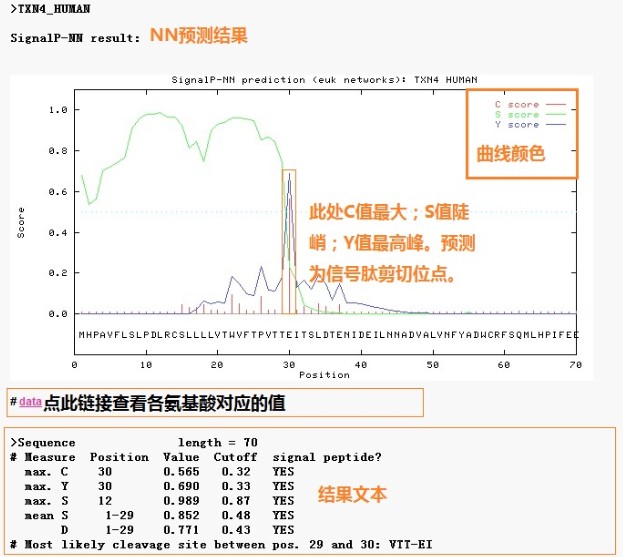
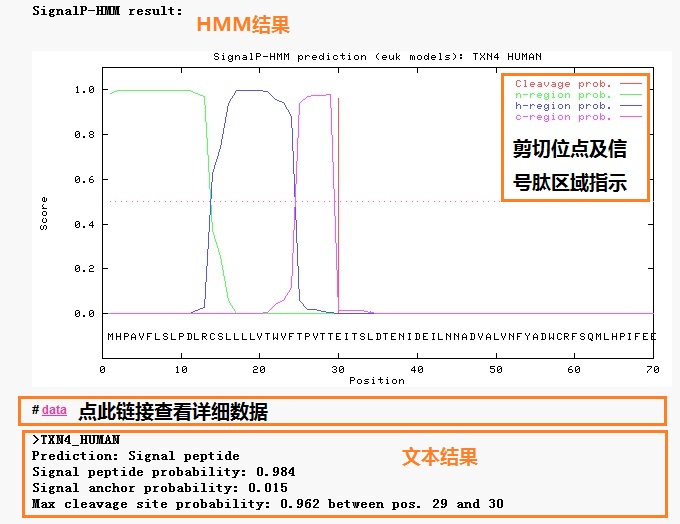
如果在输入序列时选择输出格式为“short”,显示结果如下,结果看起来就不是很直观了,建议还是不要选择short输出类型。




1F
Thanks for sharing!!给常给力!