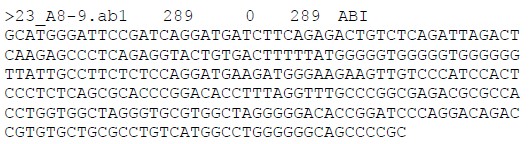简介
Phred是 phred\phrap 软件包的一部分,phred\phrap 软件包由华盛顿大学分子生物技术学院的 Phil Green 和 Brent Ewing 开发,主要用于学术科研活动。Phred 功能是处理测序仪直接 生成的色谱图,给出相应的碱基和质量值。不同的测序仪会给出不同的色谱文件,Phred 能够识别三种格式的色谱文件,SCF, ABI 和预先处理的 ESD 格式。
碱基的测序质量值 Q 和此碱基出错的概率 Pe 相关。公式:
Q = -10 log10( Pe )
下载
该软件包可以从phrap的的网站申请后免费下载,网站链接:http://www.phrap.org/consed/consed.html#howToGet
安装
1、上传 phred 的压缩包到本地 linux/unix 运算服务器;
2、解压缩:
gzip –d phred-dist-020425.c-acd.tar.gz tar –xvf phred-dist-020425.c-acd.tar
3、查看解压缩后的文件:
4、编译源程序:
在命令行键入 make all
敲入“make >& make.log”,完成 phred 的编译。
敲入“make daev”,完成 phred 程序包中 daev 程序的编译。 编译完成后,可将执行文件 phred、daev 拷到/usr/local/genome/bin 目录下面。
默认是用 cc 编译源代码,如果编译报错的话,很可能是 CC 编译器有问题,可以试一下用 gcc
编译,将 Makefile 文件中 CC= cc 改为 CC=gcc 或用命令:make CC=gcc all
5、设置环境变量
为了以后使用方便,可以把 phred 需要的环境变量设置在用户宿主目录下面的.profile和.bashrc 或.cshrc 文件里面,把配置文件的路径付给 PHRED_PARAMETER_FILE, 例如:
1.C shell,tcsh:
% setenv PHRED_PARAMETER_FILE /usr/local/PhredPar/phredpar.dat
2.sh,bash:
$ HRED_PARAMETER_FILE=/usr/local/PhredPar/phredpar.dat
$ export PHRED_PARAMETER_FILE
注意路径要根据不同用户安装目录的不同做相应的修改,不能照抄这个例子。
phredpar.dat文件内容:
最后两行:
"no_matching_string" unknown unknown unknown end chem_list
如果有如下报错信息,说明环境变量还没有设置成功,需要重新设置环境变量:
FATAL_ERROR: PHRED_PARAMETER_FILE environment variable not set. type `phred -doc' for more information
使用
程序运行命令行:
phred -id <chromat-file-dir> -pd <phd-file-dir> [other options]
键入 phred -help(-h)查看帮助信息:
| parameter | argument | default | description |
| --------- | -------- | ------- | ----------- |
| -if | <filename> | none | read input filenames from file |
| -id | <dirname> | none | read input files from <dirname> |
| -zd | <dirname> | path | uncompress program path |
| -zt | <dirname> | /usr/tmp | uncompress temporary directory |
| -st | <type> | fasta | sequence file type (fasta|xbap) |
| -s | none | nofile | write *.seq sequence file(s) |
| -s | <filename> | nofile | write <filename> sequence file |
| -sa | <filename> | none | append sequence files to <filename> |
| -sd | <dirname> | nofile | write *.seq file(s) to <dirname> |
| -qt | <type> | fasta | quality file type (fasta|xbap|mix) |
| -q | none | nofile | write *.qual quality file(s) |
| -q | <filename> | nofile | write <filename> quality file |
| -qa | <filename> | none | append quality files to <filename> |
| -qd | <dirname> | nofile | write *.qual file(s) to <dirname> |
| -qr | <filename> | nofile | write quality report to <filename> |
| -p | none | nofile | write *.phd.1 file(s) |
| -p | <filename> | nofile | write <filename> phd file |
| -pd | <dirname> | nofile | write *.phd.1 file(s) to <dirname> |
| -cv | <version> | 2 | SCF format version (2 or 3) |
| -cp | <precision> | maxval | SCF data precision in bytes (1 or 2) |
| -cs | none | no scale | always scale traces in SCF files |
| -c | none | nofile | write * phred SCF file(s) |
| -c | <filename> | nofile | write <filename> phred SCF file |
| -cd | <dirname> | nofile | write * SCF file(s) to <dirname> |
| -d | none | nofile | write *.poly poly file(s) |
| -d | <filename> | nofile | write <filename> poly file |
| -dd | <dirname> | nofile | write *.poly file(s) to <dirname> |
| -raw | <seq name> | NULL | seq name written in output files |
| -log | nolog | write phred.log file | |
| -nocall | none | call | disable basecalling |
| -trim | <enzyme seq> | notrim | enable auto trim |
| -trim_alt | <enzyme seq> | notrim | enable alternate auto trim |
| -trim_cutoff | <n> | 0.05 | trim_alt error probability |
| -trim_fasta | none | none | trim FASTA bases and qual. values |
| -trim_scf | none | none | trim SCF bases and qual. values |
| -trim_phd | none | none | trim base call data in phd files |
| -trim_out | none | none | trim data in most output files |
| -nonorm | none | normalize | disable trace normalization |
| -nosplit | none | none | no compressed peak splitting |
| -nocmpqv | none | none | no compressed peak quality values |
| -ceilqv | <ceiling qv> | none | quality value ceiling value |
| -beg_pred | <point> | none | set peak prediction start point |
| -exit_nomatch | none | none | exit immediately for chromats with |
| unmatchable primerID string | |||
| -process_nomatch | none | none | process chromats with unmatchable |
| primerID string using | |||
| '__no_matching_string__' entry | |||
| -v | <n> | none | verbose operation <n> = 1 to 63 |
| -tags | none | not tags | label common messages with tags |
| -V | none | none | show version |
| -help | none | none | help |
| -h | none | none | help |
| -doc | none | none | show phred documentation |
For the warning messages `unable to identify chemistry and dye' and `unknown chemistry (...) in chromat ...' please read the phred documentation using the command `phred -doc'.
输入
测序仪产生的峰图文件,可识别:SCF, ABI model 373 and 377 DNA sequencer chromatogram,and MegaBACE ESD chromatograms files
输出
运行过程中的屏幕输出:
chromat_dir/10_A8-9.ab1
chromat_dir/11_A8-9_R.ab1
chromat_dir/15_A8-9.ab1
chromat_dir/21_A8-9.ab1
chromat_dir/22_A8-9.ab1
chromat_dir/23_A8-9.ab1
Warn 输出:
Chromat_dir/10_A8-9.ab1
unknown chemistry (KB_3730_POP7_BDTv3.mob) in chromat tmp/10_A8-9.ab1 add a line of the form "KB_3730_POP7_BDTv3.mob" <chemistry> <dye type> <machine type>
to the file phredpar.dat type `phred -doc' for more information
程序的输出结果是文件输出,格式可以是 FASTA 格式,也可以是 XBAP, PHD 格式或 SCF 格式。
1. Phd 文件,用于组装后 consed 查看编辑,名字为<filename>.phd.1
2. Fasta 格式的核酸序列文件
FASTA 头注释行包含修饰信息(序列没有影响),此行有如下格式:
a. 序列名称
b. phred 读出的碱基数
c. 序列开始部分被修饰掉的碱基数
d. 修饰后余下的碱基数
e. 描述输入文件类型
3. Fasta 格式的质量文件(和序列文件相对应,给出每个碱基的质量值)
参数
详细的参数列表及说明可以通过键入 phred –doc 查看:
$ phred -doc
输入选项:
-id 输入文件目录
运行选项:
输出选项:
参考文献
1. Ewing B, Green P: Basecalling of automated sequencer traces using phred. II. Error probabilities. Genome Research 8:186-194 (1998).
2. Ewing B, Hillier L, Wendl M, Green P: Basecalling of automated sequencer traces using phred. I. Accuracy assessment. Genome Research 8:175-185 (1998).