你会用到的网站:
IGV 官网http://software.broadinstitute.org/software/igv/download~ Broadinstitute出品
Java version8 下载: https://www.java.com/en/download/mac_download.jsp
IGV内置的物种基因组及基因组来源:http://software.broadinstitute.org/software/igv/Genomes
完整的官方帮助文档:http://software.broadinstitute.org/software/igv/book/export/html/6
写在前面:
之前mac不小心升级了一下java,然后igv就不能用了,要写教程必须降级java
- 首先,看官方说明,需要安装Java -8,9以上版本不支持。我的mac不知道什么时候更新到了java 10,按说可以向下兼容,但是事与愿违,igv不能正常使用了。
- 需要降级Java,mac用户可以直接参考,windows可以试下直接下载安装IGV:
- 先删除原来的java
- ⚠️:不要通过/usr/bin 删除 Java 工具来卸载 Java。此目录是系统软件的一部分,下次对操作系统执行更新时,Apple 会重置所有更改。
finder中进入/Library/Java/JavaVirtualMachines,然后删除之前的jdk.版本号
terminal打开终端,复制粘贴一下三条命令:sudo rm -fr /Library/Internet Plug-Ins/JavaAppletPlugin.plugin
sudo rm -fr /Library/PreferencesPanes/JavaControlPanel.prefPane
sudo rm -fr ~/Library/Application Support/Java
- 先删除原来的java
- 下载安装新的Java version8
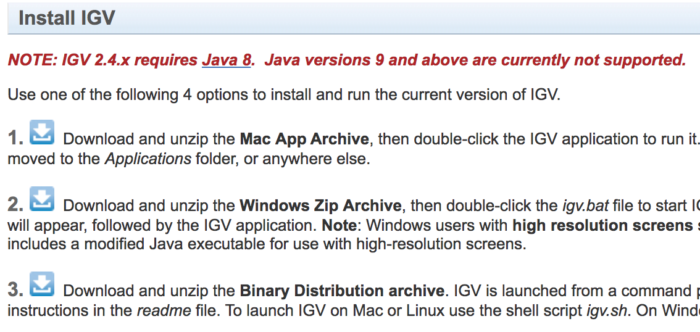
- 【Windows用户下载,解压后,点击igv.bat文件即可启动;如果启动失败,用记事本打开并编辑igv.bat文件,在文件的最后新起一行输入pause,保存后,再尝试打开,就可以在Windows下的命令行界面(cmd命令提示符)看到错误信息,再根据信息提示去解决问题;不过一般问题不大】
正文开始:
好啦,问题解决啦,开始正式IGV介绍!
什么是IGV
它是一款本地的探索基因组数据的可视化浏览器,有多个系统版本,支持多种不同类型的输入格式,包括芯片测序、二代测序、基因组注释文件等。推荐使用BAM与SAM格式,主要格式见下表
| 数据来源 | 文件格式 |
|---|---|
| 序列比对 | SAM/BAM |
| 显示覆盖率 | TDF |
| 拷贝数 | SNP、CN |
| 基因表达 | GCT、RES |
| 基因注释 | GFF3/GTF、BED |
| 突变数据 | MUT |
| 追踪参考基因组覆盖度、测序深度(UCSC) | WIG、BW |
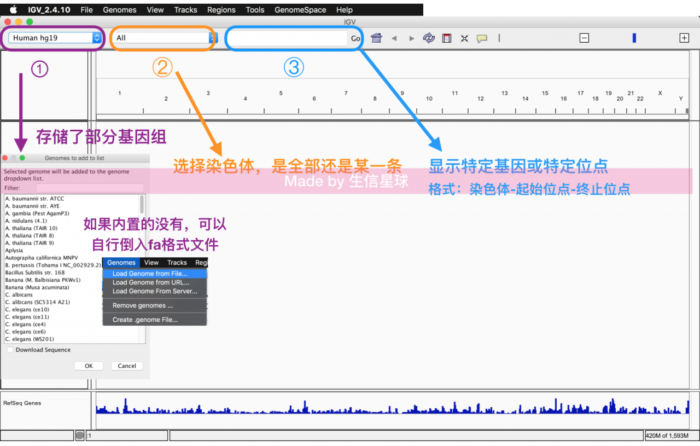
一睹IGV
每次打开会自动加载hg19.fa文件,也就是人类基因组,一会进入主界面
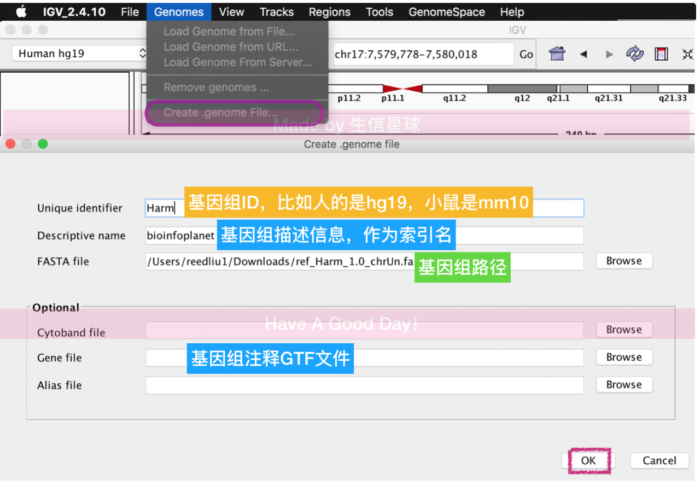
自己构建基因组信息
如果研究的物种没在上面的列表中找不到,就需要自己构建基因组信息,物种基因组在NCBI中的下载地址:ftp://ftp.ncbi.nlm.nih.gov/genomes/。下完基因组再顺便下载下来GFF或者GTF注释文件
这里我会举一个昆虫中一种——棉铃虫,这个基因组是17年2月更新在NCBI伤的,属于小众物种,IGV并没有收录。正好拿来练手,当然如果你研究的领域也有基因组被测出来,也可以试一试【注意:在提交基因组文件到IGV之前,要先构建索引】
这些工作都可以在本地进行,只需要打开你本地的git_bash或者putty/xshell或者terminal,解压缩基因组文件=》下载samtools(推荐用conda管理)=>构建索引samtools faidx genome.fasta=>IGV中 输入fasta文件路径=》提供注释文件(可以是组装基因组预测的基因注释文件,也可以是拼接转录组用的gtf文件)=〉其他选项可以忽略=》点击OK推弹出一个框让你输入存储路径
查看注释文件
下载注释文件
这里以人类基因组注释文件为例,下载gtf到电脑
#gff3和gtf都可以,gtf是gff3便于传输版而已nohup wget ftp://ftp.sanger.ac.uk/pub/gencode/Gencode_human/release_26/GRCh37_mapping/gencode.v26lift37.annotation.gtf.gz &nohuop wget ftp://ftp.sanger.ac.uk/pub/gencode/Gencode_human/release_26/GRCh37_mapping/gencode.v26lift37.annotation.gff3.gz &
构建索引
下载完不要急着导入,需要先构建索引
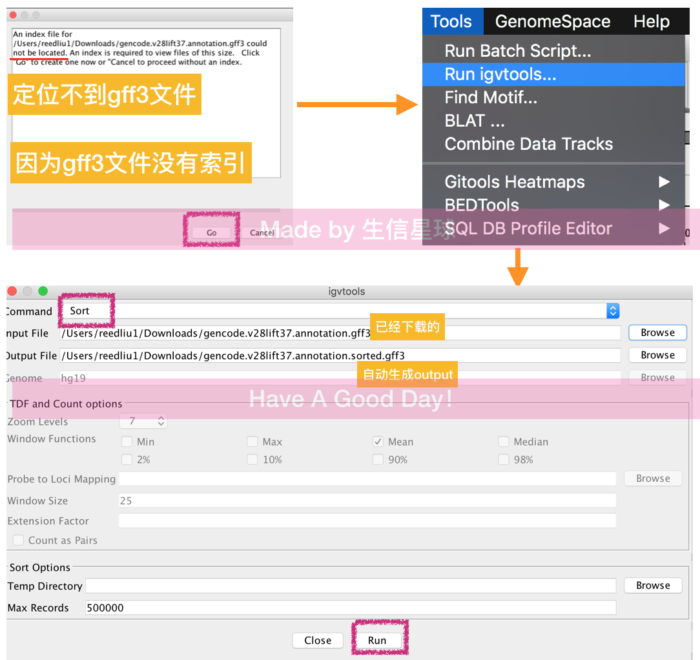
然后会生成gff3.idx或者gtf.idx文件,说明构建了索引,接着导入File -> Load from file ,选择sorted的注释文件
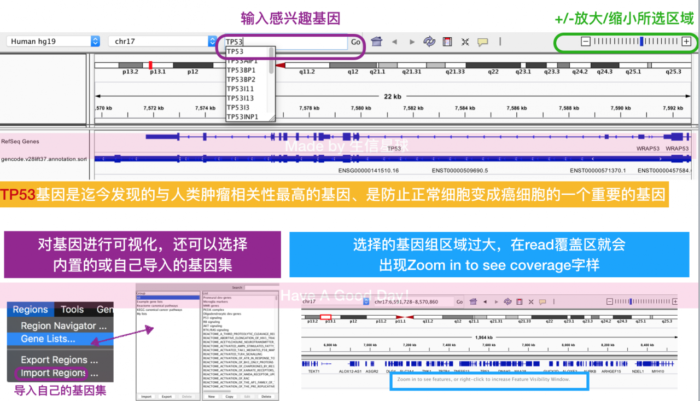
查看bam文件
这个练习数据获得很简单,有参考基因组,有测序数据就行:先把参考基因组用bwa index构建参考基因组索引,然后把测序数据用bwa mem比对到参考基因组得到sam格式,最后用samtools转换格式sam to bam
我这里准备的bam文件大小是2.8G,是由人类转录组测序数据得到的,准备的参考基因组是hg19,注释文件是gencode.v28lift37.annotation.sorted.gff3
- bam文件在导入前,要先使用samtools进行sort和index,
samtools sort test.bam test.sortsamtools index test.sort.bam,生成一个后缀为“.fai”的文件,它根据文件名自动和.bam关联, 另外这两个文件要在一个文件夹下,最后将bam导入IGV中 - 载入bam后,默认会出现两个track(翻译的话,可以理解为不同的轨道,显示不同的信息)Coverage track和Alignment track。
还有一种Splice Junction Track http://software.broadinstitute.org/software/igv/splice_junctions,可选的横跨剪切位点(spanning splice junctions)的reads视图
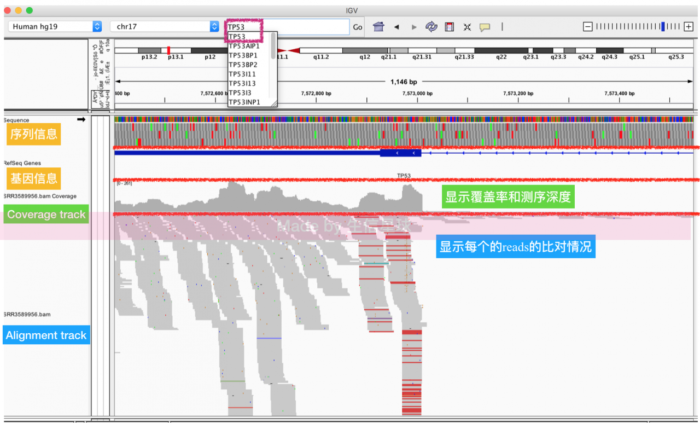
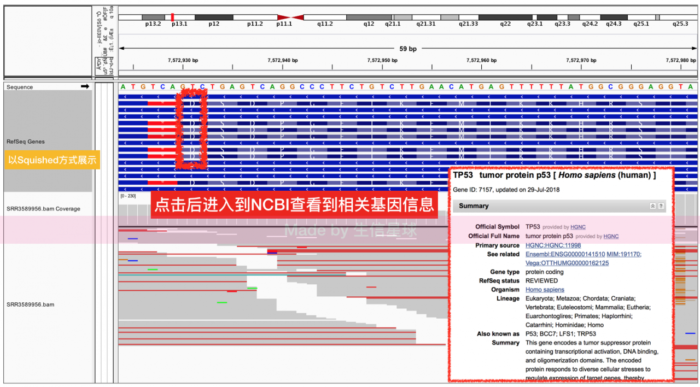
另外基因组信息也可以有collapsed、expanded、squished三种展示形式
- 查看Coverage track它的意思是显示比对文件的覆盖率和测序深度。横坐标是基因组上的位置,纵坐标是该位置的测序深度。【鼠标放在每一个位点都会显示一个小方框,其中的的内容就是显示总共有多少reads在这个位置,每个碱基各是什么】点右上角 放大reads可视化窗口后,track会以灰色的条形图来显示每个位点的测序深度。如果某一个核苷酸与参考序列相比,有超过20%的reads是不同的,条形图会显示不同的颜色
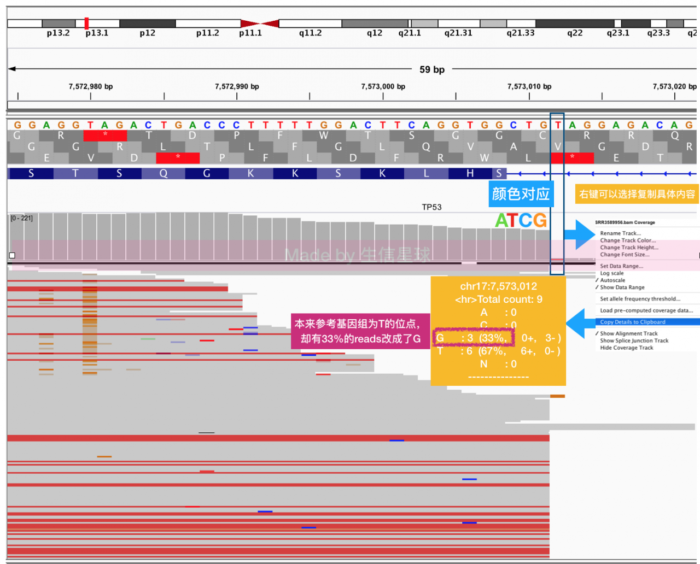

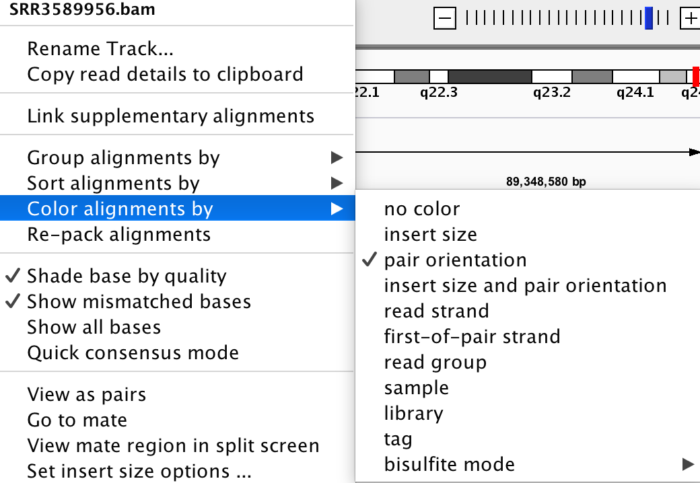
关于上图中的右键菜单,解释如下
| 功能 | 含义 |
|---|---|
| Rename Track | 更改track名 |
| Change Track Color | 更改背景色,比如把Coverage Track灰色变紫色 |
| Change Track Height | 改变每一个track的高度 |
| Change Font Size | 改变IGV最左侧字体大小 |
| Set Data Range | 覆盖深度的范围设置 |
| Log scale | 用对数尺度作图 |
| AutoScale | 是否自动缩放 【根据不同区域的深度情况自动调整深度范围】 |
- 比较多个基因
如果想比较多个基因的表达峰图,可以先在IGV中添加相应的基因列表,然后针对特定的列表选择view,就可以在一个屏幕显示所有加入的基因
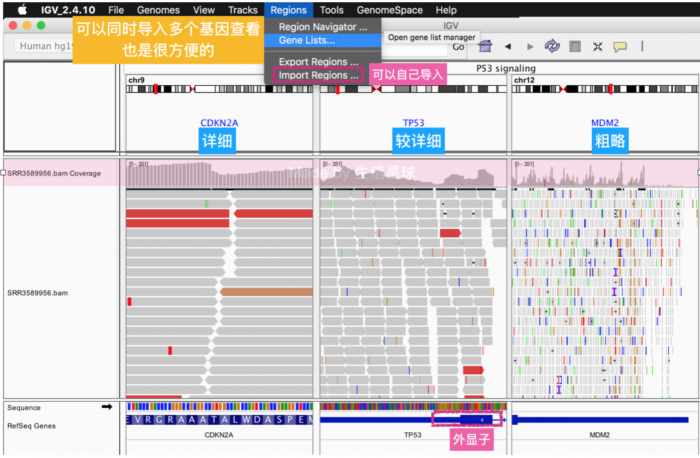
这里看到有许多颜色,这些颜色是根据定义不同比对类型而不同,
- 查找结构变异灰色:与参看基因组可以比对的reads紫色I :插入(鼠标查看插入的碱基信息)黑色横线:——缺失
补充学习:
官方链接http://software.broadinstitute.org/software/igv/book/export/html/37 讲座:https://www.youtube.com/watch?v=IILfC3Uc6Vo


