简介
Phd2fasta 是 phred\phrap 软件包的一部分,phred\phrap 软件包由华盛顿大学分子生物技 术学院的 Phil Green 和 Brent Ewing 开发,主要用于学术科研活动。Phd2fasta 将 phred 产生 的 phd 文件转换为 fasta 格式的核酸和质量文件,便于 crossmatch 和 phrap 程序应用。
下载
该软件包 可以从 phrap 的的网站申请,申请通过后邮件发送,申请链接 :
http://www.phrap.org/consed/consed.html#howToGet
安装
1、上传 phd2fasta 的压缩包到本地 linux/unix 运算服务器;
2、解压缩:
gzip –d phd2fasta-acd-dist.tar.gz tar –xvf phd2fasta-acd-dist.tar
3、编译源程序:
在命令行键入 make,编译完成后,可将执行文件 phd2fasta 拷到统一的可执行程序目录, 如:/usr/local/genome/bin 下面,源文件包可删除。
编译成功无提示信息。
使用
程序运行命令行:
phd2fasta –id phd_dir –os out.fas –oq
直接键入 phd2fasta 的屏幕提示:
no input files specified no output file specified
使用帮助可通过命令 phd2fasta -h 获取:
输入
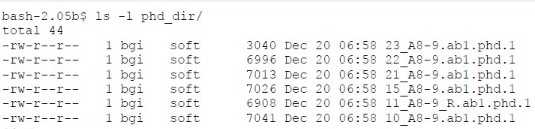
此程序的输入文件为 Phred 产生的 phd 文件:
输出
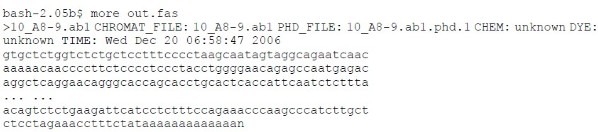
此程序运行无屏幕输出,结果为 fasta 格式的序列和质量文件。
序列文件:
质量文件:
参数
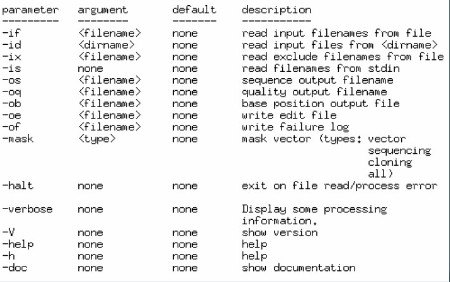
详细的参数说明可以通过键入phd2fasta –doc查看:
| Input Options | |
| ------------- | |
| -id <directory name> | 读取目录中的的文件做为输入文件 |
| -if <file name> | 读取文件列表中的文件做为输入文件 |
| -is | 读取标准输入做为输入文件 |
| -ix <file name> | 读取不需运行的文件列表 |
| Output Options | |
| -------------- | |
| -os <file name> | 输出 FASTA 序列到文件. |
| -oq <file name> | 输出 FASTA 序列的质量到文件 |
| -ob <file name> | 输出序列碱基位置信息到文件 |
| -oe <file name> | 将 phd 文件中的编辑信息提取到文件中 |
| -of <file name> | phd2fasta 处理失败的文件写入日志文件 |
| Processing Options | |
| ------------------ | |
| -mask <type> | 用 x 屏蔽序列中的载体 |
| -halt | 如有错误则停止继续运行程序 |
| Misc | |
| -verbose | 显示进程信息 |
| -V | 显示 phd2fasta 版本 |
| -h, -help | 显示命令行参数列表 |
| -doc | 显示详细的帮助文档 |