简介
Phil Green 和 Brent Ewing 开发的 phrap 软件包的一部分,用于比对两套 DNA 序列,如:可 以用来找出序列中的载体序列,并产生屏蔽了载体的序列;也可以用于 cDNA 和 cosmid 的比对等。 和 blastn 相比速度较慢但敏感度较高(因其允许 gap 存在)
下载
包含在 Phrap 软件包中,Mail to phg@u.washington.edu
安装
1、上传 phrap 的压缩包到本地 linux/unix 运算服务器;
2、解压缩:
gzip –d phrap.tar.gz tar –xvf phrap.tar
3、编译源程序:
在命令行键入 make,如果数据集多于 64,000 条序列,或者序列中含有长于 64,000 bp 的序列, 则需要使用 cross_match.manyreads或cross_match.longreads,这两个程序编译命令为 make manyreads。
使用
命令行:
cross_match seq_file1 seq_file2 -minmatch 10 -minscore 20 -screen > screen.out
输入
标准 FASTA 格式的序列文件
参数
option name & default value
1. 比对分值控制参数
2. Banded search
3. 比对筛选
4. 输入相关参数
-default_qual 15 当没有质量文件存在时,设定的每个碱基的质量值,默认为 15
5. 输出相关参数
6. 其他
-indexwordsize 10 用于索引的字符数,此参数影响运行时间和内存使用
输出
1. *.log files,程序运行日志
2. *.screen 文件,被屏蔽了相应序列后的序列文件,FASTA 格式。(此文件仅当使用-screen 参 数时输出)。
3. 标准屏幕输出,可重定向到文件,如>screen.out,然后输入more screen.out 查看
4. matching entries (first file).
Query 序列(第一个输入文件)和 subject 序列(第二个输入文件)比对的情况,如果只有一个输入文件,则是这个文件中任意两个序列的比对情况。比对情况通过命令行的-minscore 和–masklevel 参数控制, 另外也受比对分值和 band search 的参数控制。报告按 query 序列顺序 输出,例如:
| 440 | 2.38 | 1.39 | 0.79 | hh44a1.s1 | 33 | 536 | (0) | C00311 | (3084) | 8277 | 7771 | * |
对各列阐述如下:
440 = smith-waterman 比对分值
2.38 = 比对部分的替换百分比
1.39 = 比对部分的删除百分比
0.79 = 比对部分的插入百分比
hh44a1.s1 = 第一个输入序列的名称
33 = 第一个输入序列比对起始位点
536 = 第一个输入序列比对终止位点
(0) = no. of bases in 1st sequence past the ending position of match
(so 0 means that the match extended all the way to the end of the 1st sequence)
C 00311: 和输入序列 00311 的互补链比对上
(3084): 第二个输入序列(互补链)比对开始前共有 3084 个碱基
8277= 第一个输入序列比对起始位点
7771 =第一个输入序列比对起始位点
* indicates that there is a higher-scoring match whose domain partly includes the domain of this match.
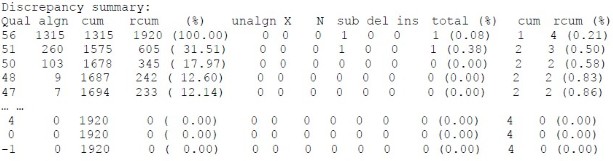
| Qual | algn | cum | rcum | (%) | unalgn | X | N | sub | del | ins | total | (%) | cum | rcum | (%) |
| 56 | 1315 | 1315 | 1920 | -100 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0.08 | 1 | 4 | 0.21 |
Qual 质量值
Algn 第一个输入序列这个质量值的碱基数
Cum 在 SWAT 比对中比上的碱基数
Rcum 累计比对上的碱基数(包含这个质量即更高的质量)
Unalgn 没有被包含进来的比对部分碱基数
每种类型的不一致的数目(sub 替换、del 删除、ins 插入)
cum(%)差异的总数和百分比
rcum(%)累计差异数和百分比