ATAC-Seq分析教程系列
ATAC-Seq分析教程:ATAC-seq的背景介绍以及与ChIP-Seq的异同
ATAC-Seq分析教程:用MACS2软件call peaks
ATAC-Seq分析教程:对ATAC-Seq/ChIP-seq的质量评估(一)phantompeakqualtools
ATAC-Seq分析教程:对ATAC-Seq/ChIP-seq的质量评估(二)ChIPQC
ATAC-Seq分析教程:用ChIPseeker对peaks进行注释和可视化
ATAC-Seq分析教程:用网页版工具做功能分析和motif分析
上一步骤用IDR对重复样本peaks的一致性进行了评估,同时得到了merge后的一致性的peaks——
sample-idr,接下来就是对peaks的注释。这篇主要介绍用Y叔的R包ChIPseeker对peaks的位置(如peaks位置落在启动子、UTR、内含子等)以及peaks临近基因的注释。
ChIPseeker
ChIPseeker虽然最初是为了ChIP-seq注释而写的一个R包,但它不只局限于ChIP-seq,也可用于ATAC-Seq等其他富集peaks注释,也可用于lincRNA注释、DNA breakpoints的断点位置注释等所有genomic coordination的注释,另外提供了丰富的可视化功能。
使用方法
使用ChIPseeker需要准备两个文件:一个就是要注释的peaks的文件,需满足BED格式。另一个就是注释参考文件,即需要一个包含注释信息的TxDb对象。Bioconductor提供了30个TxDb包,如果其中有研究的物种就可以直接下载安装此物种的TxDb信息。如果研究的物种没有已知的TxDb,可以使用GenomicFeatures包的函数(makeTxDbFromUCSC,makeTxDbFromBiomart)制作TxDb对象:
makeTxDbFromUCSC: 通过UCSC在线制作TxDb
makeTxDbFromBiomart: 通过ensembl在线制作TxDb
makeTxDbFromGRanges:通过GRanges对象制作TxDb
makeTxDbFromGFF:通过解析GFF文件制作TxDb
制作TxDb方法示例:
- 如用人的参考基因信息来做注释,从UCSC生成TxDb:
- library(GenomicFeatures)
- hg19.refseq.db <- makeTxDbFromUCSC(genome="hg19", table="refGene")
- 用GFF文件做裂殖酵母的注释
- download.file("ftp://ftp.ebi.ac.uk/pub/databases/pombase/pombe/Chromosome_Dumps/gff3/schizosaccharomyces_pombe.chr.gff3", "schizosaccharomyces_pombe.chr.gff3")require(GenomicFeatures)
- spombe <- makeTxDbFromGFF("schizosaccharomyces_pombe.chr.gff3")
具体步骤如下:
第1步:下载安装ChIPseeker注释相关的包
从Bioconductor直接下载,或从github安装最新版本
- source ("https://bioconductor.org/biocLite.R")
- biocLite("ChIPseeker")
- # 下载人的基因和lincRNA的TxDb对象
- biocLite("org.Hs.eg.db")
- biocLite("TxDb.Hsapiens.UCSC.hg19.knownGene")
- biocLite("TxDb.Hsapiens.UCSC.hg19.lincRNAsTranscripts")
- biocLite("clusterProfiler")
- #载入各种包
- library("ChIPseeker")
- library(clusterProfiler)
- library("org.Hs.eg.db")
- library(TxDb.Hsapiens.UCSC.hg19.knownGene)
- txdb <- TxDb.Hsapiens.UCSC.hg19.knownGene
- library("TxDb.Hsapiens.UCSC.hg19.lincRNAsTranscripts")
- lincRNA_txdb=TxDb.Hsapiens.UCSC.hg19.lincRNAsTranscripts
第2步:读入peaks文件
函数readPeakFile读入peaks文件
- nanog <- readPeakFile("./idr_out.bed/nanog_idr-bed")
- pou5f1 <- readPeakFile("./idr_out.bed/pou5f1_idr-bed")
第3步:注释peaks
peaks的注释是用的annotatePeak函数,可以单独对每个peaks文件进行注释,也可以将多个peaks制作成一个list,进行比较分析和可视化。
- # 制作多个样本比较的list
- peaks <- list(Nanog=nanog,Pou5f1=pou5f1)
- # promotor区间范围可以自己设定
- promoter <- getPromoters(TxDb=txdb, upstream=3000, downstream=3000)
- tagMatrixList <- lapply(peaks, getTagMatrix, windows=promoter)
- #annotatePeak传入annoDb参数,可进行基因ID转换(Entrez,ENSEMBL,SYMBOL,GENENAME)
- peakAnnoList <- lapply(peaks, annotatePeak, TxDb=txdb,tssRegion=c(-3000, 3000), verbose=FALSE,addFlankGeneInfo=TRUE, flankDistance=5000,annoDb="org.Hs.eg.db")
annotatePeak传入annoDb参数,即可进行基因ID转换,将Entrez ID转化为ENSEMBL,SYMBOL,GENENAME,peakAnnoList的结果如下:
- seqnames start end width strand annotation geneChr geneStart geneEnd geneLength geneStrand geneId transcriptId distanceToTSS ENSEMBL SYMBOL GENENAME flank_txIds flank_geneIds flank_gene_distances
- 5 chr3 196625522 196625873 352 * Intron (uc003fwz.4/205564, intron 2 of 9) 3 196594727 196661584 66858 1 205564 uc011bty.2 30795 ENSG00000119231 SENP5 SUMO specific peptidase 5 uc003fwz.4;uc011bty.2 205564;205564 0;0
第4步:可视化
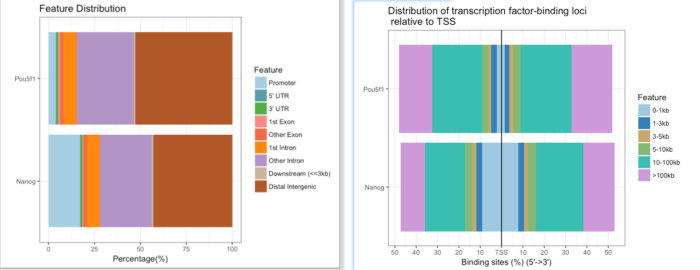
提供了多种可视化方法,如plotAnnoBar(),vennpie(),plotAnnoPie(),plotDistToTSS()等,下面展示了两个样本在基因组特征区域的分布以及转录因子在TSS区域的结合。
- plotAnnoBar(peakAnnoList)
- plotDistToTSS(peakAnnoList,title="Distribution of transcription factor-binding loci n relative to TSS")
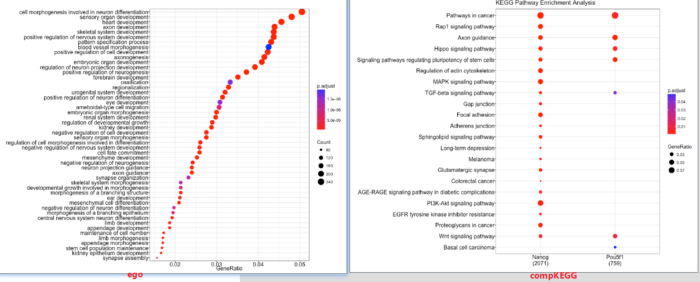
第5步:功能富集分析
提取peakAnnolist中的基因,结合clusterProfiler包对peaks内的邻近基因进行富集注释。
- # Create a list with genes from each sample
- gene = lapply(peakAnnoList, function(i) as.data.frame(i)$geneId)
- # Run GO enrichment analysis
- ego <- enrichGO(gene = entrez,
- keytype = "ENTREZID",
- OrgDb = org.Hs.eg.db,
- ont = "BP",
- pAdjustMethod = "BH",
- qvalueCutoff = 0.05,
- readable = TRUE)
- # Dotplot visualization
- dotplot(ego, showCategory=50)
- # Multiple samples KEGG analysis
- compKEGG <- compareCluster(geneCluster = gene,
- fun = "enrichKEGG",
- organism = "human",
- pvalueCutoff = 0.05,
- pAdjustMethod = "BH")
- dotplot(compKEGG, showCategory = 20, title = "KEGG Pathway Enrichment Analysis")
第6步:保存文件
- # Output peakAnnolist file
- save(peakAnnoList,file="peakAnnolist.rda")
- write.table(as.data.frame(peakAnnoList$Nanog),file="Nanog.PeakAnno",sep='t',quote = F)
- # Output results from GO analysis to a table
- cluster_summary <- data.frame(ego)
- write.csv(cluster_summary, "results/clusterProfiler_Nanog.csv")


