写在前面
以下内容均来自我在菲沙基因(Frasergen)暑期生信培训班上记录的课堂笔记
1.Compartment计算
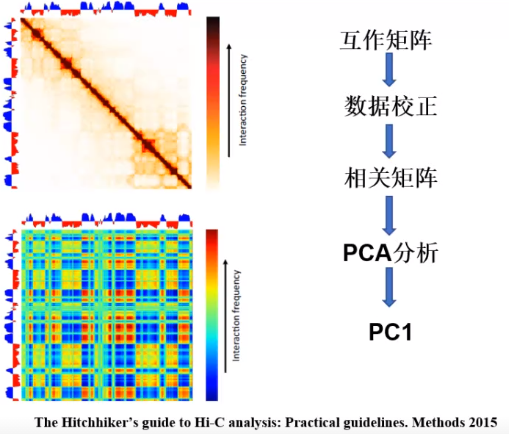
2.Compartment 分析流程
- 2.1 Cworld-dekker软件的安装
- git clone https://github.com/blajoie/cworld-dekker.git
- #Change directory to the `cworld-dekker` and install the `Perl` module:
- perl Build.PL
- ./Build
- ./Build install --install_base /your/custom/dir
- (ensure /your/custom/dir is added to your PERL5LIB path)
- #e.g.
- ./Build install --install_base ~/perl5
- # then in .bashrc
- export PERL5LIB=${PERL5LIB}:/home/<yourusername>/perl5/lib/perl5
2.2 分析所用数据
互作图谱分染色体matrix数据,如何获得请看:三维基因组技术(三):Hi-C 数据比对及HiC-Pro的使用
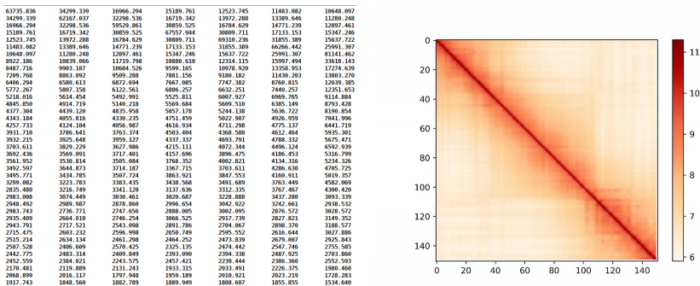
matrix数据
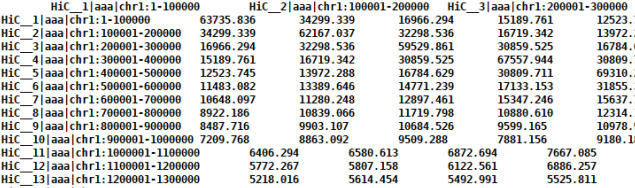
- 2.3 为矩阵添加header
header文件需要自己准备,操作采用cworld的addMatrixHeaders为矩阵文件添加header
- perl -I /software/cworld-dekker/ \
- /software/cworld-dekker/scripts/perl/addMatrixHeaders.pl \
- -i data/example.matrix \
- --xhf data/headerxchr1 \
- --yhf data/headerychr1
-I:添加cworld的库,连接到cworld软件所在目录即可
-i:matrix 文件
--xhf:横坐标表头
--yhf:纵坐标表头
自制Header文件,横纵坐标可以相同,形如:
结果文件
- 2.4 扣除背景/计算z-scale
操作采用cworld的matrix2loess.pl
- #export PATH=/software/R/R-3.5.0/bin/:$PATH
- #export PATH=/software/bedtools/bedtools2-2.28.0/bin/:$PATH
- perl -I /software/cworld-dekker/ \
- /software/cworld-dekker/scripts/perl/matrix2loess.pl \
- -i example.addedHeaders.matrix.gz
结果文件以zScore.matrix.gz结尾
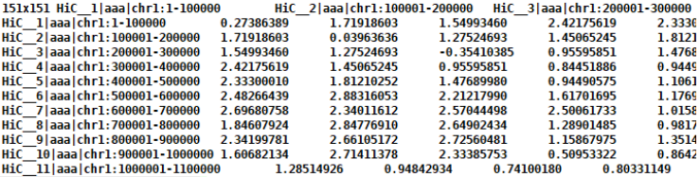
- 2.5 转换相关矩阵与PCA分析同时进行
采用cworld的matrix2EigenVectors.py,需要给一个example_gene.bed文件
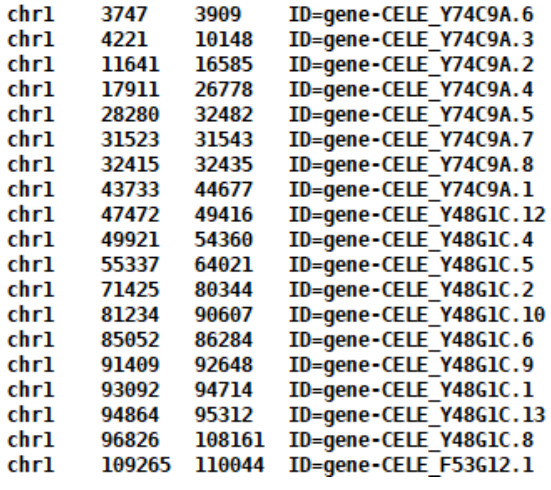
example_gene.bed
- python software/cworld-dekker/scripts/python/matrix2EigenVectors.py \
- -i example.addedHeaders.zScore.matrix.gz \
- -r data/example_gene.bed
结果文件以compartments结尾

compartments
另外生成的图片以compartments.png结尾
定义基因密度较高的区域为compartmentsA,反之为compartmentsB


