首先在Cistrome DB网站的首页(http://cistrome.org/db/#/),直接对你感兴趣的转录因子的ChIP-seq数据进行检索。网站提供了两种数据检索方式:关键词搜索和条件筛选。其中,关键词搜索区可以通过样本来源及因子搜索数据,同时可以在Options选项中对数据的质控指标进行初筛;条件筛选区可以通过直观的、按字母排序浏览和点选方式,选择你感兴趣的样本来源(各种细胞系、组织样本等)和Factor因子(转录因子、染色质调控因子、组蛋白修饰等)。这两种方式可以分别单独使用,也可以组合使用:
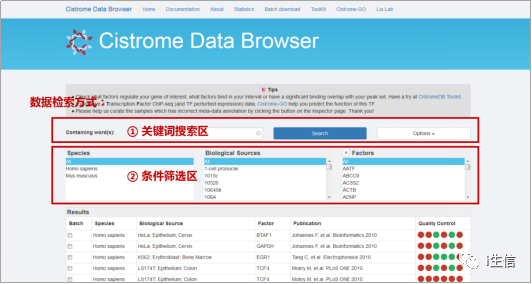
图片来源:Cistrome DB
做肿瘤相关研究的同学都清楚,肿瘤细胞中组蛋白的甲基化、乙酰化等表观遗传修饰可能会发生一定改变。而我想知道的是,在我研究的肝癌细胞HepG2中,乙酰化的组蛋白H3是否参与了我感兴趣的某基因的转录调控。因为不确定细胞系、组蛋白的拼写规则及表观遗传修饰的表示方法等,是否会对搜索结果产生影响,因此我首先尝试在关键词搜索区输入模糊关键词“liver”“H3”进行初筛。在使用一个不太熟悉的网站工具时,这种“模糊搜索法”也强烈推荐给大家使用,以防遗漏关键信息。
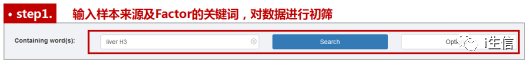
图片来源:Cistrome DB
随后,在条件筛选区,对搜索结果进行进一步筛选。这个数据库重点收录的是人类和小鼠的ChIP-seq数据,物种根据大家的研究需求选择即可。HepG2是人源细胞系,因此这里物种选择Homo,点选后样本来源框会发生相应改变。我们在其中找到HepG2,点选后就能在最后一项Factors选项中,查看数据库中包含的H3表观遗传修饰形式啦。我在其中翻看了一下这里包括乙酰化修饰形式的类型,有H3K9ac和H3K27ac等常见乙酰化修饰位点数据。
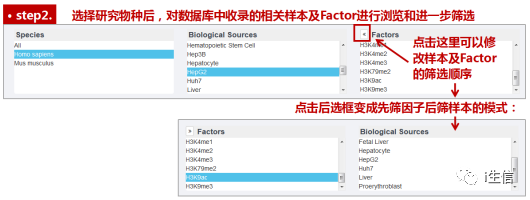
图片来源:Cistrome DB
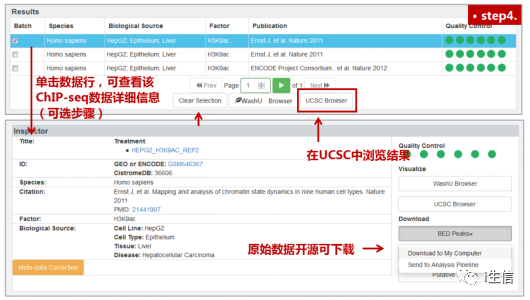
下面我们对这两种乙酰化修饰类型的组蛋白ChIP-seq数据进行一下查看和整理。分别点击H3K9ac和H3K27ac,在Results列表中查看搜索结果。一般情况下,细胞系的ChIP-seq数据质控指标优于组织样本,基本全部达标(全部为绿色标记),随意选择即可。而对于组织样本,要尽量选择绿点更多的数据。选中后直接在数据行前面打勾即可。可以一次性勾选多行数据,不用担心,重新搜索后之前勾选的内容也不会消失。
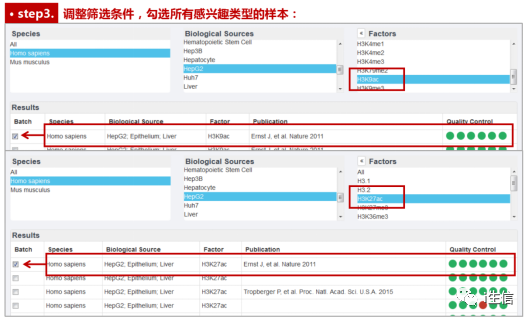
图片来源:Cistrome DB
单击数据行,可以直接在页面最下方浏览、下载或分析(方法见图.step5.1)样本信息。也可以在所有感兴趣的样本全部勾选完毕后,点击“UCSC Browser”选项跳转到UCSC基因组浏览器中,直观查看ChIP-seq结果,并对结果进行进一步处理(方法见图.step5.2)。
图片来源:Cistrome DB
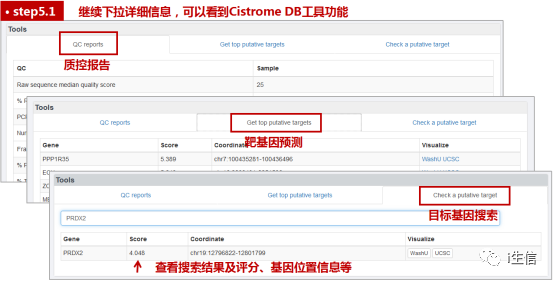
对于Cistrome DB的质控报告,一般关注唯一比对百分率、富集峰数量及覆盖率等信息;在靶基因预测选项卡中,可以找到这个样本中评分最高的前100个基因,即预测的核酸序列可能与目标蛋白结合最强烈的基因;最后的搜索靶基因功能,就是我要找的功能啦,这里可以搜索我感兴趣的基因,在选择的这个样本中,是否与该蛋白有互作,并可以查看评分信息。
图片来源:Cistrome DB
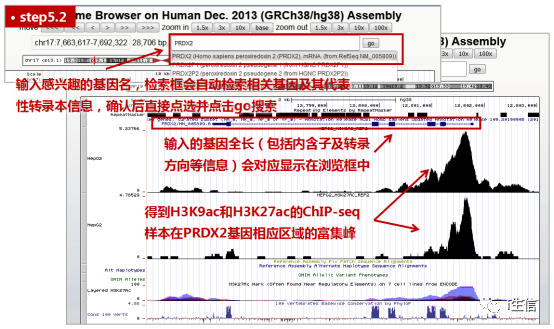
也可以跳过这步,更直观地在UCSC中查看结果信息。在UCSC的搜索框中直接搜索我感兴趣的目标基因的Symbol(或位置信息,注意和版本对应),确认并点选相应转录本信息后点击go,直接在页面下方查看目标基因区域的ChIP富集峰分布情况。页面上方的zoom in和zoom out可以相应倍数的放大缩小浏览框中包含的序列长度,前后箭头可以挪动浏览框中显示的序列位置(也可以直接用鼠标在浏览框中拖动)。大家可以根据自己的喜好进行调整。
图片来源:UCSC Genome Browser
结果真的是太明显了,无论是K9还是K27位点乙酰化的H3样本,在我们关注的这个基因的启动子区(一般认为是转录起始位点附近,至其上游不超过2000bp的区域。记得关注基因的转录方向,不要误判启动子区位置哦)都有个非常明显的富集峰。这不就说明,这个基因的启动子区和乙酰化组蛋白H3很可能有结合(调控)关系吗?下一步实验,我们就可以在这个峰的位置设计引物、开始充满自信与希望的ChIP实验验证啦!对了,上面这张图也蛮好看的,可以留着做个汇报数据或补充Figure呢!
再次强调,这样的方法也同样适用于转录因子、染色质调控因子等蛋白与目标基因结合情况的分析。比起基于序列的转录因子预测,这种基于ChIP实验的seq数据,在最后的实验验证中,阳性率会更高,提示作用也更强。但是,大数据毕竟是大数据,依然不能直接明确分子间的真实互作关系。


