人体由近 40 万亿个细胞组成,有许多不同类型。实验生物学的最新进展使探索单个细胞的遗传物质成为可能。随着单细胞基因组学这一新领域的诞生,科学家们现在可以探测人体内单个细胞的 DNA 和 RNA 。
单细胞基因组分析已经确定了人体内的新型细胞,发现了是什么使这些细胞彼此不同,以及不同类型的细胞如何对疾病或药物作出反应。单细胞基因组学也被证明是当前 COVID-19 大流行的关键,它可以识别易受感染的细胞并揭示感染患者免疫系统的变化。
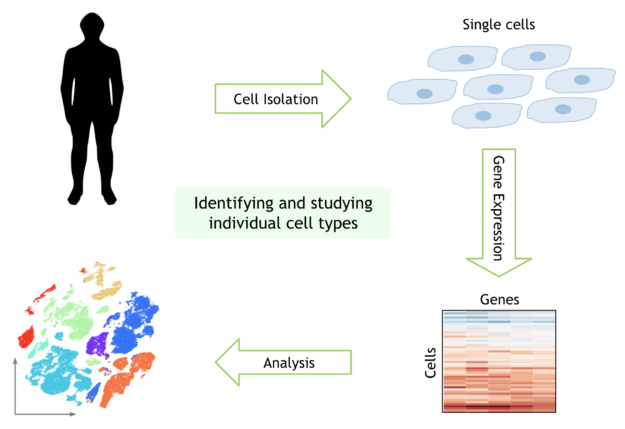
图 1 。单细胞 RNA 测序实验的工作流程。分离单个细胞并测量每个细胞的基因活性。具有相似基因活性的细胞聚集在一起以识别群体中的各种类型的细胞。
随着最近的实验对数百万个细胞进行测序,单细胞数据的可用性和数据集的大小也在不断增加。这种分析通常是探索性的,并从互动中得到进一步的好处——在更精细的尺度上识别不同类型的细胞,比较细胞类型并可视化它们之间的关系。当前的工作流仍然非常缓慢,这使得它们对于研究所需的交互分析来说是不可能的。
RAPIDS 是一套开源库,通过 GPU 加速的力量,可以加速端到端的数据科学工作流程。 RAPIDS 使得使用类似于 NumPy 、 pandas 和 scikit learn 的 Python api 对大型数据集执行交互式数据分析成为可能。
考虑执行单单元分析的典型工作流。这从一个矩阵开始,这个矩阵映射每个细胞中遇到的每个基因的数量。对数据进行预处理,滤除噪声,然后对数据进行归一化处理,得到每个细胞中每个人类基因的活性。在这一步中,机器学习也常用于纠正数据收集中的工件。接下来,在聚类和可视化之前执行维数缩减,以识别具有相似遗传活动的细胞簇。最后,你比较这些细胞群的遗传活动,以了解为什么不同类型的细胞表现和反应不同。
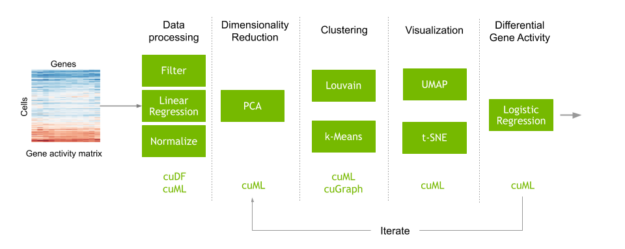
我们在 clara-parabricks/rapids-single-cell-examples GitHub repo 中发布了这个精确工作流的 GPU – 加速版本。 repo 包含一个示例 notebook ,它使用 RAPIDS 和 Scanpy 分析 70000 个人体肺细胞的数据集,以识别对 COVID-19 敏感的细胞。 Scanpy 是一个用于分析单细胞基因表达数据的工具包,提供了使用 RAPIDS 加速特定命令的选项。我们在回购中也有一个笔记本的 CPU 版本 以供比较。
例如,运行 UMAP 以使用 RAPIDS 可视化近 70000 个单元格需要以下命令:
- sc.tl.umap(adata, min_dist=umap_min_dist, spread=umap_spread, method='rapids')
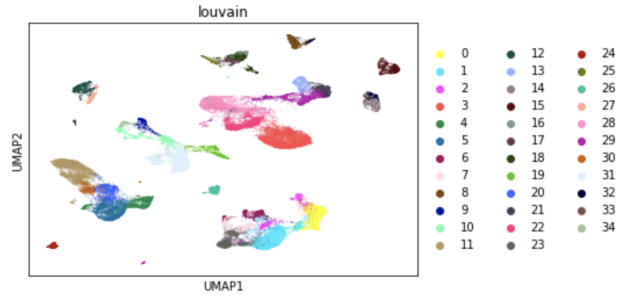
使用 RAPIDS 生成这个 UMAP 可视化需要 1 秒,而在 CPU 上则需要 80 秒。事实上, RAPIDS 可以加速整个单单元分析工作流程,甚至可以在大型数据集上进行交互式探索性数据分析。
| Instance | m5a.12xlarge | p3.2xlarge | Acceleration Factor |
| CPU/GPU type | Intel Xeon Platinum 8000, 48 vCPUs | V100-16GB | |
| Preprocessing | 311 | 84 | 4 |
| PCA | 18 | 3.4 | 5 |
| t-SNE | 208 | 2.2 | 95 |
| k-Means clustering | 31 | 0.4 | 78 |
| KNN | 25 | 6.1 | 4 |
| UMAP | 80 | 1 | 80 |
| Louvain clustering | 17 | 0.3 | 57 |
| Differential Gene Expression | 54 | 10.8 | 5 |
| End-to-end | 787 (13 Min) | 134 (2 Min) | 6 |
| Instance Price/hr ($) | 2.064 | 3.06 | |
| Total Run Cost ($) | 0.451 | 0.114 | 4 |
表 1 : CPU 运行时间, GPU 运行时间和 GPU 分析大约 70000 个人类肺细胞的每个步骤的加速度。所有时间都以秒为单位。
在 11 分钟内分析一百万个细胞
我们将我们的 RAPIDS 分析工作流程应用于现有最大的单细胞数据集之一, 100 万个小鼠脑细胞通过 10 倍基因组学测序。有关详细信息,请参阅 1M_brain_gpu_analysis_uvm.ipynb Jupyter 笔记本。
有了如此大的数据量,对 CPU 的分析变得不切实际地慢了下来;我们的端到端工作流在 awsm5a CPU 实例上运行了 3 个多小时。这使得交互式分析几乎不可能。另一方面,我们在这个更大的数据集上观察到了更高的 GPU 加速,并且能够在一个 GPU 上分析整个数据集。在 AWS 上运行 RAPIDS 分析也比 CPU 版本便宜 3 倍!
| AWS Instance | m5a.12xlarge | p3.8xlarge | Acceleration Factor |
| CPU/GPU type | Intel Xeon Platinum 8000, 48 vCPUs | V100-16GB | |
| Preprocessing | 4033 | 323 | 12.5 |
| PCA | 34 | 20.6 | 1.7 |
| t-SNE | 5417 | 41 | 132.1 |
| k-Means clustering | 106 | 2.1 | 50.5 |
| KNN | 585 | 53.4 | 11.0 |
| UMAP | 1751 | 20.3 | 86.3 |
| Louvain clustering | 597 | 2.5 | 238.8 |
| End-to-end | 13002 | 672.7 | 19.3 |
| Instance Price/hr ($) | 2.064 | 12.24 | |
| Total Run Cost ($) | 7.455 | 2.287 | 3.3 |
用于交互式单细胞分析的 GPU 功能单元浏览器
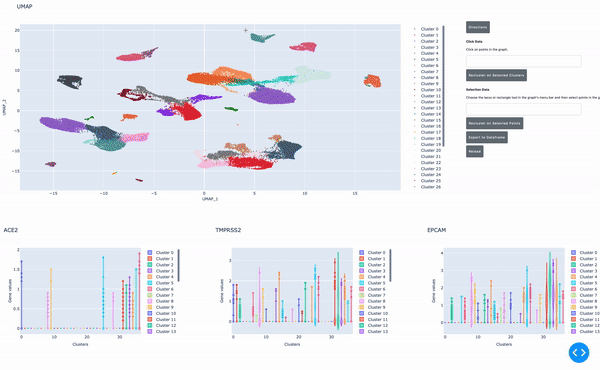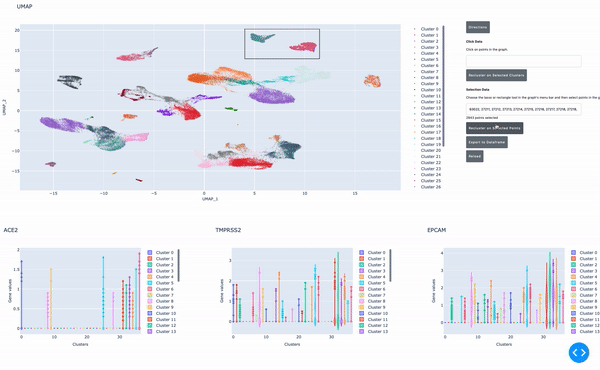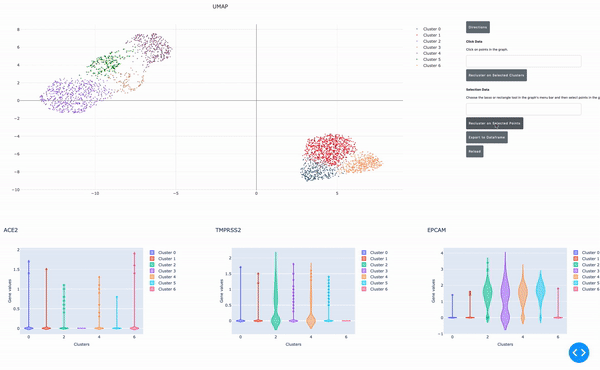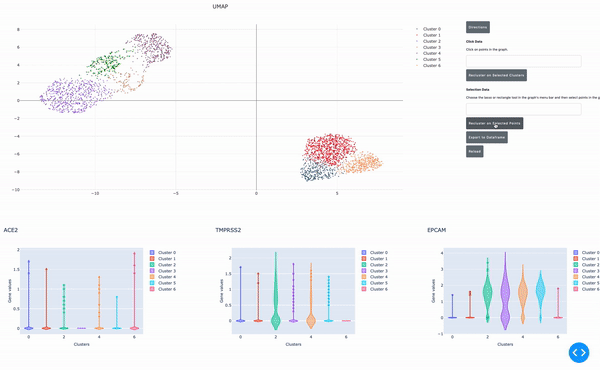
如前所述, RAPIDS 的数据分析速度使研究人员能够实时交互式地分析数据。我们开发了一个在 Jupyter 笔记本 中运行的、支持 GPU 的交互式小区浏览器,使这一过程更加简单。在这个单元格浏览器中,您可以可视化数据集中的所有单元格,并通过点击方法对数据执行聚类分析。使用 RAPIDS ,这些步骤可以实时运行。
在这篇文章中,我将向您展示如何轻松地选择一组细胞,并执行 UMAP 和 Louvain 聚类来识别这种细胞类型中的子种群。
结论
在这篇文章中,您看到了使用 RAPIDS 加速 GPUs 上的单细胞基因组分析是多么容易。使用 RAPIDS ,可以方便地实时交互地探索数据,对不同尺度的单元进行聚类,以及对具有不同参数的大型数据集进行重新分析。所有这些都有助于更快的科学发现。
除了涵盖的 API 之外, RAPIDS 还有一个大型的其他算法库,您会发现这些算法在您的工作中很有用。