同源序列比对是生物信息学普遍采用的分析方法之一,其中,编码蛋白质DNA序列比对最为常见,在比较基因组学、分子进化学、系统发育等领域具有重要的基础作用。为获取相应的比对结果,通常采用的方法是先比对后回译(back-translate),此方法比直接进行DNA序列比对更可靠、准确。然而,现有相关的工具每次只能处理一组同源序列,无法实现多组同源序列的比对工作。
前面的一篇文章介绍了用clustal进行批量序列比对的方法,请参考《 Clustal难道不能批量运行?》。Clustal只能单纯地比对蛋白序列或者核酸序列。要想将蛋白比对结果回译成核酸序列的比对结果,需要借用其他的工具或者自己写脚本(这里给大家推荐一个脚本PAL2NAL: http://www.bork.embl.de/pal2nal/)
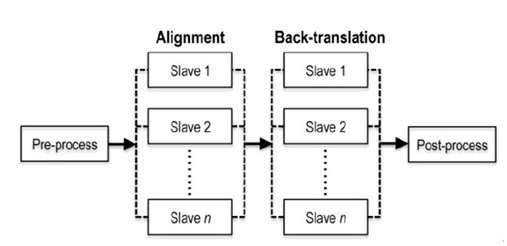
今天给大家推荐一款新的软件ParaAT。该软件相对于clustal+其他脚本的方法,自动化程度高,它自动先对蛋白进行比对,然后将蛋白序列结果回译成相应的核酸,实现多组同源编码蛋白质DNA序列的并行比对。ParaAT可大大降低运行时间,获得较好的并行加速比(speedup),因此,适合大规模、同源序列的比对工作。
该软件的计算方案如下:
ParaAT可在不同操作系统下运行,支持多种不同的输出格式,方便后续相关的生物信息学分析(诸如:用于检测自然选择压力的KaKs_Calculator,PAML等等)。
ParaAT下载地址:http://code.google.com/p/paraat/



1F
您好,我现在做kaks,想用kaks calculator计算。想请教一下,现有两个微生物基因组蛋白编码的核苷酸和氨基酸序列,接下来要怎么做呢,找直系同源单拷贝基因,在网上看到有人用InParanoid做,但是给的脚本是真核的,而且建议在unix下做,我的是win7的。请问,这个该怎么做?
谢谢!
祝好!