在现代分子进化研究中,根据现有生物基因或物种多样性来重建生物的进化史是一个非常重要的问题。一个可靠的系统发生的推断,将揭示出有关生物进化过程的顺序,有助于我们了解生物进化的历史和进化机制。
对于一个完整的进化树分析需要以下几个步骤:
⑴ 要对所分析的多序列目标进行比对(alignment)。
⑵ 要构建一个进化树(phyligenetic tree)。
构建进化树的算法主要分为两类:独立元素法(discrete character methods)和距离依靠法(distance methods)。
所谓独立元素法是指进化树的拓扑形状是由序列上的每个碱基/氨基酸的状态决定的(例如:一个序列上可能包含很多的酶切位点,而每个酶切位点的存在与否是由几个碱基的状态决定的,也就是说一个序列碱基的状态决定着它的酶切位点状态,当多个序列进行进化树分析时,进化树的拓扑形状也就由这些碱基的状态决定了)。
而距离依靠法是指进化树的拓扑形状由两两序列的进化距离决定的。进化树枝条的长度代表着进化距离。独立元素法包括最大简约性法(Maximum Parsimony methods)和最大可能性法(Maximum Likelihood methods);距离依靠法包括除权配对法(UPGMAM)和邻位相连法(Neighbor-joining)。
⑶ 对进化树进行评估,主要采用Bootstraping法。
进化树的构建是一个统计学问题,我们所构建出来的进化树只是对真实的进化关系的评估或者模拟。如果我们采用了一个适当的方法,那么所构建的进化树就会接近真实的“进化树”。模拟的进化树需要一种数学方法来对其进行评估。不同的算法有不同的适用目标。
一般来说,最大简约性法适用于符合以下条件的多序列:
i 所要比较的序列的碱基差别小,
ii 对于序列上的每一个碱基有近似相等的变异率,
iii 没有过多的颠换/转换的倾向,
iv 所检验的序列的碱基数目较多(大于几千个碱基);
用最大可能性法分析序列则不需以上的诸多条件,但是此种方法计算极其耗时。如果分析的序列较多,有可能要花上几天的时间才能计算完毕。UPGMAM(Unweighted pair group method with arithmetic mean)假设在进化过程中所有核苷酸/氨基酸都有相同的变异率,也就是存在着一个分子钟。这种算法得到的进化树相对来说不是很准确,现在已经很少使用。邻位相连法是一个经常被使用的算法,它构建的进化树相对准确,而且计算快捷。其缺点是序列上的所有位点都被同等对待,而且,所分析的序列的进化距离不能太大。另外,需要特别指出的是对于一些特定多序列对象来说可能没有任何一个现存算法非常适合它。
CLUSTALX和PHYLIP软件能够实现上述的建树步骤。CLUSTALX是Windows界面下的多重序列比对软件。PHYLIP是多个软件的压缩包,功能极其强大,主要包括五个方面的功能软件:
i,DNA和蛋白质序列数据的分析软件。
ii,序列数据转变成距离数据后,对距离数据分析的软件。
iii,对基因频率和连续的元素分析的软件。
iv,把序列的每个碱基/氨基酸独立看待(碱基/氨基酸只有0和1的状态)时,对序列进行分析的软件。
v,按照DOLLO简约性算法对序列进行分析的软件。
vi,绘制和修改进化树的软件。
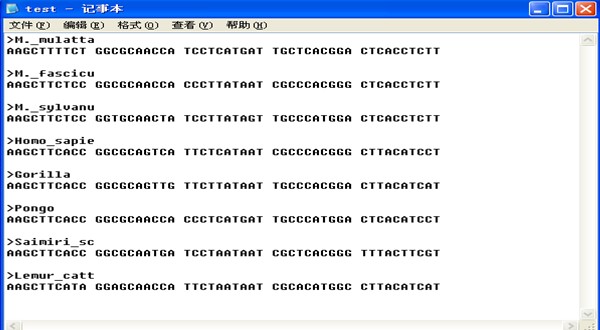
测试数据
[code lang="plain"]
M._mulatta AAGCTTTTCT GGCGCAACCA TCCTCATGAT TGCTCACGGA CTCACCTCTT
M._fascicu AAGCTTCTCC GGCGCAACCA CCCTTATAAT CGCCCACGGG CTCACCTCTT
M._sylvanu AAGCTTCTCC GGTGCAACTA TCCTTATAGT TGCCCATGGA CTCACCTCTT
Homo_sapie AAGCTTCACC GGCGCAGTCA TTCTCATAAT CGCCCACGGG CTTACATCCT
Gorilla AAGCTTCACC GGCGCAGTTG TTCTTATAAT TGCCCACGGA CTTACATCAT
Pongo AAGCTTCACC GGCGCAACCA CCCTCATGAT TGCCCATGGA CTCACATCCT
Saimiri_sc AAGCTTCACC GGCGCAATGA TCCTAATAAT CGCTCACGGG TTTACTTCGT
Lemur_catt AAGCTTCATA GGAGCAACCA TTCTAATAAT CGCACATGGC CTTACATCAT
[/code]
分析步骤
一、用CLUSTALX软件对已知DNA序列做多序列比对。
操作步骤:
1、以FASTA格式准备8个DNA序列test.seq(或txt)文件。
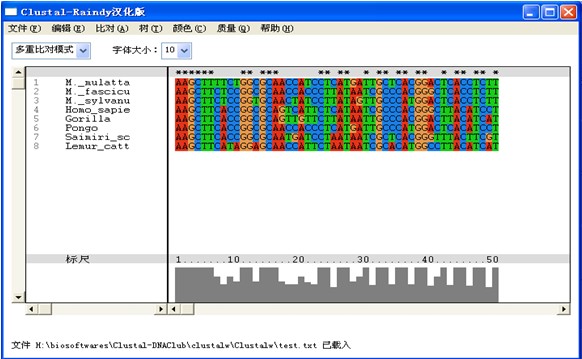
2、双击进入CLUSTALX程序,点FILE进入LOAD SEQUENCE,打开test.seq(或txt)文件。
3、点ALIGNMENT,在默认alignment parameters下,点击Do complete Alignment 。在新出现的窗口中点击ALIGN进行比对,这时输出两个文件(默认输出文件格式为Clustal格式):比对文件test.aln和向导树文件test.dnd。
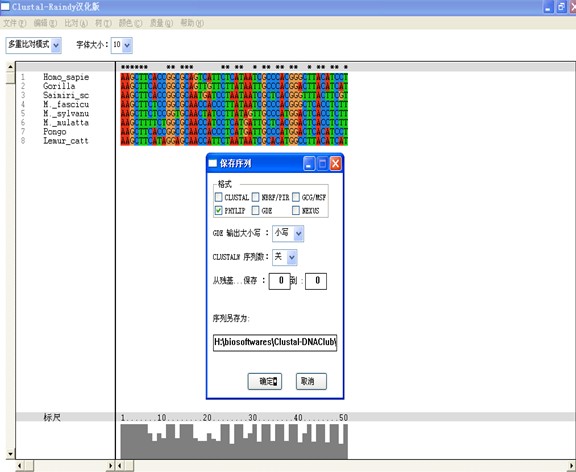
4、点FILE进入Save sequence as,在format 框中选PHYLIP,文件在PHYLIP软件目录下以test.phy存在,点击OK。
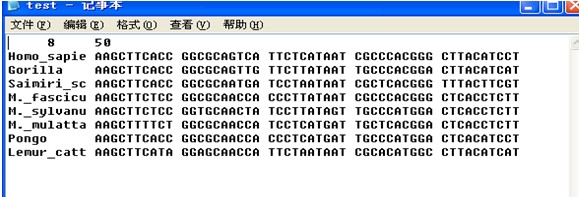
5、将PHYLIP软件目录下的test.phy文件拷贝到EXE文件夹中。用计事本方式打开的test.phy文件的部分序列如下:
图中的8和50分别表示8个序列和每个序列有50个碱基。
二、用PHYLIP软件推导进化树。
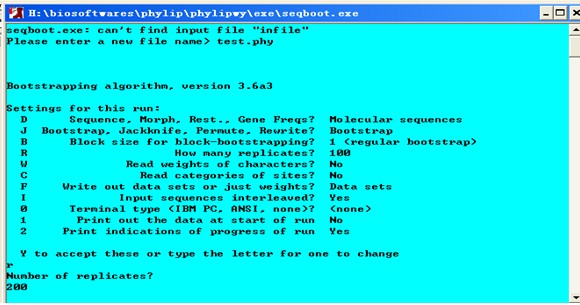
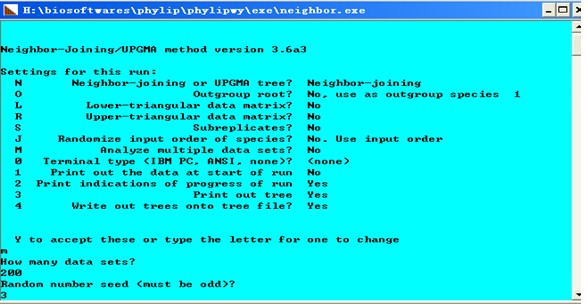
1、进入EXE文件夹,点击SEQBOOT软件输入test.phy文件名,回车。
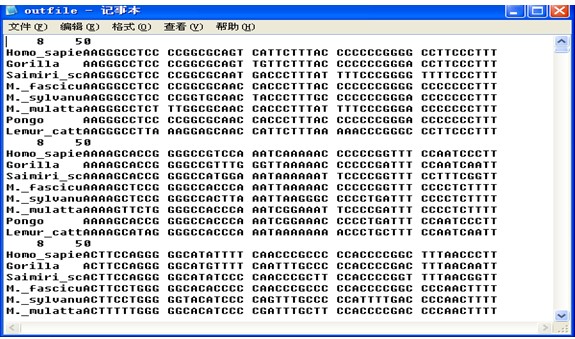
图中的D、J、R、I、O、1、2代表可选择的选项,键入这些字母,程序的条件就会发生改变。D选项无须改变。J选项有三种条件可以选择,分别是Bootstrap、Jackknife和Permute。文章上面提到用Bootstraping法对进化树进行评估,所谓Bootstraping法就是从整个序列的碱基(氨基酸)中任意选取一半,剩下的一半序列随机补齐组成一个新的序列。这样,一个序列就可以变成了许多序列。一个多序列组也就可以变成许多个多序列组。根据某种算法(最大简约性法、最大可能性法、除权配对法或邻位相连法)每个多序列组都可以生成一个进化树。将生成的许多进化树进行比较,按照多数规则(majority-rule)我们就会得到一个最“逼真”的进化树。Jackknife则是另外一种随机选取序列的方法。它与Bootstrap法的区别是不将剩下的一半序列补齐,只生成一个缩短了一半的新序列。Permute是另外一种取样方法,其目的与Bootstrap和Jackknife法不同,这里不再介绍。R选项让使用者输入republicate的数目。所谓republicate就是用Bootstrap法生成的一个多序列组。根据多序列中所含的序列的数目的不同可以选取不同的republicate,此处选200,输入Y确认参数并在Random number seed (must be odd) ?的下面输入一个奇数(比如3)。当我们设置好条件后按回车,程序开始运行,并在EXE文件夹中产生一个文件outfile,Outfile用记事本打开如下:
这个文件包括了200个republicate。
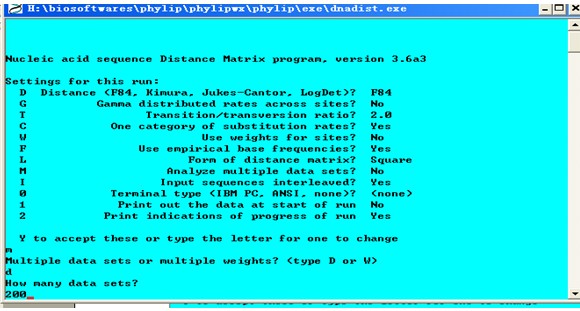
2、文件outfile改为infile。点击DNADIST程序。选项M是输入刚才设置的republicate的数目,输入D选择data sets,输入200。
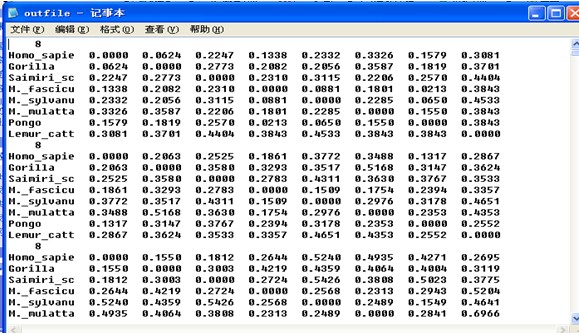
设置好条件后,输入Y确认参数。程序开始运行,并在EXE文件夹中产生outfile,部分内容如下:
将outfile文件名改为infile,为避免与原先infile文件重复,将 原先文件名改为infile1。
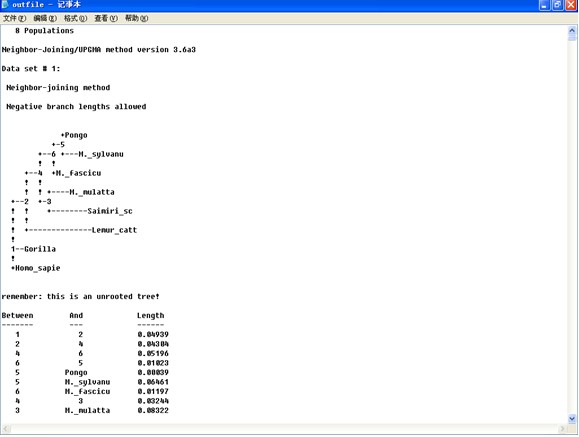
3、EXE文件夹中选择通过距离矩阵推测进化树的算法,点击NEIGHBOR程序。输入M更改参数,输入D选择data sets。输入200。输入奇数种子3。
输Y确认参数。程序开始运行,并在EXE文件夹中产生outfile和outtree两个结果输出。outtree文件是一个树文件,可以用treeview等软件打开。outfile是一个分析结果的输出报告,包括了树和其他一些分析报告,可以用记事本直接打开。部分内容如下:
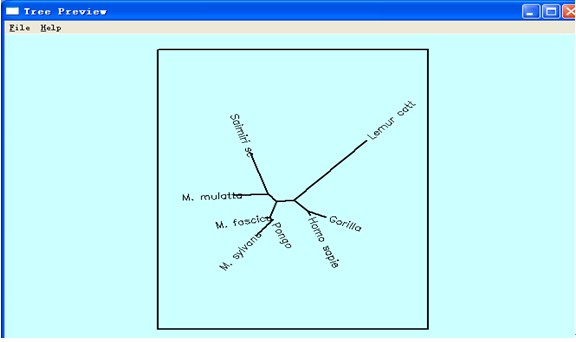
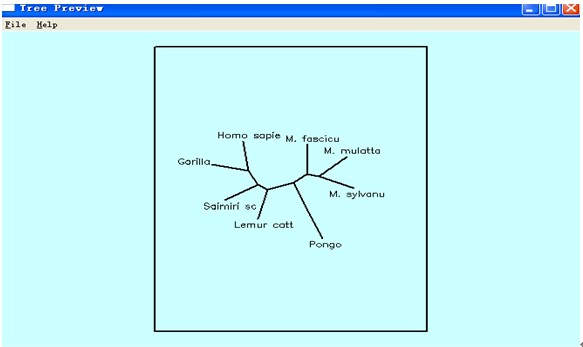
4、将outtree文件名改为intree,点击DRAWTREE程序,输入font1文件名,作为参数。输Y确认参数。程序开始运行,并出现Tree Preview图。
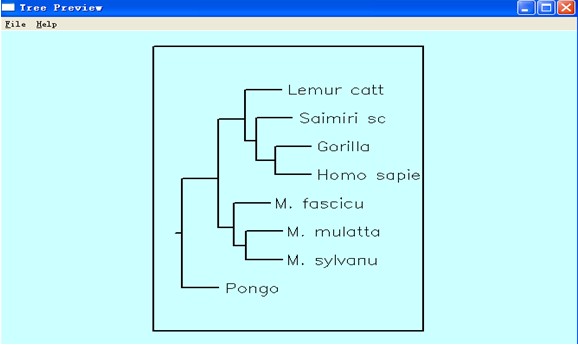
5、点击DRAWGRAM程序,输入font1文件名,作为参数。输Y确认参数。程序开始运行,并出现Tree Preview图。
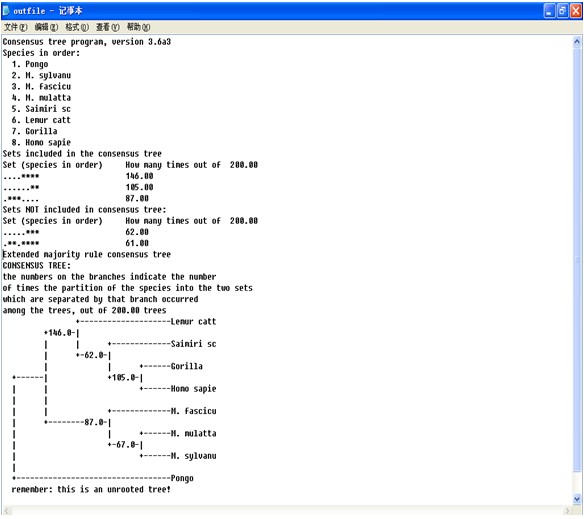
6、将EXE文件夹中的outfile文件名改为outfile1,以避免被新生成的outfile 文件覆盖。点击CONSENSE程序。输入Y确认设置。EXE文件夹中新生成outfile和outtree。Outfile文件用记事本打开,内容如下:
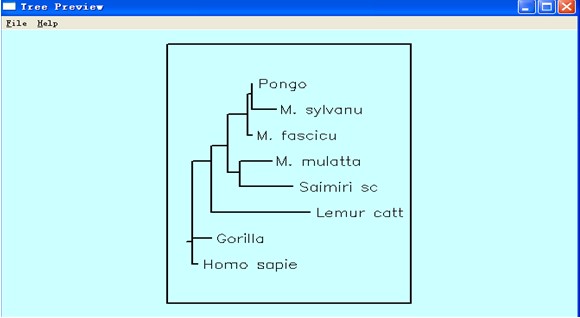
7、将EXE文件夹中的intree文件名改为intree1,将outtree改intree。点击DRAWTREE程序,输入font1文件名,作为参数。输Y确认参数。程序开始运行,并出现Tree Preview图。
8、点击DRAWGRAM程序,输入font1文件名,作为参数。输Y确认参数。程序开始运行,并出现Tree Preview图。