一、蛋白质的结构与功能预测
蛋白质结构与功能的研究已有相当长的历史,由于其复杂性,对其结构与功能的预测不论是方法论还是基础理论方面均较复杂。统计学方法曾被成功地应用于蛋白质二级结构预测中,如Chou和Fasman提出的经验参数法便是最突出的例子。该方法统计分析了各种氨基酸的二级结构分布特征,得出相应参数(Pа,Pβ和Pt)并用于预测。
二、蛋白质结构及其数据库
一般情况下,蛋白质的结构分为4个层次:
- 初级结构--蛋白质序列;
- 二级结构--а-螺旋和β-折叠片(β-sheets)模式;
- 三级结构--残基在空间的布局;
- 四级结构--蛋白质之间的互作。
近年来,另一个介于二级和三级结构之间的蛋白质结构层次--所谓蛋白质折叠(fold)已被证明非常有用。"fold"描述的是二级结构元素的混合组合方式。 根据序列或多序列列线预测蛋白质二级结构的技术已相对比较成熟,但三级结构的预测则相当困难。往往对于三级结构预测,只能通过与已知结构蛋白序列同源性比对来完成。
已有不少相关数据库被建立起来用于蛋白质结构预测。这一方法已是目前进行三级结构预测的最准确方法。但是这一方法并不总是奏效,因为大约有80%的已知蛋白质序列找不到与之相似的已知结构的蛋白质序列。
近年来,一些新方法被提出,这些方法可以不通过相似性比对来预测序列结构。
三、二级结构预测
已有大量有关根据序列预测蛋白质二级结构的文献资料,这些资料可大致分为二类: 一是有关根据单一序列预测二级结构; 二是有关根据多序列列线预测二级结构。
直到最近为止,二级结构预测才不被认为具有很高的随机性。大多数预测算法均是依据单一序列。即使是最著名的一些算法(如Chou-Fasman算法和GOR算法)也只有约60%的预测准确率,而对于一些特定的结构,如那些富含β-折叠片的结构,这些算法难以预测成功。
预测失败的原因主要是单一序列所提供的信息只是残基的顺序而没有其空间分布的信息。两个方面的研究进展改变了这一状况:一是认识到多序列列线可被用于改进预测能力。多序列列线可被视为诱变遗传学试验中的自然突变状况,其对序列上单一位点变异的分析的确提供了该位点在蛋白质三级结构中的信息;二是神经网络已开始被用于根据序列预测结构。
目前已有这样一个共识,即在有大量、高质量的多序列列线结果的情况下,蛋白质二级结构的预测将非常准确--通常准确率比以单一序列预测提高10%。
一些文献表明,一些程序(诸如PHD)预测的准确率达到了目前最高水平。 PHD (http://www.embl-heidelberg.de/predictprotein/predictprotein.html) 提供了从二级结构预测到折叠(fold)识别等一系列功能。
四、三级结构预测
比对数据库中已知结构的序列是预测未知序列三级结构的主要方法。多种途径可进行以上这种比对。最容易是使用BLASTP程序比对NRL-3D或SCOP数据库中的序列。如果发现超过100个碱基长度且有远高于40%序列相同率的匹配序列,则未知序列蛋白与该匹配序列蛋白将有非常相似的结构。
在这种情况下,同源性建模(homology modeling)在预测该未知蛋白精细结构方面会发挥非常大的作用。在序列相同率为25%~40%时,两条蛋白质将具有相同的折叠,但这时同源性建模将变得更加困难和不准确。
如果在比对NRL-3D数据库时没有发现匹配序列,接下去可试试HSSP数据库。这样做的一条最方便捷径是用Blast或FASTA法搜索蛋白质序列库(如SWISS-PROT、TREMBL或PIR),然后利用诸如SRS等工具去检索任何超过25%序列相同率的匹配序列,如果这些匹配序列在HSSP数据库中存在,则在该序列的注释(annotation)"DR"栏中将有说明。
如果未知蛋白质序列与某一HSSP数据库序列有明显大于25%的序列相同率,则有把握地假定未知序列至少有与HSSP序列相同的蛋白质折叠模式。
目前,NRL-3D和HSSP数据库的记录数量可以保证20%的蛋白质序列将找到已知结构的同源序列。
总的来说,同源性建模需要专业分子建模方法和分子图象资源的辅助才能进行。不妨到Swiss-Model网站(http://expasy.hcuge.ch/swissmod/SWISS-MODEL.html)看看。
Swiss-Model是一个蛋白质自动建模服务器,使用者可以直接发送一条序列或使用者自己完成的列线结果给该服务器用于同源性建模。
近年蛋白质结构研究的最主要进展之一,是有关"串线"(threading)算法和折叠识别。这些技术可以在不存在已知结构同源蛋白质序列的情况下,预测所有可能的蛋白质结构。
"这个未知蛋白序列会是什么结构呢?"我们也可以这样问:"我已经观察了已知结构蛋白质的各种折叠方式,未知序列是否会象这些已知结构中的某一个一样折叠呢?"第一个问题涉及几十亿种可能结构的搜索,而第二个问题涉及的是少于1000种结构的搜索。
特定的蛋白质折叠被一而再,再而三地观察到--大部分新的经晶体衍射的蛋白将会与我们已知的折叠相关,这些过程使预测的成功机率不断提高。
在串联算法中,未知序列以合适的方式被"串"到一个数据库某一折叠模板,然后计算该序列的能(energy);在该序列与数据库中所有的折叠模板均"串"好后,可以进行计分比对,决定那些匹配达到了显著。
折叠的识别技术目前还不是特别可靠的技术,只有在序列相同比率在30%~50%时,才有可能获得准确的估计。相关程序的结果也相当粗糙,大多数情况下难以作为同源性建模研究的依据。但是它是大多数蛋白质结构预测信息唯一可利用的工具。
一些相关应用程序:
TOPITS(http://www.embl-heidelberg.de/predictprotein/predictprotein.html、)
frsvr(http://www.mbi.ucla.edu/people/frsvr/frsvr.html)、
123D(http://www_lmmb.ncifcrf.gov/~nicka/123D.html、)
THREADER和THREADER2(http://globin.bio.warwick.ac.uk/~jones/threader.html)
ProFIT(http://lore.came.sbg.ac.at/Extern/software/Profit/profit.html)
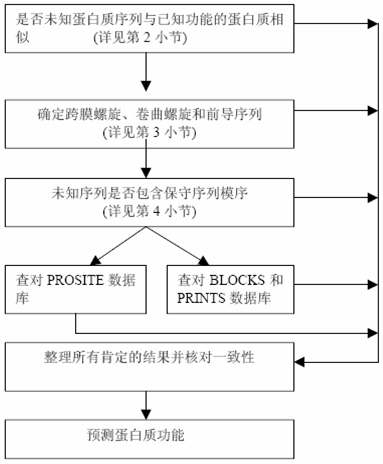
五、根据序列预测功能的一般过程
如果序列重叠群(contig)包含有蛋白质编码区,则接下来的分析任务是确定表达产物--蛋白质的功能。蛋白质的许多特性可直接从序列上分析获得,如疏水性,它可以用于预测序列是否跨膜螺旋(transmenbrane helix)或是前导序列(leader sequence)。但是,总的来说,我们根据序列预测蛋白质功能的唯一方法是通过数据库搜寻,比较该蛋白是否与已知功能的蛋白质相似。有2条主要途径可以进行上述的比较分析:
①比较未知蛋白序列与已知蛋白质序列的相似性;
②查找未知蛋白中是否包含与特定蛋白质家族或功能域有关的亚序列或保守区段
六、通过比对数据库相似序列确定功能
具有相似序列的蛋白质具有相似的功能。因此,最可靠的确定蛋白质功能的方法是进行数据库的相似性搜索。一个显著的匹配应至少有25%的相同序列和超过80个氨基酸的区段。 已有不少种类的数据库搜索工具,它们或者搜索速度慢,但灵敏;或者快速,但不灵敏。快速搜索工具(如BLASTP)很容易发现匹配良好的序列,所以没有必要再运行更花时的工具(如FASTA、BLITZ);只有在诸如BLASTP不能发现显著的匹配序列时,这些工具才被使用。
所以,一般的策略是首先进行Blast检索,如果不能提供相关结果,运行FASTA;如果FASTA也不能得到有关蛋白质功能的线索,最后可选用完全根据Smith-Waterman算法设计的搜索程序,例如BLITZ(www.ebi.ac.uk/searches/blitz.html)。 BLITZ不做近似估计(BLAST和FASTA根据Smith-Waterman算法做近似估计),所以很花时,但非常灵敏。
通常诸如BLITZ的程序能够发现超过几百个残基但序列相同比率低于20~25%的匹配,这些匹配可能达到显著,但会被那些应用近似估计的程序错过。还应注意计分矩阵(scoring matrix)的重要性。
选用不同的计分矩阵有不少重要原因:首先,选用的矩阵必须与匹配水平相一致,例如,PAM250应用于远距离匹配(<25%相同比率),PAM40应用于不很相近的蛋白质序列,而BLOSUM62是一个通用矩阵;第二,使用不同矩阵,可以发现始终出现的匹配序列,这是一条减少误差的办法。 除了选用不同的计分矩阵,同样可以考虑选用不同的数据库。通常可以使用的数据库是无冗余蛋白序列数据库SWISS-PROT和PDB。其它一些数据库也可以试试,如可用BLASTP搜索复合蛋白质序列库OWL(www.biochem.ucl.ac.uk/bsm/dbbrowser/OWL/owl_blast.html)
七、序列特性:疏水性、跨膜螺旋等
许多功能可直接从蛋白质序列预测出来。例如,疏水性信息可被用于跨膜螺旋的预测。还有不少小的模序(motif)是细胞用于特定细胞区室(cell compartment)蛋白质的定向。网上有大量数据资源帮助我们利用这些特性预测蛋白质功能。
疏水性信息可用 ExPASy(http://expasy.hcuge.ch/egibin/protscal.pl)的ProtScale程序创建并演示。这是一个很有用的工具,它能计算超过50种蛋白质的特性。程序的输入即可通过输入框将序列粘贴进去,也可输入SWISS-PROT的记录号。
仅一项需要额外设定的参数是输入框的宽度,该参数将指示系统每次运行计算和显示的残基数,其缺省值为9。如果想考虑跨膜螺旋特性,该参数设置应为20,因为一个跨膜螺旋通常有20个氨基酸长度 。
有多种方法可以预测序列的跨膜螺旋。最简单的方法是通过查找包含有20个疏水残基的区段,一些更复杂、更准确的算法不仅可以预测跨膜螺旋的位置,还能确定其在膜上的方向。这些方法都依赖于一系列已知跨膜螺旋特性的研究结果。
TMbase是一个自然发生的跨膜螺旋数据库(http://ulrec3.unil.ch/tmbase/TMBASE_doc.html)。
相关的一些程序:
TMPRED (http://ulrec3.unil.ch/software/TMPRED-form.html)、
PHDhtm (www.embl_heidelberg.de/services/sander/predictprotein/predictprotein.html)、
TMAP (http://www.embl-heidelberg.de/tmap/tmap/tmap_sin.html)
MEMSAT (ftp.biochem.ucl.ac.uk)。
这些程序将使用了不同的统计模型,总体上,预测准确率在80~95%左右。跨膜螺旋是可以根据序列数据比较准确预测的蛋白质特性之一
预测前导序列或特殊区室靶蛋白信号的程序:
SignalP (http://www.cbs.dtu.dk/services/SignalP)和PSORT (http://psort.nibbac.jp/form.html)。
另一个可从序列中确定的功能模序是卷曲(coil)螺旋。
在这一结构中,二个螺旋由于疏水作用而缠绕在一起形成非常稳定的结构。相关的2个程序:
COILS (http://ulrec3.unil.ch/software/COILS_form.html)
Paircoil (http://ostrich.lcs.mit.edu/cgi-bin/score)