汤海明1, 陈红1, 张静1, 任景怡1, 许宁2
1. 北京大学人民医院心内科, 北京 100044; 2. 瑞典卡罗林斯卡医学院医学系, 斯德哥尔摩 17176
摘要:
MicroRNAs(miRNAs)是一类在进化上高度保守的非编码小分子单链RNA(~22nt),在基因转录后调控中发挥至关重要的作用。越来越多的证据表明,miRNAs参与很多重要的生理和病理过程,例如发育、器官形成、调亡、细胞增殖、肿瘤发生等。近年来飞速发展的新一代测序技术在miRNA检测方面具有重要的应用。文章简要介绍了新一代测序技术3大平台的基本步骤和原理,测序数据的生物信息学分析方法以及新一代测序技术在miRNA方向的主要应用。相比于传统的miRNA检测方法,新一代测序技术具有通量高、对遗传物质检测完全且准确度高,可重复性好等优点,在探索新miRNA、miRNA互补链、miRNA编辑、miRNA异构体检测以及miRNA靶基因检测等方面具有巨大优势。随着新一代测序技术的不断发展,测序成本不断降低,在未来几年,新一代测序技术的使用率或将大大增加。新一代测序技术的不断应用将进一步促进人类对于miRNA在各种生理病理过程中的功能和调控的认识。
Abstract: MicroRNAs (miRNAs) are a class of ~22nt long non-coding RNAs. They are evolutionarily conserved and play essential roles in the regulation of post-transcriptional gene expression. The rapidly developing next generation se quencing (NGS) has important applications in miRNA detection. This review is focused on the mechanism of three NGS platforms and their applications in miRNA detection. In contrast to traditional methods, NGS has major advantages: high throughput, precise, accurate, and repeatable. Its application includes new miRNAs exploration, detection of miRNA*, miRNA editing, and isomiR and target mRNA detection. As NGS develops, the cost of sequencing is declining which makes it possible for NGS to be widely used in the coming years. Next generation sequencing will greatly promote re searches of miRNAs.
MicroRNAs是一类在进化上高度保守的非编码小分子单链RNA(~22nt),在基因表达调控中发挥至关重要的作用。1993年,Lee等[1]在秀丽隐杆线虫(Caenorhabditis elegans)中发现了一个可时序调控胚胎后期发育的基因lin-4。2000年Reinhart等[2]发现了具有转录后调节功能的小分子RNAlet-7。至2001年,研究者在线虫(C.elegans)、果蝇(D.melanogaster)和人体中发现了近百个这样的小RNA分子[3],国际上统一将这类小RNA正式命名为microRNA(miRNA),其研究也成为新的热点。随着新一代测序技术的发展,miRNA发现的过程大大提速,截至2011年miRbase已收录140个物种的15000条miRNAs基因位点和17000条不同的miRNAs序列[4]。每一个miRNA可以调控多个靶基因,反之,1个靶基因也可以同时被多个miRNAs调节。因此,据保守估计,约60%~70%的人类蛋白编码基因受到miRNAs的调控[5]。越来越多的证据表明,miRNAs参与很多重要的生理和病理过程,例如发育、器官形成、凋亡、细胞增殖、肿瘤发生等。
MiRNAs编码基因以单拷贝、多拷贝或基因簇等形式存在于基因组中,通常位于基因间或内含子区域。在细胞核内,RNA聚合酶从miRNA编码基因转录出比较长的miRNA初级转录本(pri-miRNA)。随后,pri-miRNA由属于RNA酶家族的Drosha和DGCR8组成的酶复合体剪切成长约65~70nt、带有茎环结构的miRNA前体(precursormiRNA,pre-miRNA)。Pre-miRNA通过Exportin-5由胞核转运至胞浆。在胞浆中,pre-miRNA被同属RNA酶三家族的Dicer酶识别并处理,形成长度约22nt的双链RNA(miRNA:miRNA*)。其中1条链很快被降解,另1条链则进入RNA诱导沉默复合体(RNA-induced silencing complex,RISC),成为成熟的miRNA。与RISC结合的miRNA,通过特异的碱基配对方式结合到靶基因mRNA的3′端非翻译区(Untranslatedregion,UTR),诱导靶mRNA的降解或抑制其翻译成蛋白或两者兼而有之,从而在转录后水平下调靶基因的表达。值得一提的是,miRNA的序列与靶mRNA的3′UTR区是不完全互补配对的,其中miRNA的“种子”区(Seed region,即miRNA5′端第2至第8个核苷酸)与靶位点序列的高度或完全配对是最为重要的,miRNA的其余序列则在与靶基因的结合过程中起辅助作用。
由于miRNA在转录后基因调节中发挥的巨大作用,快速获取特定样品的miRNA表达谱,对于探索特定生理及病理过程中发挥作用的miRNA及其作用机制十分必要。近年来发展的新一代测序技术采用全基因组测序方法,大大增加了新miRNA的检出速度[4]。并且除了获取已知miRNA的表达谱之外,新一代测序技术在miRNA互补链[6]、miRNA编辑[7]、miRNA异构体[8]检测及miRNA靶基因检测[9]方面具有新的应用。与传统的RT-PCR技术、Northern印记技术及微阵列芯片技术相比,它具有显著的优势。本文就新一代测序技术在miRNA检测方面的基本原理和应用作一综述。
1 新一代测序技术
美国ABI公司于1987年推出的基于Sanger法的第一代测序技术,在人类基因组图谱的绘制中发挥了重要的作用。自2005年发展起来的新一代测序平台取得了突破性的进展,使得测序效率大大增加,并且极大地降低了测序成本。运用新一代测序技术可以从基因组水平、转录组水平、蛋白质组水平等多个角度进行遗传物质的全面解码。新一代测序平台能够对样品中包括miRNA在内的小RNA分子进行高通量测序并定量,是很吸引人的研究方向。
1.1原理和步骤
新一代测序最常使用的3种平台是Illumina的基因组分析仪[10]、Roche454基因组测序仪[11]以及ABLifeTechnologies的SOLiD系统[12]。
1.1.1 Illumina 基因组分析仪
使用桥式PCR进行扩增。单链DNA两端加上非对称的接头,并固定在芯片表面形成寡核苷酸桥。芯片置于流通池内,其中有8个独立的道,每条道上都可固定数百万PCR产生的扩增产物簇。向扩增产物中加入测序引物,不同荧光基团标记的4种核苷酸和DNA聚合酶开始测序。封闭3′端使每次反应只能加入一个核苷酸。在采集荧光图像,获取新加入的核苷酸信息之后,3′端被打开,进行下一循环。该循环可进行50次,获取50碱基长度的DNA序列[10]。
1.1.2 Roche454 基因组测序仪
利用微乳滴PCR进行扩增。将水溶液与油混合,形成油包水结构的微乳滴。每个乳滴就是只包含一个微球及PCR试剂的微反应器。PCR在每一微球上单独进行,PCR的扩增产物也与微球结合。富集微球,并将其转移到微孔板上,进行焦磷酸测序。三磷酸核苷与DNA链结合释放焦磷酸,再通过ATP硫酰化酶和荧光素酶的一系列级联反应释放光信号。加入一种核苷酸,记录每一微孔之中光信号有无释放,从而确定该核苷酸是否与当前位点互补。焦磷酸测序读长可达400~500碱基[11]。
1.1.3 AB Life Technologies的 SOLiD 系统
SOLiD系统也采用微球和微乳滴的方法。在PCR扩增完成后,微球被结合到反应板上,采用连接法测序。连接反应的底物是8碱基长的探针,探针的5′端标记有荧光,3′端1~2位碱基对与5′端荧光信号的颜色对应。1次SOLiD测序共有5轮,每轮测序又由多个连接反应组成。第1轮的第1次连接反应将掺入1条探针,测序仪记录下反映该条探针3′端1~2位碱基信息的荧光信号,随后除去6~8位碱基及荧光基团,获得1~2位的颜色信息。以此类推,第2次连接反应得到6~7位的颜色信息,第3次连接反应得到11~12位颜色信息。多次连接后,开始第2轮测序。由于第2轮测序的引物比第1轮前移1位,这轮测序将得到0~1位、5~6位、10~11位的颜色信息,5轮测序过后,就可得到所有位置的颜色信息,并推断出相应的碱基序列[12]。
1.1.4 新一代测序平台进行miRNA 检测的步骤
使用新一代测序平台对miRNA进行检测的基本步骤主要包括:(1)构建DNA模板文库,从总RNA中分离纯化出20~30nt的小RNA后,使用T4连接酶分别在miRNA的5′端和3′端连上接头序列,进行RT-PCR得到70~80bp的DNA片段;(2)将所得单链模板文库,固定在平面或是微球的表面;(3)通过桥式PCR、微乳滴PCR或原位成簇对数据进行扩增;(4)采集并记录PCR循环中的光学事件;(5)对产生的阵列图像进行时序分析,获得DNA片段的序列。
1.2 各测序技术平台对比及选择
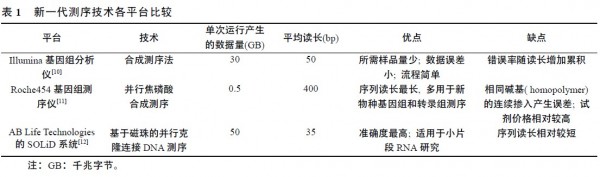
Roche454基因组测序仪以其独特的测序读长长的优势,在新物种基因组和转录组测序中占据主要地位。焦磷酸测序由于没有终止元件来阻止连续掺入,所以在一连串相同碱基如AAAAA的情况下,相同碱基的个数只能通过信号强度进行推断,由此造成插入/缺失误差。Roche454基因组测序仪的另一个缺点是焦磷酸检测依赖于一系列酶,试剂价格相对较高。AB Life Technologies的SOLiD系统在测序过程中每个碱基在两个连接反应中分别被测定,即单个碱基被测定两次,可以明显减少原始数据误差,是目前下一代基因分析技术中准确度最高的。该技术主要的缺点是序列读长相对较短。Illumina基因组分析仪每次延伸前都将上一步的反应试剂洗脱,然后加入新的反应体系,所以每次引物延伸过程掺入1个碱基。其主要的缺点在于光信号衰减和移相等原因使得序列读长较短,并且错误率随着DNA链的延长而累积。但在50bp读长的范围内,Illumina基因组分析仪的准确性可达99%以上。
3个测序平台的详细比较见表1:
因为miRNA的序列较短,较多应用于miRNA检测的新一代测序技术平台为Illumina基因组分析仪和ABLifeTechnologies的SOLiD系统。Illumina基因组分析仪具有所需样品少,数据误差小,操作流程简单等特点。SOLiD系统的读长为35nt,且准确度最高,单次运行所得的数据量最大,特别适用于miRNA的研究。而Roche454基因组测序仪测序读长可达400~500碱基,而miRNA序列长度仅为22碱基,所以该测序仪常用于新物种的基因组测序和转录组测序,极少应用于miRNA测序。
1.3 测序所得数据的生物信息学处理
1.3.1基本生物信息学分析
对测序所得的粗数据进行基本生物信息学分析及处理,得到标准化的等长度序列,方便后续处理。与现有的miRNA生物信息数据库比对,可以检索出已知miRNA的信息。
以Illumina基因组分析仪为例,将实验测得的35nt sRNA通过perl脚本去除低质量数据,再根据动态算法去除接头序列、经过去污染、统计序列长度分布等后续过程得到约22nt长度的序列。获取每一读长的序列信息及表达水平[13,14]。将所得的初数据通过与miRNA的生物信息数据库如miRBase18.0(www.mirbase.org/)进行比较,可以获取数据库中已有的miRNA的表达信息,包括结构、长度、表达水平等。
1.3.2未知 miRNA 检测
测序所得数据通过基本生物信息学分析可以获得已知miRNA的表达谱,但不能检测出未知miRNA。研究者开发了一系列软件以实现新miRNA的检测。不同软件算法各异,其基本原理大多基于pre-miRNA特异性的发卡-茎环结构。使用SOAP2.0程序(http://soap.genomics.org.cn/) 将所得的高质量序列与物种基因组进行比对[15],筛选出与基因组配对的高通量测序获得的短序列,并且提取其上游和下游各100个核苷酸序列,以获取可能的pre-miRNA序列信息。使用Rfold软件[16]或mfold3.1软件[17]进行RNA二级结构预测后,使用MIREAP[18,19]进行后续分析。
MIREAP是深圳华大基因开发的专用于新一代测序平台数据分析的软件,主要功能是检测新miRNA。新miRNA需满足以下3个条件:(1)经Rfold软件或mfold3.1软件预测的RNA二级结构符合pre-miRNA的发卡结构,且存在一条臂无内环或凸起,并包含成熟miRNA的序列;(2)二级结构稳定,杂交的自由能小于-20kcal/mol;(3)该发卡结构的编码基因位于基因间区或内含子区。
其他广泛使用的软件包括Hendrix等[20]开发的miRTRAP软件(miRNA Tests for Read Analysis and Prediction)、Mackowiak等[21]开发的miRDEEP和Buermans等[22]开发的E-miR。miRDEEP[21]开发最早,应用最为广泛,其灵敏度和准确性均较高。其原理是,Dicer酶在特定位置剪切具有发卡-茎环结构的miRNA前体形成miRNA链、miRNA互补链和一个环。在高通量测序中,这3种结构都有被测序的可能性。当把测序所得序列通过软件进行合并组装时,由于pre-miRNA特殊结构和特异性剪切,该3个结构的碱基序列彼此不相重叠,并且miRNA链和miRNA互补链所代表的区域互相配对。miRDEEP的检出率与测序深度直接相关,因为测序深度越高,互补链和环结构的检出率越高,其与成熟miRNA链配对的可能性越大。
近年开发的miTRAP软件算法同样利用了pre-miRNA的茎环-发卡结构。与miRDEEP相比,其后续的筛选算法更为巧妙。Hendrix等[20]使用miRDEEP和miRTRAP对同一组数据进行了miRNA检测,结果显示与miRDEEP相比,miRTRAP有更高的miRNA检出率和更低的假阳性率。miRTRAP[20]使用两步筛选的原理进行检测:(1)初步筛选。成熟miRNA由pre-miRNA生成,而pre-miRNA的二级结构具有特征性的发卡-茎环结构。根据编码miRNA区域上下游基因的序列,预测可能编码的RNA的二级结构。再根据该二级结构是否符合发卡-茎环结构进行初步筛选。(2)排除假阳性结果。经过初步筛选的小RNA中可能包含piRNA、snoRNA等其他RNA。为了排除假阳性结果,该软件使用了平均反义产物易位(Average antisense product displacement,AAPD)的方法。AAPD基于miRNA反义链的两个特点:(1)不编码小RNA;(2)如果编码小RNA,一定是与原miRNA位置完全相对的miRNA。
1.3.3 目前生物信息学方法的不足
新一代测序技术产生的数据量十分庞大,对生物信息学处理来说是巨大的挑战。目前可用的miRNA的分析软件众多,算法多样,其检测miRNA结果常有少许出入,造成假阴性和假阳性结果。
假阴性结果是由于软件的不足和局限造成miRNA检出的遗漏。Hansen等[23]对之前发表的人胚胎干细胞的小RNA测序数据库进行了再分析,发现了112个之前未发现的miRNAs,这些miRNAs通过整合数据库得到了验证。作者认为整合多个小RNA数据库有助于避免有利数据被错认为实验噪声而遗漏。
假阳性结果常由于miRNA与其他小RNA序列的相似性造成。Schopman等[24]发现tRNA(Lys3)和tRNA(Lys5)被误认为miRNA,进入了miRBase数据库。它们分别是miR-1274b和mir-1274a。这些tRNA-miRNA类似物有类似miRNA18nt的结构,并且经软件预测的RNA有类似发卡的二级结构。作者在已经发表的miRNA数据库和人类基因组中发现了更多的tRNA-miRNA类似物。这提示我们确认新miRNA时应更加慎重。
2新一代测序技术应用
新一代测序技术可以用于miRNA表达谱检测,具有准确、高效的特点,并且在发现新的miRNA方面具有突出优势。此外,新一代测序技术在miRNA互补链、miRNA编辑和miRNA异构体检测,探索miRNA功能及调控机制,miRNA靶基因检测方面具有新的应用。
2.1新一代测序技术应用于miRNA表达谱检测
新一代测序技术能迅速高效地获取特定样品中的miRNA种类及特定miRNA的表达水平,即miRNA表达谱。科学家们目前应用新一代测序技术检测各模型生物的miRNA表达谱,如秀丽隐杆线虫(Caenorhabditis elegans)、海胆(Cionaintest inalis)、果蝇(Drosophilam elanogaster)、斑马鱼(Danio rerio)、爪蟾(Xenopus laevis)、小鼠(Mus musculus)、拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)等[25]。检测胚胎干细胞、造血干细胞、神经元、脂肪细胞在发育过程的miRNA的表达谱变化及其靶基因,有助于我们明确各细胞的发育机制[26]。对于乙型肝炎病毒、艾滋病毒、巨细胞病毒等病毒的研究主要集中在发现病毒合成的miRNA以及参与病毒宿主反应的宿主miRNAs[27,28]。此外,miRNA高通量检测技术已经被成功运用于临床医学领域,如肿瘤、心血管疾病、糖尿病、肾脏疾病、神经系统疾病、病毒感染、自身免疫病等疾病,以获取疾病特异性的miRNA表达谱[5,25]。这些miRNA在疾病的发生发展和转归中发挥着重要的调控作用,同时也是潜在的疾病标志物和治疗靶点。
2.2 新一代测序技术应用于miRNA互补链、miRNA编辑和miRNA异构体检测
miRNA的前体(Pre-miRNA)通过RNA酶Dicer剪切产生一个miRNA二聚体,由miRNA链和miRNA互补链组成。miRNA链进入蛋白功能复合体,成为成熟的miRNA,发挥转录后基因调控作用,而它的互补链在多数情况下会很快降解,很难检测到。但对于一小部分miRNA而言,它们的互补链并不会被完全降解,它们会进入另一个蛋白功能复合体,成为成熟的miRNA行使功能。这些由互补链形成的miRNA在命名时,会在右上角标记一个星号以便区分[6,29]。因为难以检测之前人们一直认为miRNA*没有实际作用,新一代测序使得全面检测并探索miRNA*成为可能。例如,Kuchenbauer等运用测序技术发现mir-223*表达水平增高与急性髓性白血病(AML)病人的高存活率相关。
RNA编辑是指在初级转录物上增加、删除或取代某些核苷酸而改变遗传信息,是一种遗传信息在RNA水平发生改变的过程,可使RNA序列不同于基因组模板DNA序列[7,31]。近来研究发现,miRNA基因的转录产物是RNA编辑酶如ADARs的主要靶点之一[32]。miRNA编辑所致的单核苷酸变异会影响成熟miRNA的生成,并且干扰miRNA与特定靶mRNA结合[32,33]。对miRNA编辑的检测需要对同一样本中得到的基因组DNA和miRNA基因转录产物进行高通量测序,再进行比对和确认[34]。Heale等[31]认为,虽然miRNA编辑普遍存在,细胞实验已证实miRNA编辑对于特定miRNA的生成和靶向的影响,但目前仍然缺乏足够证据证明miRNA编辑对活体动物miRNA的网络及功能的存在影响。这或许与miRNA调控网络的复杂性相关。
isomiR即miRNA异构体,是指miRNA基因经过剪接修饰后形成多个序列稍有差异的成熟miRNAs,例如miRNA的3′末端转录后水平的非模板性的尿苷或腺苷添加[8]。Marti等[35]使用Illumia平台对正常人和亨廷顿脑病人的额皮质和纹状体进行了miRNA测序。高达80%~90%的miRNAs存在isomiR。
新一代测序技术可以鉴别单核苷酸变异,对于拷贝数量较少、核苷酸序列与标准探针相比存在差异的miRNA序列来说是最佳的检测方法。新一代测序技术广泛应用于miRNA遗传变异的研究,将加深我们对miRNA生物合成和调控通路的理解。
2.3 新一代测序技术应用于miRNA作用及调控机制的研究
新一代测序技术可用于检测miRNA的靶mRNA。主要检测方法主要包括基于Argonaute蛋白的交联免疫共沉淀测序技术(High-throughput sequencing of RNA isolated by crosslinking immunopre-cipitation,HITS-CLIP)以及基于降解组(degradome)测序的RNA末端平行分析法(Parallel Analysis of RNA Ends,PARE)[36]。
交联免疫共沉淀测序技术可用于与RNA结合蛋白相互作用的RNA的测序。首先,交联(Crosslink-ing)RNA和与之结合的蛋白,使用该特定蛋白特异的抗体免疫沉淀RNA和蛋白的复合体,之后提取其中的RNA,进行高通量测序并使用生物信息学方法对相关RNA进行分析。Argonaute蛋白是miRNA蛋白功能复合体(RNA-induced silencing complex,RISC)的核心催化组分。因为Argonaute蛋白同时与miRNA和靶mRNA结合,所以使用Argonaute蛋白的抗体进行免疫沉淀,可同时获取miRNA及其靶mRNA。基于Argonaute的HITS-CLIP测序已经被成功应用于获取miRNA和靶mRNA相互作用的图谱[9,36]。
RNA末端平行分析法(Parallel Analysis of RNA Ends,PARE)通过检测RNA降解组(degradome)鉴定miRNA的靶mRNA[9,37,38]。经miRNA机制剪切产生的mRNA片段含有5′端单磷酸基团和3′端polyA尾巴。提取并分离含有3′端polyA尾巴的RNA,加入包含基质金属蛋白酶12(MMP-12)结合位点的5′端RNA接头,与5′单磷酸化的mRNA片段特异结合。在进行反转录和PCR扩增后,使用MMP-12对RNA接头的特定结合位点进行剪切,获得同等大小的片段。分离该片段并与3′端双链DNA接头连接,进行PCR扩增。最后,对纯化的扩增产物进行高通量测序,产生的数据与cDNA文库进行比对,使用生物信息学方法进行分析。因为miRNA机制剪切产生的mRNA片段末端与miRNA互补配对,通过对mRNA片段的测序和分析可以了解特定miRNA的表达水平和编码信息。
与传统的生物信息学预测方法相比,这两种基于测序的方法具有假阳性率低、组织特异、定量(与miRNA的丰度无关,与实际上发挥功能的miRNA数量相关)等巨大优势,在快速确定miRNA靶mRNA方面有很大的应用。Yang等[36]建立了基于CLIP测序和PARE试验结果的数据库starBase,使我们能够更全面的了解miRNA与靶mRNA相互作用的复杂网络。
miRNA的功能和调控机制十分复杂,新一代测序技术可以判断miRNA与RNA、DNA、蛋白质的相互作用,有助我们逐步解开谜团。
Baccarini等[39]分析了哺乳动物细胞中成熟miRNA发挥调控作用后的命运。通过对miRNA和靶mRNA在平稳状态和调节状态的定量分析,作者发现每一个miRNA分子能够调节至少两个靶mRNA。由此证明,miRNA在体内并不随着靶基因的降解而降解。进入沉默基因复合体发挥功能之后,miRNA3′端被修饰,该修饰加速了miRNA的降解速度。
Brameier等[40]发现了sno-miRNA,其本质是C/Dbox snoRNAs,snoRNA或称核仁小分子RNA是一类广泛分布于真核生物细胞核仁的小分子非编码RNA,具有保守的结构元件,并以此划分为3大类:boxC/DsnoRNA、boxH/ACA snoRNA和MRPRNA。其中boxC/D和boxH/ACA是已知snoRNA的主要类型,以碱基配对的方式分别指导着核糖体RNA的甲基化和假尿嘧啶化修饰[41]。而sno-miRNA却具有类似miRNA的转录后基因调节功能。Scott等[42]发现了具有snoRNA特点的miRNA前体,并且猜测snoRNA和miRNA之间存在进化联系。作者发现miR-151、miR-605、mir-664、miR-215和miR-140的前体结构与H/ACA snoRNAs类似,并且可以与H/ACA snoRNA特异性结合蛋白dyskerin结合。
随着新一代测序技术的逐步应用,我们对于miRNA生成和降解、miRNA与RNA、DNA、蛋白质之间的关联的认识将更加深入。
3结语
MiRNAs是一类在进化上高度保守的非编码小分子单链RNA(~22nt),在基因转录后调控中发挥至关重要的作用。越来越多的证据表明,miRNAs参与很多重要的生理和病理过程,例如发育、器官形成、调亡、细胞增殖、肿瘤发生等。近年来飞速发展的新一代测序技术在miRNA检测方面具有重要的应用。相比于传统的miRNA检测方法,新一代测序技术通量高、对遗传物质检测完全且准确度高,可重复性好等优点,在探索新miRNA、miRNA互补链[6]、miRNA编辑[7]、miRNA异构体[8]检测以及miRNA靶基因检测[9]等方面具有巨大优势[4]。随着新一代测序技术的不断发展,测序成本不断降低,在未来几年,新一代测序技术的使用率或将大大增加。这对于我们明确miRNA在各种生理和病理过程中的表达情况、自身调控、基因转录后调控网络具有重要的意义。疾病特异性miRNA的检测对于疾病的诊断和预后判断极有价值。特定miRNA及其作用的调控有可能是重要的治疗靶点,有助于新药的研发。随着新一代测序技术的发展,人类对于miRNA在各种生理病理过程中的功能和调控将会有更进一步的认识。
参考文献(References):
[1] Lee RC, Feinbaum RL, Ambros V. The C. elegans hetero- chronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell, 1993, 75(5): 843–854.
[2] Reinhart BJ, Slack FJ, Basson M, Pasquinelli AE, Bet- tinger JC, Rougvie AE, Horvitz HR, Ruvkun G. The 21- nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans. Nature, 2000, 403(6772): 901–906.
[3] Lagos-Quintana M, Rauhut R, Lendeckel W, Tuschl T. Identification of novel genes coding for small expressed RNAs. Science, 2001, 294(5543): 853–858.
[4] Kozomara A, Griffiths-Jones S. miRBase: integrating mi- croRNA annotation and deep-sequencing data. Nucleic Acids Res, 2011, 39(Database issue): 152–157.
[5] Van Rooij E. The art of microRNA research. Circ Res, 2011, 108(2): 219–234.
[6] Kuchenbauer F, Mah SM, Heuser M, McPherson A, Rüschmann J, Rouhi A, Berg T, Bullinger L, Argiropoulos B, Morin RD, Lai D, Starczynowski DT, Karsan A, Eaves CJ, Watahiki A, Wang YZ, Aparicio SA, Ganser A, Krau- ter J, Döhner H, Döhner K, Marra MA, Camargo FD, Palmqvist L, Buske C, Humphries RK. Comprehensive analysis of mammalian miRNA* species and their role in myeloid cells. Blood, 2011, 118(12): 3350–3358.
[7] Dupuis DE, Maas S. MiRNA editing. Methods Mol Biol, 2010, 667: 267–279.
[8] Fernandez-Valverde SL, Taft RJ, Mattick JS. Dynamic isomiR regulation in Drosophila development. RNAna, 2010, 16(10): 1881–1888.
[9] Thomson DW, Bracken CP, Goodall GJ. Experimental strategies for microRNA target identification. Nucleic Ac- ids Res, 2011, 39(16): 6845–6853.
[10] Bentley DR, Balasubramanian S, Swerdlow HP and et al. Rapid whole-genome mutational profiling using next-generation sequencing technologies. Genome Res, 2008, 18(10): 1638–1642.
[13] Chen X, Li QB, Wang J, Guo X, Jiang XG, Ren ZJ, Weng CY, Sun GX, Wang XQ, Liu YP, Ma LJ, Chen JY, Wang J, Zen K, Zhang JF, Zhang CY. Identification and charac- terization of novel amphioxus microRNAs by Solexa se- quencing. Genome Biol, 2009, 10(7): R78.
[14] 陈洁 , 林海建 , 潘光堂 , 张志明 , 张彪 , 沈亚欧 , 覃成 , 张谦, 赵茂俊. 利用深度测序技术检测玉米根系和叶片 中已知的 microRNAs. 遗传, 2010, 32(11): 1175–1186.
[15] Li RQ, Li YR, Kristiansen K, Wang J. SOAP: short oli- gonucleotide alignment program. Bioinformatics, 2008, 24(5): 713–714.
[16] Kiryu H, Kin T, Asai K. Rfold: an exact algorithm for computing local base pairing probabilities. Bioinformatics, 2008, 24(3): 367–373.
[17] Zuker M. Mfold web server for nucleic acid folding and hybridization prediction. Nucleic Acids Res, 2003, 31(13): 3406–3415.
[18] Li GX, Li YJ, Li XJ, Ning XM, Li MH, Yang GS. Mi- croRNA identity and abundance in developing swine adi- pose tissue as determined by Solexa sequencing. J Cell Biochem, 2011, 112(5): 1318–1328.
[19] Song CN, Fang JG, Wang C, Guo L, Nicholas KK, Ma ZQ. MiR-RACE, a new efficient approach to determine the precise sequences of computationally identified trifoliate orange (Poncirus trifoliata) microRNAs. PLoS One, 2010, 5(6): e10861.
[20] Hendrix D, Levine M, Shi WY. miRTRAP, a computa- tional method for the systematic identification of miRNAs from high throughput sequencing data. Genome Biol, 2010, 11(4): R39.
[21] Friedländer MR, Chen W, Adamidi C, Maaskola J, Ein- spanier R, Knespel S, Rajewsky N. Discovering microR- NAs from deep sequencing data using miRDeep. Nat Bio- technol, 2008, 26(4): 407–415.
[22] Buermans HPJ, Ariyurek Y, van Ommen G, den Dunnen JT, 't Hoen PAC. New methods for next generation sequencing based microRNA expression profiling. BMC Genomics, 2010, 11: 716.
[23] Hansen TB, Bramsen JB, Kjems J. Re-inspection of small RNA sequence datasets reveals several novel human miRNA genes. PLoS One, 2010, 5(6): e10961.
[24] Schopman NCT, Heynen S, Haasnoot J, Berkhout B. A miRNA-tRNA mix-up: tRNA origin of proposed miRNA. RNA Biol, 2010, 7(5): 573–576.
[25] 盛熙晖, 杜立新. MicroRNA 及其在人和动物上的研究 进展. 遗传, 2007, 29 (6): 651–658.
[26] 王世华, 边春景, 赵春华. microRNA 在胚胎干细胞中的 表达及作用. 遗传, 2008, 30(12): 1545–1549.
[27] Wu FL, Jin WB, Li JH, Guo AG. Targets for human en- coded microRNAs in HBV genes. Virus Genes, 2011, 42(2): 157–161.
[28] Weng LH, Wu XW, Gao HL, Mu B, Li XJ, Wang JH, Guo C, Jin JM, Chen Z, Covarrubias M, Yuan YC, Weiss LM, Wu HQ. MicroRNA profiling of clear cell renal cell car- cinoma by whole-genome small RNA deep sequencing of paired frozen and formalin-fixed, paraffin-embedded tis- sue specimens. J Pathol, 2010, 222(1): 41–51.
[29] Buchan JR, Parker R. Molecular biology. The two faces of miRNA. Science, 2007, 318(5858): 1877–1878.
[30] Kuchenbauer F, Mah SM, Heuser M, McPherson A, Rüschmann J, Rouhi A, Berg T, Bullinger L, Argiropoulos B, Morin RD, Lai D, Starczynowski DT, Karsan A, Eaves CJ, Watahiki A, Wang YZ, Aparicio SA, Ganser A, Krau- ter J, Döhner H, Döhner K, Marra MA, Camargo FD, Palmqvist L, Buske C, Humphries RK. Comprehensive analysis of mammalian miRNA* species and their role in myeloid cells. Blood, 2011, 118(12): 3350–3358.
[31] Heale BS, Keegan LP, O'Connell MA. The effect of RNA editing and ADARs on miRNA biogenesis and function. Adv Exp Med Biol, 2010, 700: 76–84.
[32] Kawahara Y, Megraw M, Kreider E, Iizasa H, Valente L, Hatzigeorgiou AG, Nishikura K. Frequency and fate of microRNA editing in human brain. Nucleic Acids Res, 2008, 36(16): 5270–5280.
[33] Heale BSE, Eulalio A, Schulte L, Vogel J, O'Connell MA. Analysis of A to I editing of miRNA in macrophages ex- posed to Salmonella. RNA Biol, 2010, 7(5): 621–627.
[34] Reid JG, Nagaraja AK, Lynn FC, Drabek RB, Muzny DM, Shaw CA, Weiss MK, Naghavi AO, Khan M, Zhu HF, Tennakoon J, Gunaratne GH, Corry DB, Miller J, McManus MT, German MS, Gibbs RA, Matzuk MM, Gunaratne PH. Mouse let-7 miRNA populations exhibit RNA editing that is constrained in the 5'-seed/cleavage/ anchor regions and stabilize predicted mmu-let-7a: mRNA duplexes. Genome Res, 2008, 18(10): 1571–1581.
[35] MartíE, Pantano L, Bañez-Coronel M, Llorens F, Mi- ñones-Moyano E, Porta S, Sumoy L, Ferrer I, Estivill X. A myriad of miRNA variants in control and Huntington's disease brain regions detected by massively parallel se- quencing. Nucleic Acids Res, 2010, 38(20): 7219–7235.
[36] Yang JH, Li JH, Shao P, Zhou H, Chen YQ, Qu LH. star- Base: a database for exploring microRNA-mRNA interac- tion maps from Argonaute CLIP-Seq and Degradome-Seq data. Nucleic Acids Res, 2011, 39(Database issue): D202– D209.
[37] Burroughs AM, Ando Y, de Hoon MJL, Tomaru Y, Nishibu T, Ukekawa R, Funakoshi T, Kurokawa T, Suzuki H, Ha- yashizaki Y, Daub CO. A comprehensive survey of 3’ animal miRNA modification events and a possible role for 3’ adenylation in modulating miRNA targeting effective- ness. Genome Res, 2010, 20(10): 1398–1410.
[38] Leung AKL, Young AG, Bhutkar A, Zheng GX, Bosson AD, Nielsen CB, Sharp PA. Genome-wide identification of Ago2 binding sites from mouse embryonic stem cells with and without mature microRNAs. Nat Struct Mol Biol, 2011, 18(2): 237–244.
[39] Baccarini A, Chauhan H, Gardner TJ, Jayaprakash AD, Sachidanandam R, Brown BD. Kinetic analysis reveals the fate of a microRNA following target regulation in mam- malian cells. Curr Biol, 2011, 21(5): 369–376.
[40] Brameier M. Genome-wide comparative analysis of mi- croRNAs in three non-human primates. BMC Res Notes, 2010, 3: 64.
[41] Scott MS, Ono M. From snoRNA to miRNA: Dual func- tion regulatory non-coding RNAs. Biochimie, 2011,93(11): 1987−1992.
[42] Scott MS, Avolio F, Ono M, Lamond AI, Barton GJ. Hu- man miRNA precursors with box H/ACA snoRNA features. PLoS Comput Biol, 2009, 5(9): e1000507.