过去20年,美国应用生物系统公司(ABI)在测序方面一直占据着垄断地位。自公司的共同创始人Leroy Hood在上世纪80年代中期设计了第一台自动荧光测序仪之后,生命科学研究就摆脱了手工测序的繁琐和辛劳,骄傲地迈入自动测序的新时代。直到2005年,454推出了FLX焦磷酸测序平台,ABI的领先地位开始有些动摇。之后,ABI迅速收购了一家测序公司——Agencourt Personal Genomics,并在2007年底推出了SOLiD 新一代测序平台。从SOLiD到如今的SOLiD 3,短短一年多时间,它已经上演了一出精彩的“一级方程式赛车”。
SOLiD全称为supported oligo ligation detetion,它的独特之处在于以四色荧光标记寡核苷酸的连续连接合成为基础,取代了传统的聚合酶连接反应,可对单拷贝DNA片段进行大规模扩增和高通量并行测序。就通量而言,SOLiD 3系统是革命性的,目前SOLiD 3单次运行可产生50GB的序列数据,相当于17倍人类基因组覆盖度。而其无以伦比的准确性、系统可靠性和可扩展性更让它从其他新一代测序平台中脱颖而出。为什么SOLiD能轻松实现貌似不可能的任务?让生物通带你从测序原理入手,一探究竟。
SOLiD工作流程
a. 文库制备
SOLiD系统能支持两种测序模板:片段文库(fragment library)或配对末端文库(mate-paired library)。使用哪一种文库取决于你的应用及需要的信息。片段文库就是将基因组DNA打断,两头加上接头,制成文库。如果你想要做转录组测序、RNA定量、miRNA探索、重测序、3’,5’-RACE、甲基化分析、ChIP测序等,就可以用它。如果你的应用是全基因组测序、SNP分析、结构重排/拷贝数,则需要用配对末端文库。配对末端文库是将基因组DNA打断后,与中间接头连接,再环化,然后用EcoP15酶切,使中间接头两端各有27bp的碱基,再加上两端的接头,形成文库。
b. 乳液PCR/微珠富集
在微反应器中加入测序模板、PCR反应元件、微珠和引物,进行乳液PCR(Emulsion PCR)。PCR完成之后,变性模板,富集带有延伸模板的微珠,去除多余的微珠。微珠上的模板经过3’修饰,可以与玻片共价结合。看到这里,是不是有一种似曾相识的感觉呢?那就对了,此步骤与454的GS FLX基本相同。不过SOLiD系统的微珠要小得多,只有1 um。
乳液PCR最大的特点是可以形成数目庞大的独立反应空间以进行DNA扩增。其关键技术是“注水到油”,基本过程是在PCR反应前,将包含PCR所有反应成分的水溶液注入到高速旋转的矿物油表面,水溶液瞬间形成无数个被矿物油包裹的小水滴。这些小水滴就构成了独立的PCR反应空间。理想状态下,每个小水滴只含一个DNA模板和一个P1磁珠,由于水相中的P2引物和磁珠表面的P1引物所介导的PCR反应,这个DNA模板的拷贝数量呈指数级增加,PCR反应结束后,P1磁珠表面就固定有拷贝数目巨大的同来源DNA模板扩增产物。
c. 微珠沉积
3’修饰的微珠沉积在一块玻片上。在微珠上样的过程中,沉积小室将每张玻片分成1个、4个或8个测序区域。SOLiD系统最大的优点就是每张玻片能容纳更高密度的微珠,在同一系统中轻松实现更高的通量。
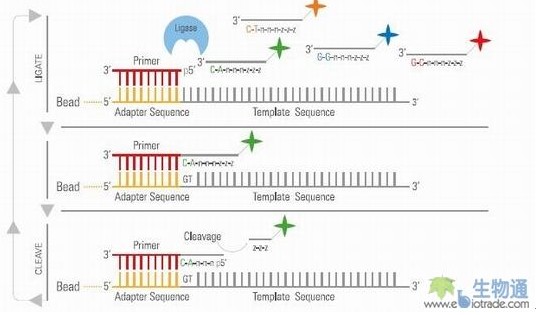
d. 连接测序
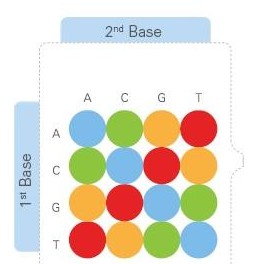
这一步可就是SOLiD的独门秘笈了。它的独特之处在于没有采用惯常的聚合酶,而用了连接酶。SOLiD连接反应的底物是8碱基单链荧光探针混合物。连接反应中,这些探针按照碱基互补规则与单链DNA模板链配对。探针的5’末端分别标记了CY5、Texas Red、CY3、6-FAM这4种颜色的荧光染料。探针3’端1~5位为随机碱基,可以是ATCG四种碱基中的任何一种碱基,其中第1、2位构成的碱基对是表征探针染料类型的编码区,下图的双碱基编码矩阵规定了该编码区16种碱基对和4种探针颜色的对应关系,而3~5位的“n”表示随机碱基,6~8位的“z”指的是可以和任何碱基配对的特殊碱基。
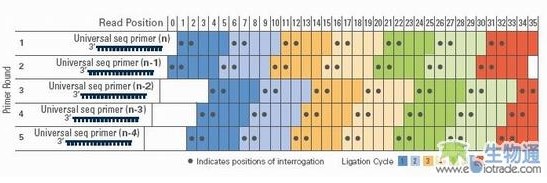
单向SOLiD测序包括五轮测序反应,每轮测序反应含有多次连接反应。第一轮测序的第一次连接反应由连接引物“n”介导,由于每个磁珠只含有均质单链DNA模板,所以这次连接反应掺入一种8碱基荧光探针,SOLiD测序仪记录下探针第1、2位编码区颜色信息,随后的化学处理断裂探针3’端第5、6位碱基间的化学键,并除去6~8位碱基及5’末端荧光基团,暴露探针第5位碱基5’磷酸,为下一次连接反应作准备。因为第一次连接反应使合成链多了5个碱基,所以第二次连接反应得到模板上第6、7位碱基序列的颜色信息,而第三次连接反应得到的是第11、12位碱基序列的颜色信息
几个循环之后,引物重置,开始第二轮的测序。由于第二轮连接引物n-1比第一轮错开一位,所以第二轮得到以0,1位起始的若干碱基对的颜色信息。五轮测序反应反应后,按照第0、1位,第1、2位... …的顺序把对应于模板序列的颜色信息连起来,就得到由“0,1,2,3…”组成的SOLiD原始颜色序列。
e. 数据分析
SOLiD测序完成后,获得了由颜色编码组成的SOLiD原始序列。理论上来说,按照“双碱基编码矩阵”,只要知道所测DNA序列中任何一个位置的碱基类型,就可以将SOLiD原始颜色序列“解码”成碱基序列。但由于双碱基编码规则中双碱基与颜色信息的简并特性(一种颜色对应4种碱基对),前面碱基的颜色编码直接影响紧跟其后碱基的解码,所以一个错误颜色编码就会引起“连锁解码错误”,改变错误颜色编码之后的所有碱基。
和其它所有测序仪一样,测序错误在所难免,关键是对测序错误的评价和后续处理。由于SOLiD系统采用了双碱基编码技术,在测序过程中对每个碱基判读两遍,从而减少原始数据错误,提供内在的校对功能。这样,双保险确保了SOLiD系统原始碱基数据的准确度大于99.94%,而在15X覆盖率时的准确度可以达到99.999%,是目前新一代基因分析技术中准确度最高的。
为避免“连锁解码错误”的发生,SOLiD数据分析软件不直接将SOLiD原始颜色序列解码成碱基序列,而是依靠reference序列进行后续数据分析。SOLiD序列分析软件首先根据“双碱基编码矩阵”把reference碱基序列转换成颜色编码序列,然后与SOLiD原始颜色序列进行比较,来获得SOLiD原始颜色序列在reference的位置,及两者的匹配性信息。Reference转换而成的颜色编码序列和SOLiD原始序列的不完全匹配主要有两种情况:“单颜色不匹配”和“两连续颜色不匹配”。由于每个碱基都被独立地检测两次,且SNP位点将改变连续的两个颜色编码,所以一般情况下SOLiD将单颜色不匹配处理成测序错误,这样一来,SOLiD分析软件就完成了该测序错误的自动校正;而连续两颜色不匹配也可能是连续的两次测序错误,SOLiD分析软件将综合考虑该位置颜色序列的一致性及质量值来判断该位点是否为SNP。
在初步了解了SOLiD系统的工作原理之后,我们才能明白它的魅力所在。
系统可扩展性
SOLiD系统采用开放玻片式的结构,使用包被DNA样品的微珠来输入基因组信息。微珠密度并不是一成不变的,系统支持更高密度的微珠富集。开放式玻片形式、微珠富集、以及软件算法的结合,能使平台轻松升级到更高的通量,而无需对基础技术和配置做重大改变。这也是SOLiD系统平均每季度将通量扩大一倍的原因所在。
无以伦比的通量
目前SOLiD 3系统单次运行能产生50 GB的人基因组序列数据,相当于基因组的17倍覆盖度,这显然是其他任一台新一代测序系统都无法达到的。今年初,ABI公司和贝勒医学院人类基因组测序中心(HGSC)的科学家总结了他们在千人基因组计划首次数据发布中的贡献。作为商业参与者以及与HGSC共同协作,ABI公司利用SOLiD系统产生了超过460 GB可作图的序列数据,比这两个机构的预定目标高出了65%。而通量的升高也有望进一步降低基因组测序的费用,成本只需1万美元的人类基因组测序指日可待。
最大的灵活性
SOLiD 3系统具有两个独立的流动室,让用户能在一台SOLiD分析仪中运行两个完全独立的实验——同时提供两套仪器。玻片也能分成1个、4个或8个小室。而20个条形码序列则提供了额外的灵活性,显著增加了定向重测序、表达和ChIP分析的经济性。目前最多能同时运行320个样品(2×8×20)。
至此,SOLiD系统已不再是一台单纯的测序仪,而是成为功能更全面的基因分析仪。除了测序和重测序,还能进行全基因表达图谱分析、SNP、microRNA、ChIP、甲基化等多种分析。
全基因表达图谱分析
芯片大概是目前应用最广泛的从全局角度分析基因表达整体模式的方法。然而,基于杂交技术的微阵列技术只限用于已知序列,无法检测新的mRNA;而且杂交技术灵敏度有限,难以检测低丰度的目标(需要更多的样品量),难以检测重复序列;也无法捕捉到目的基因表达水平的微小变化------而这恰恰是研究在刺激下或环境变化时的生物反应所必需的。
与芯片技术相比,基于测序的高灵敏SOLiD技术可对单个细胞和癌症样品中存在的痕量RNA进行整体的全基因组表达图谱分析,每次运行能定位高达2亿4千万个标签(mRNA的相对表达水平可通过系统产生的序列标签数目来计算),可检测低至每个细胞中10-40pg的总RNA,即使mRNA表达水平很低,SOLiD系统也能够无偏向性地分析样品中存在的已知和未知mRNA,从而定量特定mRNA的差异表达模式。起始样品比微阵列技术要少得多,尤其适用于来源极为有限的生物样品分析,如癌症干细胞----分析其基因和非编码RNA的表达图谱有助于有助于加速发掘潜在的生物标志物,从而更准确区分不同的疾病类型以及识别疾病易感性,帮助于研究人员更好地了解病变细胞的特性。
RNA研究
除了单细胞基因表达图谱分析,SOLiD系统在RNA方面的其他应用还包括利用SOLiD Small RNA Expression Kit来发现和筛选小分子RNA,实现在无需预先知道序列信息的情况下高通量发现新的RNA分子。这个方案有望显著地提高研究人员鉴别小分子RNA的能力,将过去不可能完成的实验变为可能。目前已发现的microRNAs还非常有限,SOLiD可在不知道目标分子DNA序列的情况下进行检测和定量小的RNA分子,可将样品制备工作从常规方法的四天缩短为仅需一天,是分析在生物样品中表达的已知和未知miRNA及其它小分子RNAs的有效工具。利用SOLiD Whole Transcriptome Kit还可以探索和鉴定全转录本。SOLiD无可比拟的高通量和测序数据的高精确性使得可以用短序列读长即可测序整个转录组。了解转录组对有助于解开导致复杂疾病的分子通路的秘密。这一系列应用补充使研究人员能在单个超高通量平台上开展综合的RNA研究。
SNP分析
尽管绝大多数的人类遗传信息在所有人中都相同,但是研究人员通常更感兴趣的是研究个体之间微小的遗传差异。这种差异包括单碱基变异,以及被称为结构变异的各种较大片段DNA序列变异。结构变异包括DNA片段的插入、缺失、倒位和易位,结构变异的DNA片段范围可从几个碱基对到数百万个碱基对,可能对基因产生重要影响,并导致人类疾病的发生。SOLiD流程获得的严密的片段范围,使研究人员可以鉴别出很宽范围内的插入和缺失片段,结构重排也能很容易鉴别出来。这个平台的超高通量使研究人员可轻而易举地获得高度基因组覆盖率的数据,精确鉴定个体基因组中存在的数百万个单碱基多态性SNP,揭示大量此前未知、具有潜在医学价值的遗传变异,从而促进我们对正常/疾病状态下DNA结构变异的了解,以及在更高的分辨率下对结构变异进行深入分析,解释个体之间的易感性差异和对疾病治疗应答的差异,最终实现个性化医疗。
甲基化分析
甲基化是自然发生的DNA化学修饰的一种。已知抑癌基因的失活与DNA序列特定区域的甲基化有关。而去甲基化则可能导致基因组不稳定和表达模式变化。DNA甲基化区域可能作为基因在癌症过程中的标记。研究人员一直致力研究从正常到癌变过程中甲基化模式如何变化的,原癌基因异常甲基化模式在癌变过程中扮演怎样的角色。SOLiD系统运行通量非常惊人,很快就可以做多个样本全基因组甲基化模式检测,使得研究人员可以鉴别基因组中对应元件的甲基化状态,从而帮助研究人员检测甲基化模式是否可以作为癌症的生物标识,以及更好了解甲基化在癌变过程中扮演的角色。