做pca大体思路:
snp raw data——转成plink二进制格式——然后用gcta生成matrix——然后用R作图
1、转二进制文件,先说把raw data转成plink的bfile二进制格式,一般来说snp结果都是从芯片或测序结果call出来的,芯片可能要写脚本把snp抠出来,这里不多说;测序结果call 的snp一般都是vcf格式,所以我们用到一个vcftools的软件,该软件只能在linux下使用。
1.1、vcf转格式,生成了tmp.map和tmp.ped两个文件。
[shell]vcftools --vcftmp.vcf --plink --out tmp[/shell]
1.2、用plink转成二进制文件,生成了分别以bed bim fam为后缀的tmp文件。
[shell]./plink --noweb--file tmp --make-bed --out tmp_bfile[/shell]
ps:plink里面--file 选项后跟文件名,不用加后缀~
2、生成matrix,gcta跟eigenstrat软件包做pca的效果是一样的,而且gcta比eigenstrat容易使用的多了,所以单纯做pca的话用gcta就好了,做gcta分两步。
2.1、[shell]./gcta--bfile tmp_bfile --make-grm --autosome --out tmp_grm[/shell]
说明:
1)tmp_bfile是你上一步plink生成的二进制文件(不包括后缀名)
2)tmp_grm是你指定输出的文件名
3)--autosome 意思是只选出常染色体来运行(对应1-22号染色体)
2.2、[shell]./gcta--grm tmp_grm --pca 3 --out tmp_pca[/shell]
说明:
1)tmp_grm是你上一步生成的文件名,不包含.grm.gz这个后缀
2)tmp_pca是输出文件
3)这样得到两个文件一个是tmp_pca.eigenval另一个是tmp_pca.eigenvec。在后者行首加入一行:1 2 pc1 pc2 pc3(分隔符为空格),并保存。
3、我们这就已经准备好了R作图用的matrix了,接下来用R作图。下载好R,然后两步走:
3.1、先把matrix读入到R中
[shell]a=read.table("c:/gcta/tmp_pca.eigenvec",header=TURE)[/shell]
3.2、这里举个例子来说明绘制散点图的参数
假如我们要做一个有5个中国人和3个日本人的全基因组的pca,那么我们的tmp_pca.eigenvec文件里面就应该是前面5个中国人的坐标信息,最后是3个日本人的坐标信息。
3.2.1绘制散点图:
[shell]plot(a$pc1,a$pc2,pch=c(rep(1,5),rep(2,3)),col=c(rep("yellow",5),rep("red",3)),main="PCA",xlab="pc1",ylab="pc2")[/shell]
说明:
1)pch=c(rep(1,5),rep(2,3))意思是把5个中国人用pch=1的图案来表示,日本人可推
2)col=c(rep("yellow",5),rep("red",3))意思就是把5个中国人用黄色标注,日本人可推
3.2.2增加图示信息:
[shell]legend("bottomright",c("chb","jpt"),pch=c(rep(1,5),rep(2,3)),col=c(rep("yellow",5),rep("red",3))) [/shell]
就能做出pca图了。
这是我用千人基因组的数据做出的pca
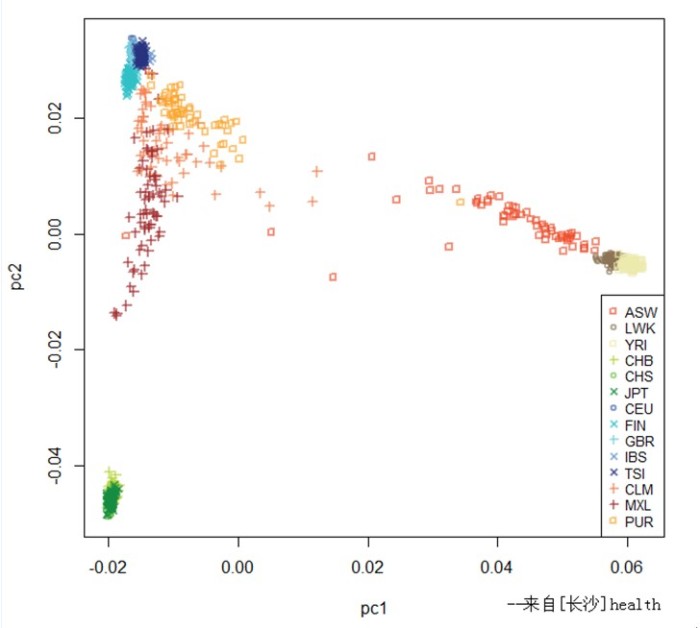 (注,这是用千人基因组得到的snp,来观察各个人种,如ASW,CEU等等人种间的关系,一定程度反应了人种之间的关系,同人种会聚集在一起,具体人种信息,可以去看看http://www.1000genomes.org/about )
(注,这是用千人基因组得到的snp,来观察各个人种,如ASW,CEU等等人种间的关系,一定程度反应了人种之间的关系,同人种会聚集在一起,具体人种信息,可以去看看http://www.1000genomes.org/about )
附1:常用颜色col的代号
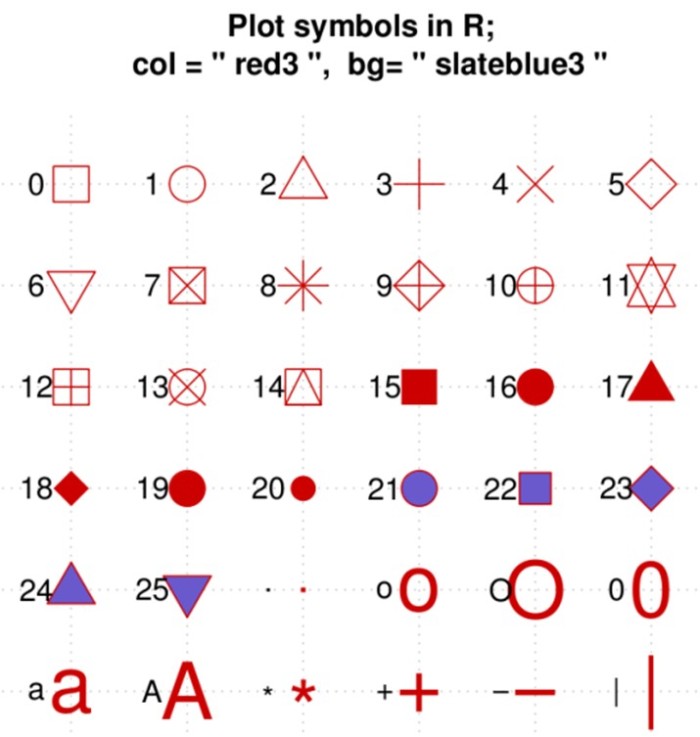
附2:图形符号pch的代号
上图来自http://rgraphics.limnology.wisc.edu/pch.php
本文由【 长沙】 health撰稿提供。
原帖地址:http://seq.cn/forum.php?mod=viewthread&tid=10085&page=1&extra=#pid22622




1F
人种之间差异这么大呀~
2F
支持
3F
咋搞这么一个头像,忒难看了吧
4F
GCAT收费吗?在哪下载?
5F
请问在用vcftools转换成plink所用的文件,怎样用于多个样本呢
6F
为什么我做到a=read.table(“c:/gcta/tmp_pca.eigenvec”,header=TURE)这一步的时候R告诉我TURE这个没找到呢……是不是前面加1 2 pc1 pc2 pc3的时候出错了啊……求教!
7F
a=read.table(“c:/gcta/tmp_pca.eigenvec”,header=TURE)
楼主,你的TRUE写错啦,难怪R识别不了的…………囧
8F
最后legend一步又写错了,被你坑了一下午
B1
@ ClytiaXu 你好,正确的应该是怎样的呀?
谢谢
9F
不过还是很有用,楼主威武
10F
我没办法在plob上更改编辑我的帖子,但在文章后面的seq.cn的原贴中已经做了更改,谢谢网友的提醒。
11F
你好~我想请教一下在gcta的第二部–pca这个参数如何确定?为什么画图的时候你选择了pc1和pc2而没有选择pc3,在三列选2列时的原则是什么?
灰常感谢灰常感谢
12F
你好,我想请教一下,group数很多的时候,是怎样用不同的颜色和图形符号标注的呢?