前面《DAVID使用说明文档(1)》中已经对DAVID做了一个简单的介绍,包括DAVID的简介以及其中的使用工具。下面介绍一下DAVID使用步骤:
1、向DAVID网站提交一个基因列表。
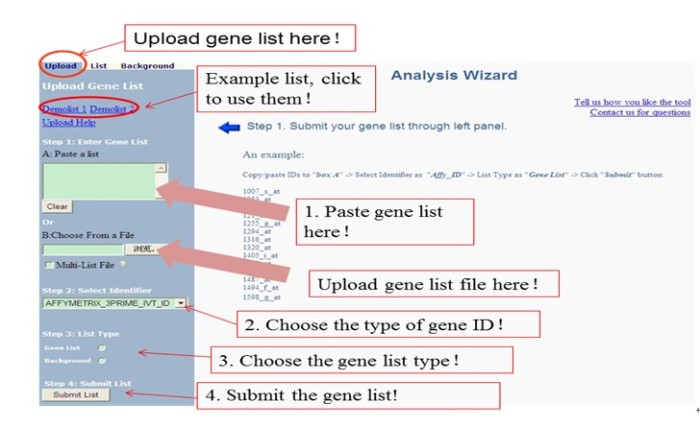
首先登录到网站http://david.abcc.ncifcrf.gov/ 的首页(见图9)。点击页面顶端的“Start Analysis”在弹出页面的左边有一个面板“Gene List Manager”,在该面板的“upload”标签下提交基因列表(基因列表的格式为每行一个基因或者行内的多个基因以逗号分隔,可以将基因列表黏贴到输入窗口或者以文件形式上传);接着选取输入基因列表的ID类型;最后确定列表的类型,是基因列表还是作为背景文件。点击use,进入分析。
图9 网站首页
上传的数据可以供所有的分析模块共享,而不需要重复上传。基因列表文件可以选取如图10中所示的Demolist1 和 Demolist2。本文中使用DAVID提供的Demolist1作为基因列表。附件一是人的血瘀和正常样品之间的显著差异表达基因列表,可供使用。
如果你要做的是全基因组背景或者是接近全基因组背景的研究,就不需要上传那个背景文件,网站会自动根据上传的基因列表类型,选择对应物种的所有基因作为背景文件。如果你要自己设定背景,也可以在“upload”标签中上传,然后在“Background”标签中选定所需的列表作为背景。本文中选取默认的背景文件,即人的全基因作为背景。
图10 上传数据窗口
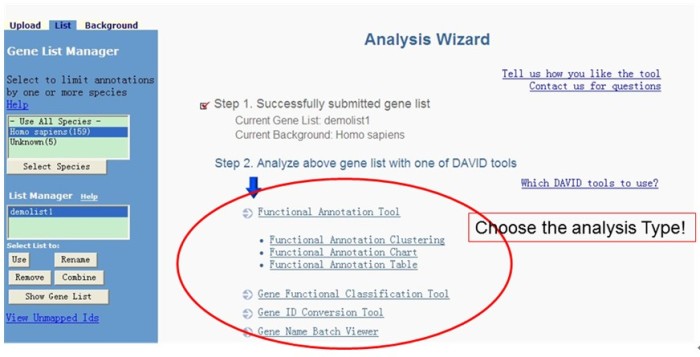
在“List”标签中,可以看到所有上传的列表。在图11所示的右侧中,选择分析项。
图11 Lsit标签和分析工具选择窗口
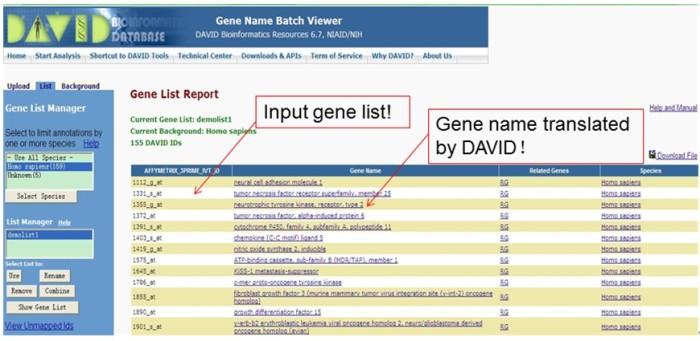
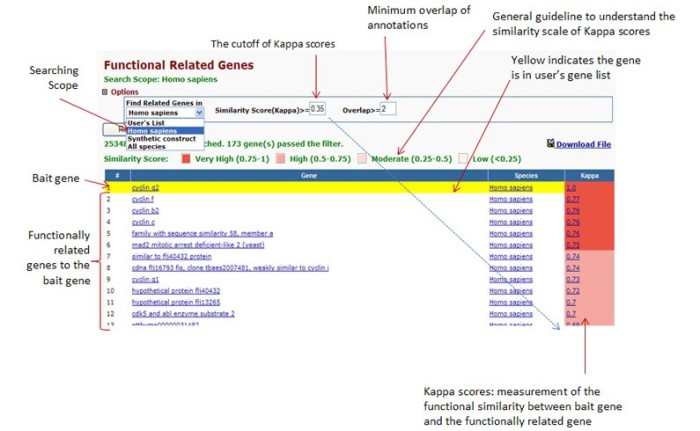
(1)选择Gene Name Batch Viewer这项分析,弹出的窗口显示分析结果,即基因ID和对应的基因名称、相关基因以及所属物种。用户可以据此初步判断,列表中是否含有感兴趣的基因(见图12)。Species栏指向物种相关的ncbi信息网页。
图12 Gene Name Batch Viewer分析结果
点击Related Genes栏中的 RG,将会出现跟该行基因功能相关的基因列表,如图13所示的结果。
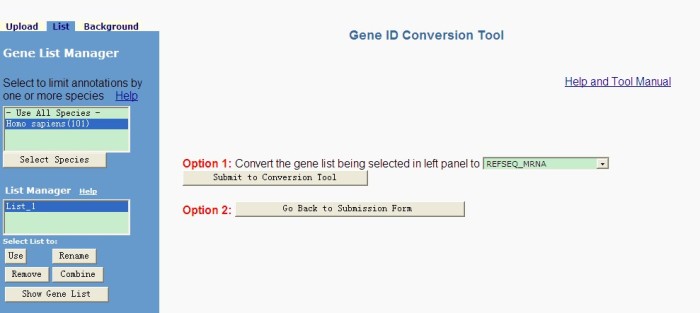
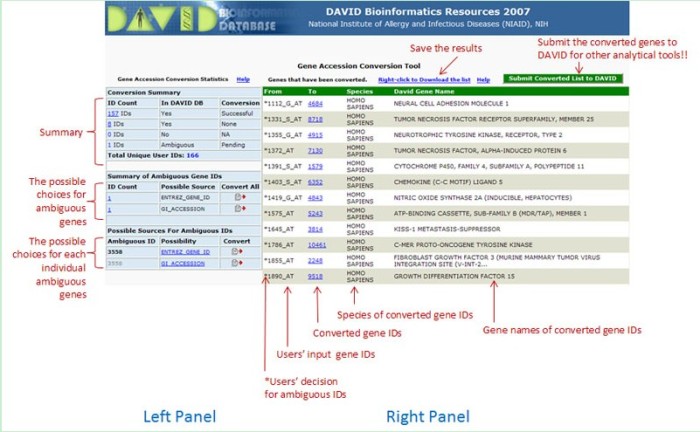
(2)选择“Gene ID Conversion Tool”这项分析工具,在弹出窗口(见图14)中,选择目的标识类型,然后点击“Submit to Conversion Tool”,弹出结果窗口,详细说明见图15。(参照网页http://david.abcc.ncifcrf.gov/helps/conversion.html#result)
图14 Gene ID Conversion Tool窗口
图15 Gene ID Conversion Tool结果说明文件
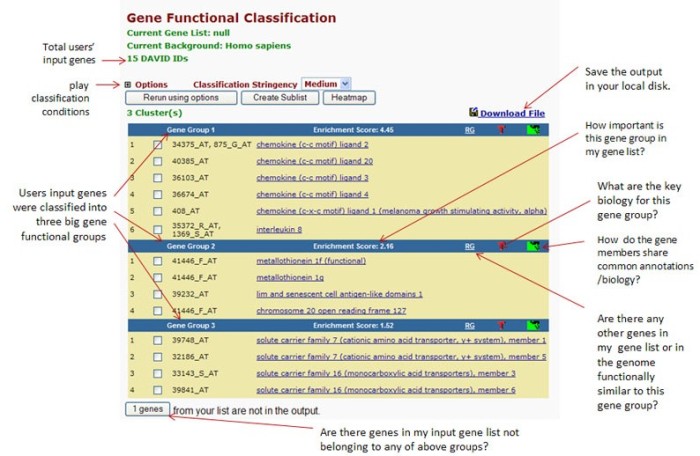
(3)选择“Gene Functional Classification”这项分析工具, 弹出的结果和具体说明见图16(具体说明参照网页http://david.abcc.ncifcrf.gov/helps/functional_classification.html#textmode)。
图16 Gene Functional Classfication
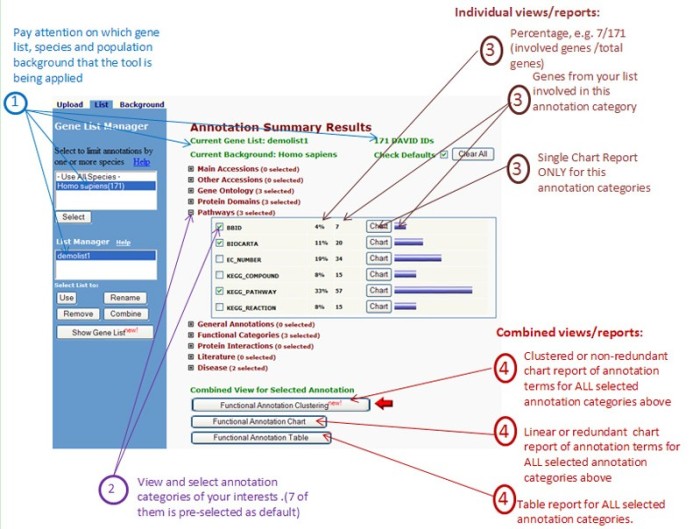
(3)选择“Functional Annotation Tool”这项分析工具,会弹出图17所示窗口(具体说明参照http://david.abcc.ncifcrf.gov/helps/functional_annotation.html#summary)。
图17 Functional Annotation Tool界面
点击页面底部的三个选项“Functional Annotation Clustering”、“Functional Annotation Chart”、“Functional Annotation Table”:
- 选择Functional Annotation Clustering这一项分析,可以对被注释上的Terms做聚类,弹出结果见图18,图中绿色的图标可以显示2-D view热图(详见之前提到)。
 图18 Functional Annotation Clustering分析结果
图18 Functional Annotation Clustering分析结果
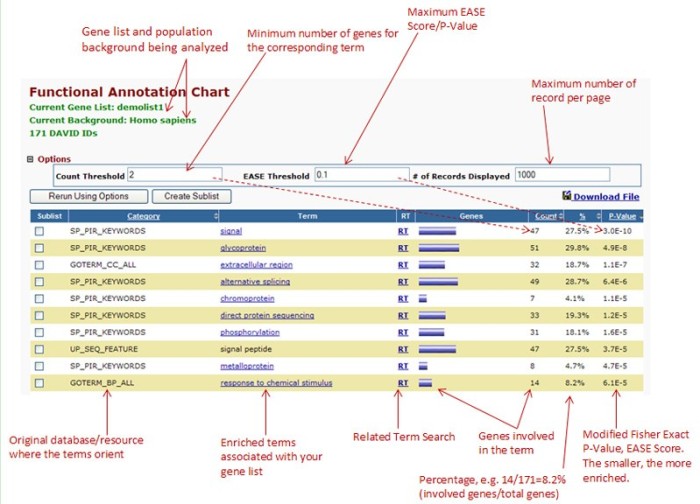
- 选择“Functional Annotation Chart“这一项分析,可以实现Terms的富集分析。结果见图19(具体说明见网页http://david.abcc.ncifcrf.gov/helps/functional_annotation.html#E3)。
图19 Functional Annotation Chart结果展示
- 选择“Functional Annotation Table “这项分析,可以实现对基因的所有选定数据库注释。具体结果见图20。
图20 Functional Annotation Table页面解说
参考文献:
Systematic and Integrative Analysis of Large Gene Lists Using DAVID Bioinformatics Resources 发表在Nature Protocols。
感谢@生物信息-小郑 在生物信息学QQ群(235461986)提供资料。