本文主要介绍 phred\phrap-基因组的序列装配软件包。Phred 是 phred\phrap 软件包 的一部分,主要是用来分析和装配基因组中大片段序列。phred\phrap 软件包由华盛顿大学 分子生物技术学院的 Phil Green 和 Brent Ewing 开发,主要用于学术科研活动。Phred 能处 理测序仪直接生成的色谱图,并且产生相关的信息。
数据来源(source data)
最 主 要 的 数 据 来 源 是 测 序 仪 生 成 的 峰 图 ( trace files ) 。 峰 图 在 计 算 机 上 可 以 用 色 谱 图(chromatograms)表示:(图 1 测序仪生成的色谱图,chromatograms) 不同的测序仪会给出不同的色谱文件,Phred 能够识别三种格式的色谱文件,SCF, ABI 和 预先处理的 ESD 格式。
(图1 测序仪生成的色谱图,chromatograms)
Phred 参数文件(Phred Parameter File)
使用 phred 首先就得配置化学物质参数文件(Phred Parameter File)。phred\phrap 软件包中 phredpar.dat 文件,就是有关测序仪每个色谱峰所代表的化学物质的参数配置文件。用户可 以直接编辑它,需要时也可以加入新的化学物质的描述信息。文件格式如下:
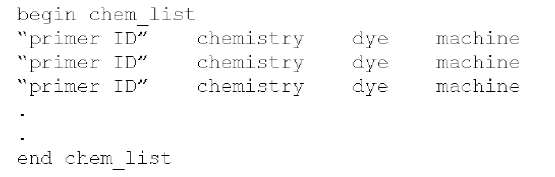
(图 2 Phred 参数文件(Phred Parameter File)的格式)
其中:
1. primer ID 代表:染色物质的 ID 号(编号),此 ID 号应该和色谱中染料的 ID 号一致(如果您不知道如何确定 Primer ID 号,不必担心,Phred 程序会给出提示的)。
2. chemistry 代表发生的化学反应,可供选择的选项有“primer,terminator,unknown”。3. dye 代 表 染 料 的 类 型 , 有 rhodamine, d-rhodamine, big-dye, energy-transfer, bodipy, unknown 等选项供选择。
4. machine 代 表 测 序 仪 型 号 。 phred ( 版 本 0.020425.c ) 现 在 可 识 别 的 仪 器 有 :ABI_373_377, ABI_3100, ABI_3700, Beckman_CEQ_2000, LI-COR_4000, andMolDyn_MegaBACE。
phredpar.dat 文 件 必 须 放 在 所 有 用 户 均 可 访 问 的 目 录 , 并 且 可 以 通 过PHRED_PARAMETER_FILE 环境变量来自定义其存放位置。 例如,在 Unix 系统中:export PHRED_PARAMETER_FILE=\usr\local\etc\phredpar.dat在 Windows 系统中:set PHRED_PARAMETER_FILE=\usr\local\etc\phredpar.dat
Phred 输入参数(Phred input parameters)
输入参数表示色谱图文件在计算机里面的路径,有以下两种设置方式:
-id <directory>
## -id 选项表示所有色谱文件的所在目录。
-if <text file>
## -if 选项表示每个色谱文件的绝对路径。
Phred 输出参数(Phred output parameters)
输出参数用来设置输出文件的格式。输出参数分为 base calling, quality, SCF, PHD 和 poly 六 大类(每类对应一个选项族),其不同组合能够产生几种完全不同的输出格式。
Base calling 选项族主要是关于输出的 DNA 序列,可供选择的选项有如下几种:
-st <fasta/xbap >
## -st 设定输出文件格式,能被识别的格式有 fasta 和 xbap,默认设置为 fasta 格式。
-s
## -s 在当前目录下创建序列文件,并且沿用色谱图(chromatogram)文件名,贯于.sep 的后缀。
-sd <directory>
## -sd 在指定目录下创建序列文件,并且沿用色谱图(chromatogram)文件名,贯于.seq 的后缀。
-sa <file>
## -sa 创建单个序列文件,包括所有处理过的色谱图(chromatograms)的结果。
Quality 选项族主要是 DNA 序列的测序质量信息输出的相关选项,有如下几种:
-qt <fasta/xbap/mix>
## -qt 各 种 输 出 文 件 格 式 的 测 序 质 量 ,fasta 选 项 对 应 修 整 过 后 的 FASTA(trimmed FASTA,在“phred processing options”中设定)格式,xbap 选项对应 XBAP 格式,mix 选 项对应未修整过后的 FASTA(untrimmed FASTA,这也是默认的 FASTA 格式);在设定选 项时请参照“phred processing options”中的对应选项。
-q
## -q 在当前目录下创建测序质量文件,并且沿用色谱图(chromatogram)文件名,附加“ .qual ”后缀。
-qd <directory>
## -qd 在指定目录下创建测序质量文件,并且沿用色谱图(chromatogram)文件名,附加“. qual”后缀。
-qa <file>
## -qa 创建单个测序质量文件,包括所有的处理过的色谱图(chromatogram)的结 果。
-qr <file>
## -qr 创 建 一 个 柱 状 图 文 件 , 统 计 每 一 个 色 谱 图 ( chromatogram )中高质量碱基(high quality bases)的数目。
SCF 选项族设置 Phred 产生基于色谱图(chromatogram)SCF 格式的文件,这些文件可以 用于那些不能识别 ABI 和 ESD 格式的程序。有如下几种:
-c
## -c 在当前目录下创建 SCF 文件,并且沿用色谱图(chromatogram)文件名。
-cd <directory>
## -cd 在指定目录下创建 SCF 文件,并且沿用色谱图(chromatogram)文件名。
-cp <1/2>
## -cp 编码色谱图(chromatogram)中每个色谱峰值的比特数
-cv <1/2/3>
## -cv 用 SCF1,SCF2 或者 SCF3 格式输出 SCF 文件。
-cs
## -cs 确保色谱图(chromatogram)中最大的峰度值代表 SCF 文件中最高值。
PHD 选项族设置 Phred 程序基于色谱图(chromatogram)产生 PHD 格式的文件,这些文件可供人直接阅读,并且含有 base calling 和 quality(测序质量)的信息。有如下几种:
-p
## -p 在当前目录下创建 PHD 文件,并且沿用色谱图(chromatogram)文件名,附加.phd..l 的后缀。
-pd <directory>
## -pd 在指定目录下创建 PHD 文件,并且沿用色谱图(chromatogram)文件名,附 加 .phd..l 的后缀。
poly 选 项 族 设 置 Phred 程 序 产 生 poly 文 件 ( poly files ) 。 这 些 文 件 包 含 了 色 谱 图(chromatogram)中的每个峰值,可用于检测多态性碱基(polymorphic bases)。有如下几 种:
-d
## -d 在当前目录下创建测序质量文件,并且沿用色谱图(chromatogram)文件名, 附加 .poly 的后缀。
-dd <directory>
## -dd 在指定目录下创建测序质量文件,并且沿用色谱图(chromatogram)文件名, 附加 . poly 的后缀。
其他不能归为上述几类但与输出相关的选项有:
-raw <name>
## -raw 当处理单个文件时,以 name 为标题。
-log
## -log 在当前目录下生成“phred.log”日志文件。
-v <n>
## -v 设置增加输出的冗余(increase verbosity of output by n)。
-tag
## -tag 做一些公共的标记(tag)以便于程序解析文档。
-h
## -h 显示一个简要的帮助。
-doc
## -doc 显示 phred 的全部文档(documentation)。
-V
## -V 显示 phred 的版本信息。
Phred 处理选项(Phred processing options)
Phred 处理选项是为经验丰富的用户提供的,可以改变 phred 的处理流程。可供配置的选项 如下:
-nocall
## -nocall 不用 base calling 算法对色谱图(chromatogram)的峰值进行检测,输出的 序列是由色谱图(chromatogram)中的峰值直接转换而来,这会影响到一些碱基修整和输出选项(This affects the base trimming and output options)。
-nonorm
## -nonorm 设 置 phred 不 对 色 谱 图 ( chromatogram ) 中 的 峰 值 进 行 标 准 化 处 理(normalization)。如果进行标准化处理,就会用每个核苷酸信号的中值(medium value) 来代替峰值(peak)。这个选项不推荐使用,除非 base calling 算法由于有很多的峰值噪声 而失效。
-nosplit
## -nosplit 设置 phred 不对色谱图(chromatogram)中已压缩的峰值进行分割处理。 默认情况下,phred 对相连的 G , C 峰(merged CC and GG peaks)进行识别并分割开来。
-nocmpqv
## -nocmpqv 设置 phred 不对色谱图(chromatogram)中的峰值进行压缩处理。默认 情况下,phred 会降低相连的 G , C 峰(merged CC and GG peaks)的测序质量分值,因此 如果这个打开该选项会影响输出文件中的测序质量。
-ceilqv <value>
## -ceilqv 为每个碱基设定最高的测序质量值,当碱基的测序质量值超过该值时用该 值替代。
-beg_pred <position>
## -beg_pred 设 定 开 始 进 行 峰 值 预 测 的 位 置 。 该 位 置 应 该 落 在 一 个 非 常 好 的 区 域(region)中,在此区域(region)中每个峰值间的间隔都很均一(even)。
-exit_nomatch
## 如果在 Phred 参数文件(Phred Parameter File)中没发现相对应的 primer ID 则停 止执行。
-process _nomatch
## 如果在 Phred 参数文件(Phred Parameter File)中没发现相对应的 primer ID 则搜 索 Phred 参数文件(Phred Parameter File)中的“_no_matching_string_”条目并用该条目来 识别色谱图中的峰,如果还是没有定义则停止执行。
以下的选项是针 对 phred 修整( trimming)碱基的设定。 这些选项在定位高测序 质量区(high quality regions)非常有用,并且能裁减掉一些低质量的区域。可供选择的选项如 下:
-trim <enzyme sequence>
## -trim 查 找 并 定 位 色 谱 图 ( chromatogram ) 中 的 高 测 序 质 量 区 ( high quality regions)。如果提供了限制性酶的序列,phred 会从该酶切位点的开始位置开始进行修整, 推荐将酶切序列置为空(enzyme sequence 即用空的双引号"")。
-trim_alt <enzyme sequence>
## -trim_alt 同 -trim 一 样 定 位 高 测 序 质 量 区 ( high quality regions) , 不 过 使 用 的 是“最大分值区域”(“Maximum Score Subsequence”)的算法。推荐使用。“Maximum Score Subsequence”大体思路是将每个碱基的错误概率(由机器提供)减去一个 cutoff(默认 的为 0.05),所得的结果再相加直到分值最大为止,可见不一定序列越长就分值越高,关 键是看每个碱基的测序质量。
-trim_cutoff <value>
## 在用最大分值区域”(“Maximum Score Subsequence”)的算法时设定一个错误阈值。默认的为 0.05。
-trim_fasta
## 修整后的序列和质量分值写入 FASTA 格式的文件中。
-trim_scf
## 修整后的序列和质量分值写入 SCF 格式的文件中。
-trim_phd
## 修整后的序列和质量分值写入 PHD 格式的文件中。
-trim_out
## 修整后的序列和质量分值写入 FASTA SCF PHD 三种格式的文件中。
phred 的质量分值(Phred quality determination)
为了确定最后的质量分值,phred 分析四种碱基的在色谱图(chromatogram)中的峰
轨迹(trace),利用各种识别方法尽量识别每个峰轨迹(trace),同时兼顾全局的峰轨迹
(trace)的识别;言下之意就是说可能某个峰轨迹(trace)用某种方法可以很好的识别但 如果这造成全局其他的峰轨迹(trace)不能得到很好的识别,这样的方法不可取。选定了 某种方法后,这样在保证全局的识别情况下必然会有单个的峰轨迹(trace)被认为是错误 的需要校正,这样就可以统计出碱基测序错误的频度,比如说每 100 个碱基就有一个错 误。然后就可以用这个错误频度来度量测序的质量了。公式如下:
Q = -10 log10 (P)
公式中的 Q 代表了碱基的测序质量值,P代表每个碱基测序出错的概率。例如,如果每100 个碱基就有一个错误,那么 P=0.01,这样 Q 就为 20;如果 P=0.001, Q 为 30。注意 当 P 为错误阈值(cutoff,默认为 0.05)时,Q 近似为 13,所以 13 就可用作背景来估计总 体的质量值。



