目的:
- 鉴定该物种中某个基因家族的所有成员;
- 每个基因的功能鉴定;
什么是HMM:
HMM(Hidden Markov Model,隐马尔科夫模型)是一种用参数表示的用于描述随机过程统计特性的概率模型,是一个双重随机过程, 由两个部分组成:马尔可夫链和一般随机过程。 其中马尔可夫链用来描述状态的转移,用转移概率描述。一般随机过程用来描述状态与观察序列间的关系,用观察值概率描述。对于HMM模型,其的状态转换过程是不可观察的,因而称之为“隐”马尔可夫模型。
为什么要用HMM:
HMM是一种序列特征谱(Profile),其搜索是基于蛋白质序列多重比对结果中的保守序列区域进行搜索,由于考虑了不同保守度的氨基酸在相应位置的权重,可以更为敏感的检测到进化距离较远的蛋白质相关性,得到比序列模式方法更为灵敏的结果。
需要做什么,如何做
鉴定该物种中某个基因家族的所有成员:
- 得到已知的基因家族多重比对序列
- 从多重比对中构建HMM模型,也可以直接从pfam下载某个基因家族的HMM模型
- 用模型搜寻目标基因组的蛋白质库
每个基因的功能鉴定:
- 下载构建HMM数据库
- 使用基因的蛋白质序列搜索HMM数据库
局限性
受限于可靠的序列特征谱数目,因此该方法在进行新基因功能预测时受到了较大的障碍。
基于HMM的数据库与工具
- 数据库PFAM
- 程序hmmer
- 程序HHsearch
- 基于hmmer的实现
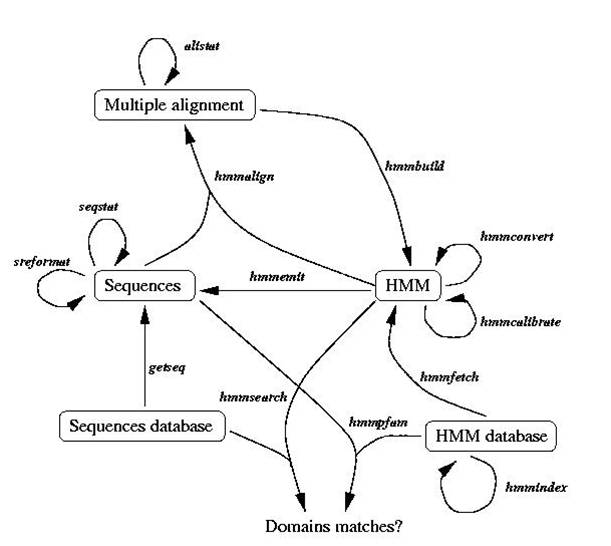
hmmer是应用广泛的,功能全面的HMM应用的实现,目前最新版本3.0(28 March 2010发布)。由一组程序组成,包括多重比对方面的、模型构建方面的、蛋白质序列方面的、序列数据库方面、HMM数据库方面的,其关系参看下图:
官方介绍:
HMMER is used for searching sequence databases for homologs of protein sequences, and for making protein sequence alignments. It implements methods using probabilistic models called profile hidden Markov models (profile HMMs).Compared to BLAST, FASTA, and other sequence alignment and database search tools based on older scoring methodology, HMMER aims to be significantly more accurate and more able to detect remote homologs because of the strength of its underlying mathematical models. In the past, this strength came at significant computational expense, but in the new HMMER3 project, HMMER is now essentially as fast as BLAST.
主要应用的程序:
- hmmbuild 由多重比对序列构建hmm模型
- hmmsearch 通过hmm模型搜索蛋白质序列数据库
- hmmpfam 基因的蛋白质序列搜索HMM数据库,鉴定结构域
参考:
- HMM的描述: http://zh.wikipedia.org/wiki/%E9%A9%AC%E5%B0%94%E5%8F%AF%E5%A4%AB%E8%BF%87%E7%A8%8B
- hmmer主页:http://hmmer.janelia.org/
- 统计学方面的博客: http://www.52nlp.cn/category/hidden-markov-model